-検索条件
-検索結果
検索 (著者・登録者: huang & pc)の結果53件中、1から50件目までを表示しています

EMDB-15595: 
cryo-EM structure of carboxysomal mini-shell: oblate structure from C1 construct (T=7 Q=6)

EMDB-15611: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from C2 construct (T=4)

EMDB-15719: 
cryo-EM structure of carboxysome mini-shell: icosahedral structure from C1 construct (T=4)

EMDB-15720: 
cryo-EM structure of carboxysome mini-shell: icosahedral structure from C1 construct (T=7)

EMDB-15722: 
cryo-EM structure of carboxysome mini-shell: icosahedral structure from C1 construct (T=9)

EMDB-15723: 
cryo-EM structure of carboxysomal mini-shell: prolate structure from C1 construct (T=4 Q=6)

EMDB-15724: 
cryo-EM structure of carboxysomal mini-shell: prolate structure from C1 construct (T=4 Q=6) form 2

EMDB-15758: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from C3 construct (T=4)

EMDB-15759: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from C3 construct (T=4-P)

EMDB-15760: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from C3 construct (T=3)

EMDB-15761: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from C3 construct (T=3-P)

EMDB-15762: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from C1 ITG mutant construct (T=4)

EMDB-15792: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from CsoS4A-1A (T=4)

EMDB-15798: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from CsoS4A/1A co-expression (T = 3)

EMDB-15799: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from CsoS4A/1A and CsoS2 co-expression (T = 4)

EMDB-15801: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from CsoS4A/1A and CsoS2 co-expression (T = 9)

EMDB-15834: 
cryo-EM structure of carboxysomal mini-shell: icosahedral assembly from C1 ITG mutant construct (T=3)

EMDB-17756: 
Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein

EMDB-27112: 
S728-1157 IgG in complex with SARS-CoV-2-6P-Mut7 Spike protein (global refinement)

EMDB-27113: 
S728-1157 IgG in complex with SARS-CoV-2-6P-Mut7 Spike protein (focused refinement)

EMDB-32352: 
Structural of the filamentous Escherichia coli glutamine synthetase

EMDB-25634: 
Negative stain map of monoclonal Fab 047-09 4F04 binding the anchor epitope of H1 HA

EMDB-25635: 
Negative stain map of monoclonal Fab 241 IgA 2F04 binding the anchor epitope of H1 HA

EMDB-25636: 
Negative stain map of polyclonal Fab 236.7 binding the anchor and esterase epitopes of H1 HA

EMDB-25637: 
Negative stain map of polyclonal Fab 236.7 binding the RBS epitope of H1 HA

EMDB-25638: 
Negative stain map of polyclonal Fab 236.14 binding an epitope on the top of the head of H1 HA
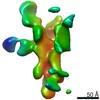
EMDB-25639: 
Negative stain map of polyclonal Fab 236.14 binding the esterase epitope of H1 HA

EMDB-25640: 
Negative stain map of polycolonal Fab 236.14 binding the RBS epitope of H1 HA

EMDB-25641: 
Negative stain map of polyclonal Fab 236.14 binding the anchor epitope of H1 HA
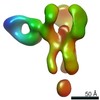
EMDB-25642: 
Negative stain map of polyclonal Fab 241.7 binding the esterase epitope of H1 HA
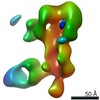
EMDB-25643: 
Negative stain map of polyclonal Fab 241.14 binding the anchor epitope of H1 HA
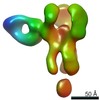
EMDB-25644: 
Negative stain map of polyclonal Fab 241.14 binding the esterase epitope of H1 HA

EMDB-25645: 
Negative stain map of polyclonal Fab 241.14 binding an epitope on the top of the head of H1 HA

EMDB-25646: 
Negative stain map of polyclonal Fab 241.14 binding the RBS epitope of H1 HA

EMDB-25655: 
CryoEM map of anchor 222-1C06 Fab and lateral patch 2B05 Fab binding H1 HA

EMDB-23792: 
CryoEM structure of monoclonal Fab 045-09 2B05 binding the lateral patch of influenza virus H1 HA
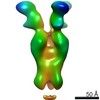
EMDB-23793: 
Negative stain map of monoclonal Fab SFV009 2G01 binding the RBS of H1 HA

EMDB-23794: 
Negative stain map of monoclonal Fab 045-09 2B05 binding the lateral patch of H1 HA

EMDB-23795: 
Negative stain map of monoclonal Fab SFV019 2A06 binding the lateral patch of H1 HA
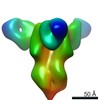
EMDB-23796: 
Negative stain map of monoclonal Fab SFV015 2F02 binding the lateral patch of H1 HA

EMDB-23797: 
Negative stain map of monoclonal Fab 047-09 4G02 binding the lateral patch of H1 HA
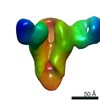
EMDB-23798: 
Negative stain map of monoclonal Fab 047-09 4B06 binding the lateral patch of H1 HA

EMDB-23977: 
Rhodopsin kinase (GRK1)-S5E/S488E/T489E in complex with rhodopsin

EMDB-23978: 
Rhodopsin kinase (GRK1) in complex with rhodopsin

EMDB-23979: 
Rhodopsin kinase (GRK1)-S5E/S488E/T489E in complex with rhodopsin and Fab1

EMDB-23980: 
Rhodopsin kinase (GRK1)-S5E/S488E/T489E in complex with rhodopsin and Fab6
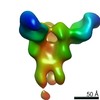
EMDB-23313: 
Negative stain map of monoclonal Fab 23 binding the lateral patch of H1 HA

EMDB-23314: 
Negative stain map of monoclonal Fab 45 binding the esterase domain of H1 HA
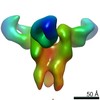
EMDB-23315: 
Negative stain map of monoclonal Fab 50 binding the lateral patch of H1 HA
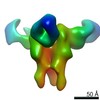
EMDB-23316: 
Negative stain map of monoclonal Fab 56 binding the lateral patch of H1 HA
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
