-検索条件
-検索結果
検索 (著者・登録者: henderson & n)の結果303件中、1から50件目までを表示しています

EMDB-40853: 
CH505 Disulfide Stapled SOSIP Bound to b12 Fab

EMDB-40854: 
CH505 Disulfide Stapled SOSIP Bound to CH235.12 Fab

PDB-8sxi: 
CH505 Disulfide Stapled SOSIP Bound to b12 Fab

PDB-8sxj: 
CH505 Disulfide Stapled SOSIP Bound to CH235.12 Fab

EMDB-41810: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env Local Refinement

EMDB-41820: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env

EMDB-41823: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env

EMDB-41838: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to partially open HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env

PDB-8u1d: 
Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env Local Refinement

EMDB-17958: 
Structure of DPS determined by cryoEM at 100 keV

EMDB-17959: 
Structure of bacterial ribosome determined by cryoEM at 100 keV

EMDB-17960: 
Structure of GABAAR determined by cryoEM at 100 keV

EMDB-17961: 
Structure of mouse heavy-chain apoferritin determined by cryoEM at 100 keV

EMDB-17962: 
Structure of catalase determined by cryoEM at 100 keV

EMDB-17963: 
Structure of AHIR determined by cryoEM at 100 keV

EMDB-17964: 
Structure of GAPDH determined by cryoEM at 100 keV

EMDB-17965: 
Structure of E. coli glutamine synthetase determined by cryoEM at 100 keV

EMDB-17966: 
Structure of human apo ALDH1A1 determined by cryoEM at 100 keV

EMDB-17967: 
Structure of PaaZ determined by cryoEM at 100 keV

EMDB-17968: 
Structure of lumazine synthase determined by cryoEM at 100 keV

PDB-8pv9: 
Structure of DPS determined by cryoEM at 100 keV

PDB-8pva: 
Structure of bacterial ribosome determined by cryoEM at 100 keV

PDB-8pvb: 
Structure of GABAAR determined by cryoEM at 100 keV

PDB-8pvc: 
Structure of mouse heavy-chain apoferritin determined by cryoEM at 100 keV

PDB-8pvd: 
Structure of catalase determined by cryoEM at 100 keV

PDB-8pve: 
Structure of AHIR determined by cryoEM at 100 keV

PDB-8pvf: 
Structure of GAPDH determined by cryoEM at 100 keV

PDB-8pvg: 
Structure of E. coli glutamine synthetase determined by cryoEM at 100 keV

PDB-8pvh: 
Structure of human apo ALDH1A1 determined by cryoEM at 100 keV

PDB-8pvi: 
Structure of PaaZ determined by cryoEM at 100 keV

PDB-8pvj: 
Structure of lumazine synthase determined by cryoEM at 100 keV

EMDB-27703: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace

PDB-8dtk: 
Structure of RBD directed antibody DH1047 in complex with SARS-CoV-2 spike: Local refinement of RBD-Fab interace

PDB-7tcn: 
Cryo-EM structure of CH235.12 in complex with HIV-1 Env trimer CH505TF.N279K.SOSIP.664 with high-mannose glycans

PDB-7tco: 
Cryo-EM structure of CH235.12 in complex with HIV-1 Env trimer CH505TF.N279K.G458Y.SOSIP.664 with high-mannose glycans

EMDB-27624: 
Cryo-EM structure of HIV-1 Env(CH848 10.17 DS.SOSIP_DT) in complex with DH1030.1 Fab

EMDB-27628: 
Cryo-EM structure of HIV-1 Env(BG505.T332N SOSIP) in complex with DH1030.1 Fab
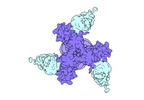
EMDB-27706: 
Vaccine elicited Antibody MU89 bound to CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 Env trimer
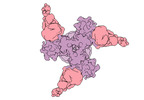
EMDB-27776: 
Vaccine elicited Antibody MU89+S27Y bound to CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 Env trimer

PDB-8dto: 
Vaccine elicited Antibody MU89 bound to CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 Env trimer

PDB-8dy6: 
Vaccine elicited Antibody MU89+S27Y bound to CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 Env trimer

EMDB-40273: 
CryoEM structure of VRC01-CH848.0358.80

EMDB-40274: 
CryoEM structure of VRC01-CH848.0836.10

EMDB-40275: 
CryoEM structure of DH270.6-CH848.0526.25

EMDB-40277: 
CryoEM structure of DH270.6-CH848.10.17

EMDB-40278: 
CryoEM structure of DH270.5-CH848.10.17

EMDB-40279: 
CryoEM structure of VRC01-CH848.10.17

EMDB-40280: 
CryoEM structure of DH270.4-CH848.10.17

EMDB-40281: 
CryoEM structure of VRC01-CH848.0526.25

EMDB-40282: 
CryoEM structure of DH270.UCA.G57R-CH848.10.17DT
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
