+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of 5-subunit Smc5/6 arm region | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CELL CYCLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Smc5-Smc6 complex / resolution of DNA recombination intermediates / DNA double-strand break attachment to nuclear envelope / chromosome separation / SUMO ligase activity / SUMOylation of DNA damage response and repair proteins / chromatin looping / protein serine/threonine kinase inhibitor activity / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / SUMO transferase activity ...Smc5-Smc6 complex / resolution of DNA recombination intermediates / DNA double-strand break attachment to nuclear envelope / chromosome separation / SUMO ligase activity / SUMOylation of DNA damage response and repair proteins / chromatin looping / protein serine/threonine kinase inhibitor activity / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / SUMO transferase activity / recombinational repair / regulation of telomere maintenance / protein sumoylation / double-strand break repair via homologous recombination / nuclear envelope / site of double-strand break / single-stranded DNA binding / chromosome, telomeric region / damaged DNA binding / DNA repair / ATP hydrolysis activity / mitochondrion / zinc ion binding / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.97 Å | |||||||||
 データ登録者 データ登録者 | Qian L / Jun Z / Xiang Z / Cheng T / Zhaoning W / Zhenguo C / Wang L | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Cryo-EM structures of Smc5/6 in multiple states reveal its assembly and functional mechanisms. 著者: Qian Li / Jun Zhang / Cory Haluska / Xiang Zhang / Lei Wang / Guangfeng Liu / Zhaoning Wang / Duo Jin / Tong Cheng / Hongxia Wang / Yuan Tian / Xiangxi Wang / Lei Sun / Xiaolan Zhao / Zhenguo ...著者: Qian Li / Jun Zhang / Cory Haluska / Xiang Zhang / Lei Wang / Guangfeng Liu / Zhaoning Wang / Duo Jin / Tong Cheng / Hongxia Wang / Yuan Tian / Xiangxi Wang / Lei Sun / Xiaolan Zhao / Zhenguo Chen / Lanfeng Wang /   要旨: Smc5/6 is a member of the eukaryotic structural maintenance of chromosomes (SMC) family of complexes with important roles in genome maintenance and viral restriction. However, limited structural ...Smc5/6 is a member of the eukaryotic structural maintenance of chromosomes (SMC) family of complexes with important roles in genome maintenance and viral restriction. However, limited structural understanding of Smc5/6 hinders the elucidation of its diverse functions. Here, we report cryo-EM structures of the budding yeast Smc5/6 complex in eight-subunit, six-subunit and five-subunit states. Structural maps throughout the entire length of these complexes reveal modularity and key elements in complex assembly. We show that the non-SMC element (Nse)2 subunit supports the overall shape of the complex and uses a wedge motif to aid the stability and function of the complex. The Nse6 subunit features a flexible hook region for attachment to the Smc5 and Smc6 arm regions, contributing to the DNA repair roles of the complex. Our results also suggest a structural basis for the opposite effects of the Nse1-3-4 and Nse5-6 subcomplexes in regulating Smc5/6 ATPase activity. Collectively, our integrated structural and functional data provide a framework for understanding Smc5/6 assembly and function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35185.map.gz emd_35185.map.gz | 117.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35185-v30.xml emd-35185-v30.xml emd-35185.xml emd-35185.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35185.png emd_35185.png | 40.2 KB | ||
| Filedesc metadata |  emd-35185.cif.gz emd-35185.cif.gz | 7 KB | ||
| その他 |  emd_35185_half_map_1.map.gz emd_35185_half_map_1.map.gz emd_35185_half_map_2.map.gz emd_35185_half_map_2.map.gz | 115.9 MB 115.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35185 http://ftp.pdbj.org/pub/emdb/structures/EMD-35185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35185 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35185_validation.pdf.gz emd_35185_validation.pdf.gz | 706.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35185_full_validation.pdf.gz emd_35185_full_validation.pdf.gz | 705.7 KB | 表示 | |
| XML形式データ |  emd_35185_validation.xml.gz emd_35185_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_35185_validation.cif.gz emd_35185_validation.cif.gz | 16 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35185 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35185 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8i4vMC  7ylmC  7ymdC  7yqhC  8hqsC  8i13C  8i21C  8i4uC  8i4wC  8i4xC  8wjlC  8wjnC  8wjoC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35185.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35185.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 2.128 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_35185_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
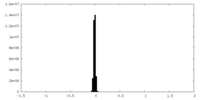
| 投影像・断面図 |
| ||||||||||||
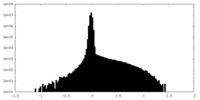
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35185_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
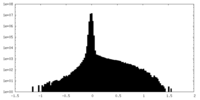
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of 5-subunit Smc5/6 arm region
| 全体 | 名称: Cryo-EM structure of 5-subunit Smc5/6 arm region |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of 5-subunit Smc5/6 arm region
| 超分子 | 名称: Cryo-EM structure of 5-subunit Smc5/6 arm region / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Structural maintenance of chromosomes protein 5
| 分子 | 名称: Structural maintenance of chromosomes protein 5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 123.390031 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KRVKIAKPDL SSFQPGSIIK IRLQDFVTYT LTEFNLSPSL NMIIGPNGSG KSTFVCAVCL GLAGKPEYIG RSKKVEDFIK NGQDVSKIE ITLKNSPNVT DIEYIDARDE TIKITRIITR SKRRSDYLIN DYQVSESVVK TLVAQLNIQL DNLCQFLSQE R VEEFARLK ...文字列: KRVKIAKPDL SSFQPGSIIK IRLQDFVTYT LTEFNLSPSL NMIIGPNGSG KSTFVCAVCL GLAGKPEYIG RSKKVEDFIK NGQDVSKIE ITLKNSPNVT DIEYIDARDE TIKITRIITR SKRRSDYLIN DYQVSESVVK TLVAQLNIQL DNLCQFLSQE R VEEFARLK SVKLLVETIR SIDASLLDVL DELRELQGNE QSLQKDLDFK KAKIVHLRQE SDKLRKSVES LRDFQNKKGE IE LHSQLLP YVKVKDHKEK LNIYKEEYER AKANLRAILK DKKPFANTKK TLENQVEELT EKCSLKTDEF LKAKEKINEI FEK LNTIRD EVIKKKNQNE YYRGRTKKLQ ATIISTKEDF LRSQEILAQT HLPEKSVFED IDIKRKEIIN KEGEIRDLIS EIDA KANAI NHEMRSIQRQ AESKTKSLTT TDKIGILNQD QDLKEVRDAV LMVREHPEMK DKILEPPIMT VSAINAQFAA YLAQC VDYN TSKALTVVDS DSYKLFANPI LDKFKVNLRE LSSADTTPPV PAETVRDLGF EGYLSDFITG DKRVMKMLCQ TSKIHT IPV SRRELTPAQI KKLITPRPNG KILFKRIIHG NRLVDIKQSA YGSKQVFPTD VSIKQTNFYQ GSIMSNEQKI RIENEII NL KNEYNDRKST LDALSNQKSG YRHELSELAS KNDDINREAH QLNEIRKKYT MRKSTIETLR EKLDQLKREA RKDVSQKI K DIDDQIQQLL LKQRHLLSKM ASSMKSLKNC QKELISTQIL QFEAQNMDVS MNDVIGFFNE READLKSQYE DKKKFVKEA RDTPEFQSWM REIRSYDQDT KEKLNKVAEK YEEEGNFNLS FVQDVLDKLE SEIAMVNHDE SAVTILDQVT AELRELEHTV PQQSKDLET IKAKLKEDHA VLEPKLDDIV SKISARFARL FNNVGSAGAV RLEKPKDYAE WKIEIMVKFR DNAPLKKLDS H TQSGGERA VSTVLYMIAL QEFTSAPFRV VDEINQGMDS RNERIVHKAM VENACAENTS QYFLITPKLL TGLHYHEKMR IH CVMAGSW IPNPSEDPKM IHFGETSNYS FD UniProtKB: Structural maintenance of chromosomes protein 5 |
-分子 #2: Structural maintenance of chromosomes protein 6
| 分子 | 名称: Structural maintenance of chromosomes protein 6 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 128.199727 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MISTTISGKR PIEQVDDELL SLTAQQENEE QQQQRKRRRH QFAPMTQFNS NTLDEDSGFR SSSDVATADQ DNFLEESPSG YIKKVILRN FMCHEHFELE LGSRLNFIVG NNGSGKSAIL TAITIGLGAK ASETNRGSSL KDLIREGCYS AKIILHLDNS K YGAYQQGI ...文字列: MISTTISGKR PIEQVDDELL SLTAQQENEE QQQQRKRRRH QFAPMTQFNS NTLDEDSGFR SSSDVATADQ DNFLEESPSG YIKKVILRN FMCHEHFELE LGSRLNFIVG NNGSGKSAIL TAITIGLGAK ASETNRGSSL KDLIREGCYS AKIILHLDNS K YGAYQQGI FGNEIIVERI IKRDGPASFS LRSENGKEIS NKKKDIQTVV DYFSVPVSNP MCFLSQDAAR SFLTASTSQD KY SHFMKGT LLQEITENLL YASAIHDSAQ ENMALHLENL KSLKAEYEDA KKLLRELNQT SDLNERKMLL QAKSLWIDVA HNT DACKNL ENEISGIQQK VDEVTEKIRN RQEKIERYTS DGTTIEAQID AKVIYVNEKD SEHQNARELL RDVKSRFEKE KSNQ AEAQS NIDQGRKKVD ALNKTIAHLE EELTKEMGGD KDQMRQELEQ LEKANEKLRE VNNSLVVSLQ DVKNEERDIQ HERES ELRT ISRSIQNKKV ELQNIAKGND TFLMNFDRNM DRLLRTIEQR KNEFETPAIG PLGSLVTIRK GFEKWTRSIQ RAISSS LNA FVVSNPKDNR LFRDIMRSCG IRSNIPIVTY CLSQFDYSKG RAHGNYPTIV DALEFSKPEI ECLFVDLSRI ERIVLIE DK NEARNFLQRN PVNVNMALSL RDRRSGFQLS GGYRLDTVTY QDKIRLKVNS SSDNGTQYLK DLIEQETKEL QNIRDRYE E KLSEVRSRLK EIDGRLKSTK NEMRKTNFRM TELKMNVGKV VDTGILNSKI NERKNQEQAI ASYEAAKEEL GLKIEQIAQ EAQPIKEQYD STKLALVEAQ DELQQLKEDI NSRQSKIQKY KDDTIYYEDK KKVYLENIKK IEVNVAALKE GIQRQIQNAC AFCSKERIE NVDLPDTQEE IKRELDKVSR MIQKAEKSLG LSQEEVIALF EKCRNKYKEG QKKYMEIDEA LNRLHNSLKA R DQNYKNAE KGTCFDADMD FRASLKVRKF SGNLSFIKDT KSLEIYILTT NDEKARNVDT LSGGEKSFSQ MALLLATWKP MR SRIIALD EFDVFMDQVN RKIGTTLIVK KLKDIARTQT IIITPQDIGK IADIDSSGVS IHRMRDPERQ NNSNFYN UniProtKB: Structural maintenance of chromosomes protein 6 |
-分子 #3: E3 SUMO-protein ligase MMS21
| 分子 | 名称: E3 SUMO-protein ligase MMS21 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: 転移酵素; アシル基を移すもの; アミノアシル基を移すもの |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 28.600305 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NDNPIPKSVP LHPKSGKYFH NLHARDLSNI YQQCYKQIDE TINQLVDSTS PSTIGIEEQV ADITSTYKLL STYESESNSF DEHIKDLKK NFKQSSDACP QIDLSTWDKY RTGELTAPKL SELYLNMPTP EPATMVNNTD TLKILKVLPY IWNDPTCVIP D LQNPADED ...文字列: NDNPIPKSVP LHPKSGKYFH NLHARDLSNI YQQCYKQIDE TINQLVDSTS PSTIGIEEQV ADITSTYKLL STYESESNSF DEHIKDLKK NFKQSSDACP QIDLSTWDKY RTGELTAPKL SELYLNMPTP EPATMVNNTD TLKILKVLPY IWNDPTCVIP D LQNPADED DLQIEGGKIE LTCPITCKPY EAPLISRKCN HVFDRDGIQN YLQGYTTRDC PQAACSQVVS MRDFVRDPIM EL RCKIAKM KESQ UniProtKB: E3 SUMO-protein ligase MMS21 |
-分子 #4: DNA repair protein KRE29
| 分子 | 名称: DNA repair protein KRE29 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.337511 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PILKRTIISK RKAPSNNEDE EIVKTPRKLV NYVPLKIFNL GDSFDDTITT TVAKLQDLKK EILD UniProtKB: DNA repair protein KRE29 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: DARK FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 5.97 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 227122 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X