+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of Nse1/3/4 | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | CELL CYCLE | |||||||||
| Function / homology |  Function and homology information Function and homology informationSmc5-Smc6 complex / Platelet degranulation / SUMOylation of DNA damage response and repair proteins / chromatin looping / DNA damage tolerance / regulation of telomere maintenance / double-strand break repair via homologous recombination / RING-type E3 ubiquitin transferase / ubiquitin-protein transferase activity / chromosome, telomeric region ...Smc5-Smc6 complex / Platelet degranulation / SUMOylation of DNA damage response and repair proteins / chromatin looping / DNA damage tolerance / regulation of telomere maintenance / double-strand break repair via homologous recombination / RING-type E3 ubiquitin transferase / ubiquitin-protein transferase activity / chromosome, telomeric region / DNA repair / zinc ion binding / nucleus / cytoplasm Similarity search - Function | |||||||||
| Biological species |   | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 4.176 Å | |||||||||
 Authors Authors | Qian L / Jun Z / Zhenguo C / Lanfeng W | |||||||||
| Funding support | 1 items
| |||||||||
 Citation Citation |  Journal: Nat Struct Mol Biol / Year: 2024 Journal: Nat Struct Mol Biol / Year: 2024Title: Cryo-EM structures of Smc5/6 in multiple states reveal its assembly and functional mechanisms. Authors: Qian Li / Jun Zhang / Cory Haluska / Xiang Zhang / Lei Wang / Guangfeng Liu / Zhaoning Wang / Duo Jin / Tong Cheng / Hongxia Wang / Yuan Tian / Xiangxi Wang / Lei Sun / Xiaolan Zhao / ...Authors: Qian Li / Jun Zhang / Cory Haluska / Xiang Zhang / Lei Wang / Guangfeng Liu / Zhaoning Wang / Duo Jin / Tong Cheng / Hongxia Wang / Yuan Tian / Xiangxi Wang / Lei Sun / Xiaolan Zhao / Zhenguo Chen / Lanfeng Wang /   Abstract: Smc5/6 is a member of the eukaryotic structural maintenance of chromosomes (SMC) family of complexes with important roles in genome maintenance and viral restriction. However, limited structural ...Smc5/6 is a member of the eukaryotic structural maintenance of chromosomes (SMC) family of complexes with important roles in genome maintenance and viral restriction. However, limited structural understanding of Smc5/6 hinders the elucidation of its diverse functions. Here, we report cryo-EM structures of the budding yeast Smc5/6 complex in eight-subunit, six-subunit and five-subunit states. Structural maps throughout the entire length of these complexes reveal modularity and key elements in complex assembly. We show that the non-SMC element (Nse)2 subunit supports the overall shape of the complex and uses a wedge motif to aid the stability and function of the complex. The Nse6 subunit features a flexible hook region for attachment to the Smc5 and Smc6 arm regions, contributing to the DNA repair roles of the complex. Our results also suggest a structural basis for the opposite effects of the Nse1-3-4 and Nse5-6 subcomplexes in regulating Smc5/6 ATPase activity. Collectively, our integrated structural and functional data provide a framework for understanding Smc5/6 assembly and function. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_33927.map.gz emd_33927.map.gz | 9.8 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-33927-v30.xml emd-33927-v30.xml emd-33927.xml emd-33927.xml | 19.7 KB 19.7 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_33927.png emd_33927.png | 79.3 KB | ||
| Filedesc metadata |  emd-33927.cif.gz emd-33927.cif.gz | 6.6 KB | ||
| Others |  emd_33927_half_map_1.map.gz emd_33927_half_map_1.map.gz emd_33927_half_map_2.map.gz emd_33927_half_map_2.map.gz | 7.9 MB 7.9 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33927 http://ftp.pdbj.org/pub/emdb/structures/EMD-33927 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33927 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33927 | HTTPS FTP |
-Related structure data
| Related structure data |  7ymdMC  7ylmC  7yqhC  8hqsC  8i13C  8i21C  8i4uC  8i4vC  8i4wC  8i4xC  8wjlC  8wjnC  8wjoC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_33927.map.gz / Format: CCP4 / Size: 10.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_33927.map.gz / Format: CCP4 / Size: 10.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.044 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #2
| File | emd_33927_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
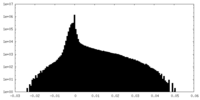
| Projections & Slices |
| ||||||||||||
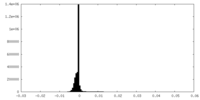
| Density Histograms |
-Half map: #1
| File | emd_33927_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
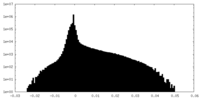
| Projections & Slices |
| ||||||||||||
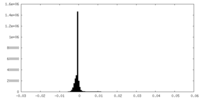
| Density Histograms |
- Sample components
Sample components
-Entire : Cryo-EM structure of Nse1/3/4
| Entire | Name: Cryo-EM structure of Nse1/3/4 |
|---|---|
| Components |
|
-Supramolecule #1: Cryo-EM structure of Nse1/3/4
| Supramolecule | Name: Cryo-EM structure of Nse1/3/4 / type: complex / ID: 1 / Parent: 0 / Macromolecule list: all |
|---|---|
| Source (natural) | Organism:  |
-Macromolecule #1: Non-structural maintenance of chromosome element 4
| Macromolecule | Name: Non-structural maintenance of chromosome element 4 / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 46.195945 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MSSTVISRKR RNSTVTEPDS SGETRKQKKS RSDEKSSSSK DGDPQLEFKV LQGYRDLESE MHKGRAQVTR TGDIGVAMDN LNAVDSLFN KVIGIKNNGL FAHDARAMVS ISELAQISVR NLKFDDSRSM VNLENIVNSL KRYMLKEHFK LNNIAENRND L TLAADEQS ...String: MSSTVISRKR RNSTVTEPDS SGETRKQKKS RSDEKSSSSK DGDPQLEFKV LQGYRDLESE MHKGRAQVTR TGDIGVAMDN LNAVDSLFN KVIGIKNNGL FAHDARAMVS ISELAQISVR NLKFDDSRSM VNLENIVNSL KRYMLKEHFK LNNIAENRND L TLAADEQS AADQQEESDG DIDRTPDDNH TDKATSSFKA TSMRHSYLQQ FSHYNEFSQF NWFRIGALYN TISKNAPITD HL MGPLSIE KKPRVLTQRR RNNDQVGEKI TAEKITQHSL NSTQQETTPE QVKKCFKKLS KKLGPEGSIN LFKFIIDPNS FSR SIENLF YTSFLIKEGK LLMEHDEEGL PTIKIKQSIS HTDSRSKEIE RQRRRAAHQN HIIFQMDMPT WRKLIKKYNI TSPF LD UniProtKB: Non-structural maintenance of chromosome element 4 |
-Macromolecule #2: Non-structural maintenance of chromosomes element 1
| Macromolecule | Name: Non-structural maintenance of chromosomes element 1 / type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO / EC number: RING-type E3 ubiquitin transferase |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 38.373387 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MEVHEEQVSA PVTGDATAKY LLQYILSARG ICHENALILA LMRLETDAST LNTEWSIQQW VDKLNDYINA INVKLNLLGY KIIRINHGI GRNAVTLKAK QNFESFEDNT AIRAHNNDYA VLQSIVLPES NRFFVYVNLA STEETKLATR FNQNEIEFMK W AIEQFMIS ...String: MEVHEEQVSA PVTGDATAKY LLQYILSARG ICHENALILA LMRLETDAST LNTEWSIQQW VDKLNDYINA INVKLNLLGY KIIRINHGI GRNAVTLKAK QNFESFEDNT AIRAHNNDYA VLQSIVLPES NRFFVYVNLA STEETKLATR FNQNEIEFMK W AIEQFMIS GETIVEGPAL ETSIIVKEVN RILVAATGDS NLAKWRKFST FTVGSTNLFQ FQELTATDIE DLLLRLCELK WF YRTQEGK FGIDLRCIAE LEEYLTSMYN LNTCQNCHKL AIQGVRCGNE SCREENEETG ENSLSQIWHV DCFKHYITHV SKN CDRCGS SLITEGVYVI UniProtKB: Non-structural maintenance of chromosomes element 1 |
-Macromolecule #3: Non-structural maintenance of chromosome element 3
| Macromolecule | Name: Non-structural maintenance of chromosome element 3 / type: protein_or_peptide / ID: 3 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 34.005531 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MSSIDNDSDV DLTEDLAVAK IVKENPVARK MVRYILSRGE SQNSIITRNK LQSVIHEAAR EENIAKPSFS KMFMDINAIL YNVYGFELQ GLPSKNNMNA GGNGSNSNTN KSMPEPLGHR AQKFILLNNV PHSKNFDDFK ILQSAHTYEE LIVTGEYIGD D IASGTSNT ...String: MSSIDNDSDV DLTEDLAVAK IVKENPVARK MVRYILSRGE SQNSIITRNK LQSVIHEAAR EENIAKPSFS KMFMDINAIL YNVYGFELQ GLPSKNNMNA GGNGSNSNTN KSMPEPLGHR AQKFILLNNV PHSKNFDDFK ILQSAHTYEE LIVTGEYIGD D IASGTSNT LESKLSTDRD LVYKGVLSVI LCIVFFSKNN ILHQELIKFL ETFGIPSDGS KIAILNITIE DLIKSLEKRE YI VRLEEKS DTDGEVISYR IGRRTQAELG LESLEKLVQE IMGLEKEQTK SLHDDIIKSI GDSYSI UniProtKB: Non-structural maintenance of chromosome element 3 |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 8 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: SPOT SCAN / Imaging mode: DARK FIELD / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.5 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION |
|---|---|
| Startup model | Type of model: OTHER |
| Final reconstruction | Resolution.type: BY AUTHOR / Resolution: 4.176 Å / Resolution method: FSC 0.143 CUT-OFF / Number images used: 610732 |
| Initial angle assignment | Type: PROJECTION MATCHING |
 Movie
Movie Controller
Controller



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)