[English] 日本語
 Yorodumi
Yorodumi- EMDB-22816: Full-length human mitochondrial Hsp90 (TRAP1) in complex with Sdh... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-22816 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Full-length human mitochondrial Hsp90 (TRAP1) in complex with SdhB in the presence of AMP-PNP | |||||||||||||||||||||
 Map data Map data | ||||||||||||||||||||||
 Sample Sample |
| |||||||||||||||||||||
 Keywords Keywords | Hsp90 / CHAPERONE / TRAP1 / SdhB / mitochondria | |||||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationtranslational attenuation / negative regulation of cellular respiration / : / mitochondrial electron transport, succinate to ubiquinone / succinate dehydrogenase / succinate dehydrogenase (quinone) activity / Citric acid cycle (TCA cycle) / Respiratory electron transport / tumor necrosis factor receptor binding / negative regulation of intrinsic apoptotic signaling pathway in response to hydrogen peroxide ...translational attenuation / negative regulation of cellular respiration / : / mitochondrial electron transport, succinate to ubiquinone / succinate dehydrogenase / succinate dehydrogenase (quinone) activity / Citric acid cycle (TCA cycle) / Respiratory electron transport / tumor necrosis factor receptor binding / negative regulation of intrinsic apoptotic signaling pathway in response to hydrogen peroxide / 3 iron, 4 sulfur cluster binding / proton motive force-driven mitochondrial ATP synthesis / ubiquinone binding / chaperone-mediated protein folding / negative regulation of reactive oxygen species biosynthetic process / tricarboxylic acid cycle / aerobic respiration / respiratory electron transport chain / cell periphery / mitochondrial membrane / ATP-dependent protein folding chaperone / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / unfolded protein binding / protein folding / 4 iron, 4 sulfur cluster binding / mitochondrial inner membrane / electron transfer activity / mitochondrial matrix / protein kinase binding / ATP hydrolysis activity / mitochondrion / RNA binding / nucleoplasm / ATP binding / membrane / metal ion binding / plasma membrane Similarity search - Function | |||||||||||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||||||||||||||
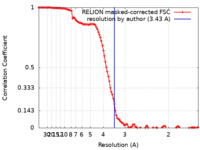
| Method | single particle reconstruction / cryo EM / Resolution: 3.43 Å | |||||||||||||||||||||
 Authors Authors | Liu YX / Agard DA | |||||||||||||||||||||
| Funding support |  United States, 6 items United States, 6 items
| |||||||||||||||||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Cryo-EM reveals the dynamic interplay between mitochondrial Hsp90 and SdhB folding intermediates Authors: Liu YX / Agard DA / Elnatan D / Sun M / Myasnikov AG | |||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_22816.map.gz emd_22816.map.gz | 117.1 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-22816-v30.xml emd-22816-v30.xml emd-22816.xml emd-22816.xml | 15.6 KB 15.6 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_22816_fsc.xml emd_22816_fsc.xml | 11.5 KB | Display |  FSC data file FSC data file |
| Images |  emd_22816.png emd_22816.png | 98.9 KB | ||
| Filedesc metadata |  emd-22816.cif.gz emd-22816.cif.gz | 6.2 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22816 http://ftp.pdbj.org/pub/emdb/structures/EMD-22816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22816 | HTTPS FTP |
-Validation report
| Summary document |  emd_22816_validation.pdf.gz emd_22816_validation.pdf.gz | 613.9 KB | Display |  EMDB validaton report EMDB validaton report |
|---|---|---|---|---|
| Full document |  emd_22816_full_validation.pdf.gz emd_22816_full_validation.pdf.gz | 613.4 KB | Display | |
| Data in XML |  emd_22816_validation.xml.gz emd_22816_validation.xml.gz | 12.2 KB | Display | |
| Data in CIF |  emd_22816_validation.cif.gz emd_22816_validation.cif.gz | 16.3 KB | Display | |
| Arichive directory |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22816 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22816 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22816 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22816 | HTTPS FTP |
-Related structure data
| Related structure data |  7kcmMC  7kckC  7kclC C: citing same article ( M: atomic model generated by this map |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_22816.map.gz / Format: CCP4 / Size: 125 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_22816.map.gz / Format: CCP4 / Size: 125 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Voxel size | X=Y=Z: 0.814 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : human Trap1 in complex with SdhB in the presence of AMP-PNP
| Entire | Name: human Trap1 in complex with SdhB in the presence of AMP-PNP |
|---|---|
| Components |
|
-Supramolecule #1: human Trap1 in complex with SdhB in the presence of AMP-PNP
| Supramolecule | Name: human Trap1 in complex with SdhB in the presence of AMP-PNP type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#2 |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 162 KDa |
-Macromolecule #1: Heat shock protein 75 kDa, mitochondrial
| Macromolecule | Name: Heat shock protein 75 kDa, mitochondrial / type: protein_or_peptide / ID: 1 / Number of copies: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 74.17757 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: GIDPFTSTQT AEDKEEPLHS IISSTESVQG STSKHEFQAE TKKLLDIVAR SLYSEKEVFI RELISNASDA LEKLRHKLVS DGQALPEME IHLQTNAEKG TITIQDTGIG MTQEELVSNL GTIARSGSKA FLDALQNQAE ASSKIIGQFG VGFYSAFMVA D RVEVYSRS ...String: GIDPFTSTQT AEDKEEPLHS IISSTESVQG STSKHEFQAE TKKLLDIVAR SLYSEKEVFI RELISNASDA LEKLRHKLVS DGQALPEME IHLQTNAEKG TITIQDTGIG MTQEELVSNL GTIARSGSKA FLDALQNQAE ASSKIIGQFG VGFYSAFMVA D RVEVYSRS AAPGSLGYQW LSDGSGVFEI AEASGVRTGT KIIIHLKSDC KEFSSEARVR DVVTKYSNFV SFPLYLNGRR MN TLQAIWM MDPKDVGEWQ HEEFYRYVAQ AHDKPRYTLH YKTDAPLNIR SIFYVPDMKP SMFDVSRELG SSVALYSRKV LIQ TKATDI LPKWLRFIRG VVDSEDIPLN LSRELLQESA LIRKLRDVLQ QRLIKFFIDQ SKKDAEKYAK FFEDYGLFMR EGIV TATEQ EVKEDIAKLL RYESSALPSG QLTSLSEYAS RMRAGTRNIY YLCAPNRHLA EHSPYYEAMK KKDTEVLFCF EQFDE LTLL HLREFDKKKL ISVETDIVVD HYKEEKFEDR SPAAECLSEK ETEELMAWMR NVLGSRVTNV KVTLRLDTHP AMVTVL EMG AARHFLRMQQ LAKTQEERAQ LLQPTLEINP RHALIKKLNQ LRASEPGLAQ LLVDQIYENA MIAAGLVDDP RAMVGRL NE LLVKALERH UniProtKB: Heat shock protein 75 kDa, mitochondrial |
-Macromolecule #2: Succinate dehydrogenase [ubiquinone] iron-sulfur subunit, mitocho...
| Macromolecule | Name: Succinate dehydrogenase [ubiquinone] iron-sulfur subunit, mitochondrial type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 15.097539 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: GSAQTAAATA PRIKKFAIYR WDPDKAGDKP HMQTYEVDLN KCGPMVLDAL IKIKNEVDST LTFRRSCREG ICGSCAMNIN GGNTLACTR RIDTNLNKVS KIYPLPHMYV IKDLVPDLSN FYAQYKSIEP YLKKK UniProtKB: Succinate dehydrogenase [ubiquinone] iron-sulfur subunit, mitochondrial |
-Macromolecule #3: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| Macromolecule | Name: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / type: ligand / ID: 3 / Number of copies: 2 / Formula: ANP |
|---|---|
| Molecular weight | Theoretical: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-Macromolecule #4: MAGNESIUM ION
| Macromolecule | Name: MAGNESIUM ION / type: ligand / ID: 4 / Number of copies: 2 / Formula: MG |
|---|---|
| Molecular weight | Theoretical: 24.305 Da |
-Macromolecule #5: POTASSIUM ION
| Macromolecule | Name: POTASSIUM ION / type: ligand / ID: 5 / Number of copies: 2 / Formula: K |
|---|---|
| Molecular weight | Theoretical: 39.098 Da |
-Macromolecule #6: FE2/S2 (INORGANIC) CLUSTER
| Macromolecule | Name: FE2/S2 (INORGANIC) CLUSTER / type: ligand / ID: 6 / Number of copies: 1 / Formula: FES |
|---|---|
| Molecular weight | Theoretical: 175.82 Da |
| Chemical component information |  ChemComp-FES: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Grid | Model: Quantifoil / Material: COPPER / Mesh: 300 / Support film - Material: CARBON / Support film - topology: HOLEY |
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Detector mode: SUPER-RESOLUTION / Average electron dose: 72.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Initial model |
| ||||||
|---|---|---|---|---|---|---|---|
| Refinement | Space: REAL / Protocol: FLEXIBLE FIT | ||||||
| Output model |  PDB-7kcm: |
 Movie
Movie Controller
Controller