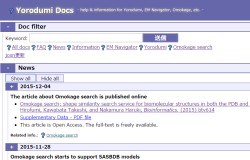
-表示する文書の検索と絞り込み
 ありがちな質問と回答,
ありがちな質問と回答,  お知らせ,
お知らせ,  解説,
解説,  EM Navigator,
EM Navigator,  万見,
万見,  Omokage検索,
Omokage検索,  サービス一覧,
サービス一覧,  すべての文書
すべての文書
-お知らせ (10)
+2022年2月9日: EMDBエントリの付随情報ファイルのフォーマットが新しくなりました
EMDBエントリの付随情報ファイルのフォーマットが新しくなりました
- EMDBのヘッダファイルのバージョン3が、公式のフォーマットとなりました。
- これまでは公式だったバージョン1.9は、アーカイブから削除されます。
関連情報: EMDBヘッダ
EMDBヘッダ
+2020年8月12日: 新型コロナ情報
新型コロナ情報

URL: https://pdbj.org/emnavi/covid19.php
新ページ: EM Navigatorに新型コロナウイルスの特設ページを開設しました。
関連情報: Covid-19情報 /
Covid-19情報 /  2020年3月5日: 新型コロナウイルスの構造データ
2020年3月5日: 新型コロナウイルスの構造データ
+2020年3月5日: 新型コロナウイルスの構造データ
新型コロナウイルスの構造データ
- 国際ウイルス分類委員会(ICTV)は、新型コロナウイルス感染症(COVID-19)の病原ウイルスの略称を「SARS-CoV-2」と決定しました。
- 万見で扱っている構造データベースでは、"COVID-19 virus"、"2019-nCoV"といった別称・仮称でも登録されています。こちらでウイルスの詳細と構造データの一覧を見られます。
関連情報: 万見生物種 /
万見生物種 /  2020年8月12日: 新型コロナ情報
2020年8月12日: 新型コロナ情報
+2019年1月31日: EMDBのIDの桁数の変更
EMDBのIDの桁数の変更
- EMDBエントリに付与されているアクセスコード(EMDB-ID)は4桁の数字(例、EMD-1234)でしたが、間もなく枯渇します。これまでの4桁のID番号は4桁のまま変更されませんが、4桁の数字を使い切った後に発行されるIDは5桁以上の数字(例、EMD-12345)になります。5桁のIDは2019年の春頃から発行される見通しです。
- EM Navigator/万見では、接頭語「EMD-」は省略されています。
関連情報: Q: 「EMD」とは何ですか? /
Q: 「EMD」とは何ですか? /  万見/EM NavigatorにおけるID/アクセスコードの表記
万見/EM NavigatorにおけるID/アクセスコードの表記
+2017年7月12日: PDB大規模アップデート
PDB大規模アップデート
- 新バージョンのPDBx/mmCIF辞書形式に基づくデータがリリースされました。
- 今回の更新はバージョン番号が4から5になる大規模なもので、全エントリデータの書き換えが行われる「Remediation」というアップデートに該当します。
- このバージョンアップで、電子顕微鏡の実験手法に関する多くの項目の書式が改定されました(例:em_softwareなど)。
- EM NavigatorとYorodumiでも、この改定に基づいた表示内容になります。
外部リンク: wwPDB Remediation /
wwPDB Remediation /  OneDepデータ基準に準拠した、より強化された内容のモデル構造ファイルが、PDBアーカイブで公開されました。
OneDepデータ基準に準拠した、より強化された内容のモデル構造ファイルが、PDBアーカイブで公開されました。
+2017年6月16日: Omokage検索で絞り込み
Omokage検索で絞り込み
Omokage検索の結果をキーワードとデータベースの種類で絞り込むことができるようになりました。
関連情報: Omokage検索
Omokage検索
+2016年9月15日: 新しくなったEM Navigatorと万見
新しくなったEM Navigatorと万見
EM Navigatorと万見を刷新しました
関連情報: 新しいEM Navigatorと万見の変更点
新しいEM Navigatorと万見の変更点
+2016年8月31日: 新しいEM Navigatorと万見
新しいEM Navigatorと万見
- これまで開発版として公開していたEM Navigatorと万見が、9月15日から正式版となります。
- 現行版も「旧版」としてしばらく公開を継続します。
関連情報: 新しいEM Navigatorと万見の変更点 /
新しいEM Navigatorと万見の変更点 /  EM Navigator /
EM Navigator /  万見 (Yorodumi)
万見 (Yorodumi)
+2016年4月13日: Omokage検索が速くなりました
Omokage検索が速くなりました
- データアクセスプロセスを見直し、計算時間をこれまでの半分程度に短縮しました。
- これまで以上に、生体分子の「カタチの類似性」をお楽しみください!
関連情報: Omokage検索
Omokage検索
+2016年3月3日: 2月19日の蛋白研セミナーのプレゼンテーション(PDFファイル)
2月19日の蛋白研セミナーのプレゼンテーション(PDFファイル)
外部リンク: 蛋白研セミナー・2016年2月19日 /
蛋白研セミナー・2016年2月19日 /  プレゼンテーション(PDF)
プレゼンテーション(PDF)
-ありがちな質問と回答 (3)
+Q: 構造データの画像はどうやって作っているのですか?色はどういう意味があるのですか?
A: EMDBの画像はEM Navigatorの画像、PDBデータの画像は万見の画像です。色付けは複数のパターンがあります。
詳細はについては、それぞれの項目をご覧ください。
関連情報: Q: EM Navigatorのムービーはどうやって作っているのですか? /
Q: EM Navigatorのムービーはどうやって作っているのですか? /  Q: 万見やOmokageのEMDBの画像は、どうやって作っていますか? /
Q: 万見やOmokageのEMDBの画像は、どうやって作っていますか? /  Q: 万見やOmokageのPDBの画像は、どうやって作っていますか?
Q: 万見やOmokageのPDBの画像は、どうやって作っていますか?
+Q: 万見やOmokageのEMDBの画像は、どうやって作っていますか?
A: Chimeraを利用して半自動的に作成しています
- 方法:
- EM Navigatoの構造エントリの動画・画像は、動画はUSCF Chimeraを利用して半自動的に作成しています。
- 方向:
- 手作業で、そのデータに相応しい方向(論文が出版・発行されている場合はその図を参考にします)、あるいは同様の構造がある場合は極力それに似た方向になるようにしています。
- 正20面体対称の構造体の場合は、一般的な描画では2回対称軸に沿った方向にしますが、EM Navigatorでは5回対称軸を縦に向けています。これは尾構造を持つバクテリオファージなどに見られる擬似的な正20面体対称構造と方向を統一するためです。
- 色:
- EM Navigatorでは、ほとんどのデータに対して、着色したものと単色の画像を用意していますが、Omokage検索では着色したものが表示されます。
- 色は中心からの距離か、円筒半径か、あるいはある軸に沿った「高さ」によって決まります。個々の動画の着色法ついては、データの詳細ページに表示されています。
関連情報: Q: 万見やOmokageのPDBの画像は、どうやって作っていますか? /
Q: 万見やOmokageのPDBの画像は、どうやって作っていますか? /  Q: EM Navigatorのムービーはどうやって作っているのですか? /
Q: EM Navigatorのムービーはどうやって作っているのですか? /  Q: 構造データの画像はどうやって作っているのですか?色はどういう意味があるのですか?
Q: 構造データの画像はどうやって作っているのですか?色はどういう意味があるのですか?
外部リンク: UCSF Chimera Home Page
UCSF Chimera Home Page
+Q: 万見やOmokageのPDBの画像は、どうやって作っていますか?
A: Jmolを利用して全自動で作成しています。スタイルはデータの種類によります。
- 方向:
- 正20面体対称構造: そのままの方向
- らせん対称構造: まず6方向(+/- X,Y,Z 方向)からの画像をJPEG形式で作成し、ファイルのサイズが一番大きかったものを採用しています。(同じサイズの画像のJPEG形式のファイルのサイズは、圧縮の難しさ、つまり画像の複雑さによるからです)
- その他: Jmolの"roate best"コマンドを利用
- リボソーム: Jmolの"roate best"コマンドを利用(ただしRNAの座標のみ考慮)
- 色:
- モノマーのAU: 配列順による虹色 (N->C, 青->赤)
- モノマーのAUのBU: Jmolの"color molecule"
- マルチマーのAUと、そのBU (BUがモノマーの場合も含む): "color chain" (Jmolの "color chain"は虹色着色ではなく、鎖IDごとに定められた色による着色です)
- (ここでいうモノマーとは、DNAかRNAかポリペプチドの鎖が一つしかない構造です)
- 問題点:
- 集合体のモデル作成はJmolのシステムを利用しています。ほとんどのデータについてはうまく動作していますが、一部うまく作成できていないものがあります。
- 現在、画像のチェックと再作成を進めています。正しく描画されていない画像が残っています。
関連情報: Q: 万見やOmokageのEMDBの画像は、どうやって作っていますか?
Q: 万見やOmokageのEMDBの画像は、どうやって作っていますか?
外部リンク: Jmol: an open-source Java viewer for chemical structures in 3D
Jmol: an open-source Java viewer for chemical structures in 3D
-解説 (15)
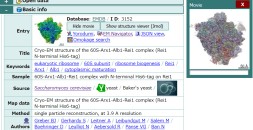
+万見 (Yorodumi)
幾万の構造データを、幾万の視点から
URL: https://pdbj.org/emnavi/quick.php
- 万見(Yorodumi)は、EMDB/PDB/SASBDBなどの構造データを閲覧するためのページです。
- EM Navigatorの詳細ページの後継、Omokage検索のフロントエンドも兼ねています。
関連情報: EMDB /
EMDB /  PDB /
PDB /  SASBDB /
SASBDB /  3つのデータバンクの比較 /
3つのデータバンクの比較 /  万見検索 /
万見検索 /  2016年8月31日: 新しいEM Navigatorと万見 /
2016年8月31日: 新しいEM Navigatorと万見 /  万見文献 /
万見文献 /  Jmol/JSmol /
Jmol/JSmol /  機能・相同性情報 /
機能・相同性情報 /  新しいEM Navigatorと万見の変更点
新しいEM Navigatorと万見の変更点
+万見検索
EMDB/PDB/SASBDBなどの横断検索
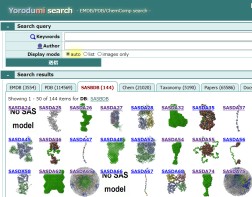
URL: https://pdbj.org/emnavi/ysearch.php
関連情報: EMDB /
EMDB /  PDB /
PDB /  SASBDB /
SASBDB /  3つのデータバンクの比較 /
3つのデータバンクの比較 /  EMN検索 /
EMN検索 /  万見 (Yorodumi) /
万見 (Yorodumi) /  実験手法・装置(施設・設備・機器)・ソフトウェアのデータ /
実験手法・装置(施設・設備・機器)・ソフトウェアのデータ /  機能・相同性情報 /
機能・相同性情報 /  新しいEM Navigatorと万見の変更点
新しいEM Navigatorと万見の変更点
+万見文献
EMDB/PDB/SASBDBから引用されている文献のデータベース
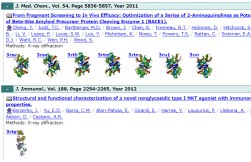
URL: https://pdbj.org/emnavi/pap.php
- EMDB/PDB/SASBDBのエントリから引用されている文献のデータベースです
- Pubmedのデータを利用しています
関連情報: EMDB /
EMDB /  PDB /
PDB /  SASBDB /
SASBDB /  万見 (Yorodumi) /
万見 (Yorodumi) /  EMN文献 /
EMN文献 /  新しいEM Navigatorと万見の変更点
新しいEM Navigatorと万見の変更点
+万見生物種
EMDB/PDB/SASBDBの生物種情報
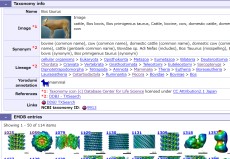
URL: https://pdbj.org/emnavi/taxo.php
EMDB/PDB/SASBDBの構造データの試料情報、由来する生物種に関するデータベース
関連情報: EMDB /
EMDB /  PDB /
PDB /  SASBDB /
SASBDB /  3つのデータバンクの比較 /
3つのデータバンクの比較 /  2020年3月5日: 新型コロナウイルスの構造データ
2020年3月5日: 新型コロナウイルスの構造データ
+万見文書
+Covid-19情報
+F&H 検索
機能・相同性の類似性検索
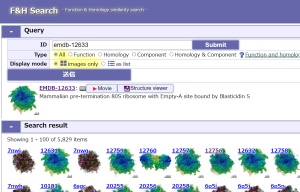
URL: https://pdbj.org/emnavi/fh-search.php
構造データに関連付けられた機能・相同性アイテムの類似性に基づき、類似するデータを構造データベースから検索します。
関連情報: 機能・相同性情報
機能・相同性情報
+Jmol/JSmol
オープンソースの3次元化学構造ビューア
- マウス・タッチ操作
X-Y回転 X-Y移動 拡大・縮小 マウス操作 左ボタンドラッグ [Ctrl] + 右ボタンドラッグ 中ボタンドラッグ(縦方向) ・ ホイール回転 タッチ操作 1本指 2本指 ピンチ(つまむ動作) - Jmolメニュー: [Ctrl] + クリック / 右クリック
関連情報: Molmil /
Molmil /  万見 (Yorodumi)
万見 (Yorodumi)
外部リンク: Jmol: an open-source Java viewer for chemical structures in 3D
Jmol: an open-source Java viewer for chemical structures in 3D
+Molmil
WebGLを利用した分子構造ビューア
- PCだけでなく、スマートフォンやタブレットでも動作します。(WebGLと呼ばれる仕組みを利用しています)
- マウス・タッチ操作
X-Y回転 X-Y移動 拡大・縮小 マウス操作 左ボタンドラッグ 中ボタンドラッグ / [Shift] + 左ボタンドラッグ 右ボタンドラッグ(縦方向) ・ ホイール回転 タッチ操作 1本指 _ ピンチ(つまむ動作) - 機能メニュー: ビューアースクリーンの左上部ボタン
関連情報: Jmol/JSmol /
Jmol/JSmol /  SurfView /
SurfView /  新しいEM Navigatorと万見の変更点
新しいEM Navigatorと万見の変更点
外部リンク: Documentation of Molmil in PDBj site /
Documentation of Molmil in PDBj site /  Molmil page in GitHub
Molmil page in GitHub
+糖鎖の表現
PDBにおける糖鎖・炭水化物の情報の表現について
- 2020年7月にPDBでの糖鎖データの表現について、大規模な更新が実施されました。
- 糖鎖の画像はGlycanBuilder2で作成されています。
- その他、詳細は下記のリンクを参照してください。
外部リンク: Implementation of GlycanBuilder to draw a wide variety of ambiguous glycans - ScienceDirect /
Implementation of GlycanBuilder to draw a wide variety of ambiguous glycans - ScienceDirect /  GlycanBuilder2 - RINGS (Resource For INformatics Of Glycomes at Soka) /
GlycanBuilder2 - RINGS (Resource For INformatics Of Glycomes at Soka) /  Carbohydrate Remediation - wwPDB documentation /
Carbohydrate Remediation - wwPDB documentation /  7月29日に糖鎖分子の表現を改善したPDBデータを公開します - PDBjお知らせ
7月29日に糖鎖分子の表現を改善したPDBデータを公開します - PDBjお知らせ
+実験手法・装置(施設・設備・機器)・ソフトウェアのデータ
EMDB・PDB・SABDBから収集した実験情報のデータベース
URL: https://pdbj.org/emnavi/ysearch.php?&act_tab=met
関連情報: 3つのデータバンクの比較 /
3つのデータバンクの比較 /  万見検索
万見検索
+機能・相同性情報
関連データベースから収集した分子機能・ドメイン・相同性などの情報
- 万見と万見検索では、構造データの機能の理解や類似構造データの検索に役立つように、分子機能や相同性に関するデータベースの情報を表示・活用しています。
- EMDBのヘッダXML、PDBx/mmcif、sasCIFの公式のデータに含まれるEC、EMBL、GeneBank、GO、InterPro、UniProtなどの情報に加えて、PDBMLplus経由、EMDB-PDBフィッティングデータ経由、UniProt経由で収集された、Pfam、PROSITE、Reactome、UniProtKBなどの情報が整理されています。
カテゴリ データベース名・定義名 分子機能 Gene ontology, Enzyme Commission number, Reactome, etc. ドメイン・相同性 CATH, InterPro, SMART, etc. 構成要素 UniProt, GenBank, PDB chemical component, etc.
関連情報: F&H 検索 /
F&H 検索 /  PDBML-plusからの情報 /
PDBML-plusからの情報 /  万見検索 /
万見検索 /  万見 (Yorodumi) /
万見 (Yorodumi) /  F&H 検索
F&H 検索
+新しいEM Navigatorと万見の変更点
新しいEM Navigatorと万見の変更点(2016年9月)
- 変更点
- ページ外観や操作性を刷新しました。新しい「万見」と「Omokage検索」と共通化し、PCだけでなくモバイル機器にも対応しました。
- EM Navigatorの個別のデータエントリのページ(旧版の「詳細ページ」)は、新しい「万見」が受け持ちます。「Omokage検索」のフロントエンド・詳細情報表示も兼ねた、EMDB、PDB、SASBDB用の共通の詳細ページです。
- 構造ビューア、ムービービューアは個別のポップアップウインドウに表示されます。PCでは一度に複数のビューアウインドウの表示が可能です。モバイル機器ではタッチ操作にも対応しています。
- 新しい機能
- 3次元構造の閲覧に、分子構造ビューア「Molmil」と、EMDBマップ表面モデルビューア「SurfView」が利用できるようになりました。
- 万見はSASBDBのデータエントリの表示に対応しました。
- その他新しいページ (引用文献のデータベース「万見文献」「EMN文献」、キーワードによる横断検索「万見検索」など)
- 旧版のページも、当面は継続します。
関連情報: EM Navigator /
EM Navigator /  万見 (Yorodumi) /
万見 (Yorodumi) /  SurfView /
SurfView /  Molmil /
Molmil /  ムービービューア /
ムービービューア /  EMN文献 /
EMN文献 /  万見文献 /
万見文献 /  万見検索 /
万見検索 /  2016年8月31日: 新しいEM Navigatorと万見
2016年8月31日: 新しいEM Navigatorと万見
+PDBML-plusからの情報
PDBMLplusはXML形式のPDBデータフォーマットである「PDBML」のデータに対し、個々の分子に関する情報をPDBjで独自に追加したものです。
関連情報: PDBj /
PDBj /  機能・相同性情報
機能・相同性情報
+万見注釈
万見・EM Navigatorの管理者による注釈
関連情報: Q: この文書を書いているのは誰ですか?EM Navigator、万見、Omokage検索などの開発者は?
Q: この文書を書いているのは誰ですか?EM Navigator、万見、Omokage検索などの開発者は?
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア