| 登録情報 | データベース: PDB / ID: 2kkf
|
|---|
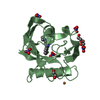
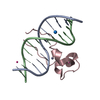
| タイトル | Solution structure of MLL CXXC domain in complex with palindromic CPG DNA |
|---|
 要素 要素 | - 5'-D(*CP*CP*CP*TP*GP*CP*GP*CP*AP*GP*GP*G)-3'
- Histone-lysine N-methyltransferase HRX
|
|---|
 キーワード キーワード | DNA BINDING PROTEIN/DNA /  PROTEIN-DNA COMPLEX (デオキシリボ核酸) / CXXC DOMAIN / MLL / CPG DNA / PROTEIN-DNA COMPLEX (デオキシリボ核酸) / CXXC DOMAIN / MLL / CPG DNA /  CHROMOSOMAL REARRANGEMENT / DNA-BINDING / METAL-BINDING / CHROMOSOMAL REARRANGEMENT / DNA-BINDING / METAL-BINDING /  NUCLEUS (細胞核) / NUCLEUS (細胞核) /  ZINC-FINGER (ジンクフィンガー) / DNA BINDING PROTEIN-DNA COMPLEX / ZINC-FINGER (ジンクフィンガー) / DNA BINDING PROTEIN-DNA COMPLEX /  Alternative splicing (選択的スプライシング) / Alternative splicing (選択的スプライシング) /  Apoptosis (アポトーシス) / Apoptosis (アポトーシス) /  Bromodomain (ブロモドメイン) / Chromatin regulator / Bromodomain (ブロモドメイン) / Chromatin regulator /  Isopeptide bond / Isopeptide bond /  Methyltransferase (メチルトランスフェラーゼ) / Methyltransferase (メチルトランスフェラーゼ) /  Phosphoprotein / Polymorphism / Phosphoprotein / Polymorphism /  Proto-oncogene (がん遺伝子) / Proto-oncogene (がん遺伝子) /  S-adenosyl-L-methionine (S-アデノシルメチオニン) / S-adenosyl-L-methionine (S-アデノシルメチオニン) /  Transcription (転写 (生物学)) / Transcription (転写 (生物学)) /  Transcription regulation / Transcription regulation /  Transferase (転移酵素) / Ubl conjugation / Transferase (転移酵素) / Ubl conjugation /  Zinc (亜鉛) Zinc (亜鉛) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
protein-cysteine methyltransferase activity / response to potassium ion / [histone H3]-lysine4 N-methyltransferase / histone H3K4 monomethyltransferase activity / unmethylated CpG binding / histone H3K4 trimethyltransferase activity / negative regulation of DNA methylation-dependent heterochromatin formation / regulation of short-term neuronal synaptic plasticity / definitive hemopoiesis / histone H3K4 methyltransferase activity ...protein-cysteine methyltransferase activity / response to potassium ion / [histone H3]-lysine4 N-methyltransferase / histone H3K4 monomethyltransferase activity / unmethylated CpG binding / histone H3K4 trimethyltransferase activity / negative regulation of DNA methylation-dependent heterochromatin formation / regulation of short-term neuronal synaptic plasticity / definitive hemopoiesis / histone H3K4 methyltransferase activity / T-helper 2 cell differentiation / embryonic hemopoiesis / exploration behavior / anterior/posterior pattern specification /  histone methyltransferase complex / histone methyltransferase complex /  : / Formation of WDR5-containing histone-modifying complexes / minor groove of adenine-thymine-rich DNA binding / : / Formation of WDR5-containing histone-modifying complexes / minor groove of adenine-thymine-rich DNA binding /  脱分極 / 脱分極 /  MLL1 complex / negative regulation of fibroblast proliferation / homeostasis of number of cells within a tissue / spleen development / transcription initiation-coupled chromatin remodeling / cellular response to transforming growth factor beta stimulus / MLL1 complex / negative regulation of fibroblast proliferation / homeostasis of number of cells within a tissue / spleen development / transcription initiation-coupled chromatin remodeling / cellular response to transforming growth factor beta stimulus /  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / post-embryonic development / circadian regulation of gene expression / protein modification process / lysine-acetylated histone binding / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / post-embryonic development / circadian regulation of gene expression / protein modification process / lysine-acetylated histone binding / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function /  visual learning / PKMTs methylate histone lysines / Transcriptional regulation of granulopoiesis / RUNX1 regulates transcription of genes involved in differentiation of HSCs / fibroblast proliferation / protein-containing complex assembly / apoptotic process / visual learning / PKMTs methylate histone lysines / Transcriptional regulation of granulopoiesis / RUNX1 regulates transcription of genes involved in differentiation of HSCs / fibroblast proliferation / protein-containing complex assembly / apoptotic process /  chromatin binding / positive regulation of DNA-templated transcription / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / zinc ion binding / chromatin binding / positive regulation of DNA-templated transcription / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / zinc ion binding /  核質 / identical protein binding / 核質 / identical protein binding /  細胞核 / 細胞核 /  細胞質基質類似検索 - 分子機能 細胞質基質類似検索 - 分子機能  KMT2A, ePHD domain / KMT2A, ePHD domain /  KMT2A, PHD domain 1 / KMT2A, PHD domain 1 /  KMT2A, PHD domain 2 / KMT2A, PHD domain 2 /  KMT2A, PHD domain 3 / KMT2A, PHD domain 3 /  Methyltransferase, trithorax / Methyltransferase, trithorax /  : / FY-rich, N-terminal / F/Y-rich N-terminus / PHD-like zinc-binding domain / FYR domain FYRN motif profile. ... : / FY-rich, N-terminal / F/Y-rich N-terminus / PHD-like zinc-binding domain / FYR domain FYRN motif profile. ... KMT2A, ePHD domain / KMT2A, ePHD domain /  KMT2A, PHD domain 1 / KMT2A, PHD domain 1 /  KMT2A, PHD domain 2 / KMT2A, PHD domain 2 /  KMT2A, PHD domain 3 / KMT2A, PHD domain 3 /  Methyltransferase, trithorax / Methyltransferase, trithorax /  : / FY-rich, N-terminal / F/Y-rich N-terminus / PHD-like zinc-binding domain / FYR domain FYRN motif profile. / "FY-rich" domain, N-terminal region / FY-rich, C-terminal / F/Y rich C-terminus / FYR domain FYRC motif profile. / "FY-rich" domain, C-terminal region / CXXC zinc finger domain / : / FY-rich, N-terminal / F/Y-rich N-terminus / PHD-like zinc-binding domain / FYR domain FYRN motif profile. / "FY-rich" domain, N-terminal region / FY-rich, C-terminal / F/Y rich C-terminus / FYR domain FYRC motif profile. / "FY-rich" domain, C-terminal region / CXXC zinc finger domain /  Zinc finger, CXXC-type / Zinc finger CXXC-type profile. / Cysteine-rich motif following a subset of SET domains / Post-SET domain / Post-SET domain profile. / SET (Su(var)3-9, Enhancer-of-zeste, Trithorax) domain / SET domain superfamily / Zinc finger, CXXC-type / Zinc finger CXXC-type profile. / Cysteine-rich motif following a subset of SET domains / Post-SET domain / Post-SET domain profile. / SET (Su(var)3-9, Enhancer-of-zeste, Trithorax) domain / SET domain superfamily /  SET domain / SET domain profile. / SET domain / SET domain profile. /  SET domain / Extended PHD (ePHD) domain / Extended PHD (ePHD) domain profile. / PHD-finger / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / SET domain / Extended PHD (ePHD) domain / Extended PHD (ePHD) domain profile. / PHD-finger / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. /  Zinc finger, PHD-finger / Zinc finger, PHD-finger /  Zinc finger, PHD-type / PHD zinc finger / Zinc finger, PHD-type / PHD zinc finger /  Zinc finger, FYVE/PHD-type / Bromodomain profile. / Zinc finger, FYVE/PHD-type / Bromodomain profile. /  bromo domain / bromo domain /  ブロモドメイン / Bromodomain-like superfamily / ブロモドメイン / Bromodomain-like superfamily /  Zinc finger, RING/FYVE/PHD-type類似検索 - ドメイン・相同性 Zinc finger, RING/FYVE/PHD-type類似検索 - ドメイン・相同性 デオキシリボ核酸 / DNA (> 10) / Histone-lysine N-methyltransferase 2A類似検索 - 構成要素 デオキシリボ核酸 / DNA (> 10) / Histone-lysine N-methyltransferase 2A類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / 溶液NMR /  SIMULATED ANNEALING, DISTANCE GEOMETRY SIMULATED ANNEALING, DISTANCE GEOMETRY |
|---|
| Model details | lowest energy, model 1 |
|---|
 データ登録者 データ登録者 | Cierpicki, T. / Riesbeck, J.E. / Grembecka, J.E. / Lukasik, S.M. / Popovic, R. / Omonkowska, M. / Shultis, D.S. / Zeleznik-Le, N.J. / Bushweller, J.H. |
|---|
 引用 引用 |  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2010 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2010
タイトル: Structure of the MLL CXXC domain-DNA complex and its functional role in MLL-AF9 leukemia.
著者: Cierpicki, T. / Risner, L.E. / Grembecka, J. / Lukasik, S.M. / Popovic, R. / Omonkowska, M. / Shultis, D.D. / Zeleznik-Le, N.J. / Bushweller, J.H. |
|---|
| 履歴 | | 登録 | 2009年6月18日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2009年12月8日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2022年3月16日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード PROTEIN-DNA COMPLEX (デオキシリボ核酸) / CXXC DOMAIN / MLL / CPG DNA /
PROTEIN-DNA COMPLEX (デオキシリボ核酸) / CXXC DOMAIN / MLL / CPG DNA /  CHROMOSOMAL REARRANGEMENT / DNA-BINDING / METAL-BINDING /
CHROMOSOMAL REARRANGEMENT / DNA-BINDING / METAL-BINDING /  NUCLEUS (細胞核) /
NUCLEUS (細胞核) /  ZINC-FINGER (ジンクフィンガー) / DNA BINDING PROTEIN-DNA COMPLEX /
ZINC-FINGER (ジンクフィンガー) / DNA BINDING PROTEIN-DNA COMPLEX /  Alternative splicing (選択的スプライシング) /
Alternative splicing (選択的スプライシング) /  Apoptosis (アポトーシス) /
Apoptosis (アポトーシス) /  Bromodomain (ブロモドメイン) / Chromatin regulator /
Bromodomain (ブロモドメイン) / Chromatin regulator /  Isopeptide bond /
Isopeptide bond /  Methyltransferase (メチルトランスフェラーゼ) /
Methyltransferase (メチルトランスフェラーゼ) /  Phosphoprotein / Polymorphism /
Phosphoprotein / Polymorphism /  Proto-oncogene (がん遺伝子) /
Proto-oncogene (がん遺伝子) /  S-adenosyl-L-methionine (S-アデノシルメチオニン) /
S-adenosyl-L-methionine (S-アデノシルメチオニン) /  Transcription (転写 (生物学)) /
Transcription (転写 (生物学)) /  Transcription regulation /
Transcription regulation /  Transferase (転移酵素) / Ubl conjugation /
Transferase (転移酵素) / Ubl conjugation /  Zinc (亜鉛)
Zinc (亜鉛) 機能・相同性情報
機能・相同性情報 histone methyltransferase complex /
histone methyltransferase complex /  : / Formation of WDR5-containing histone-modifying complexes / minor groove of adenine-thymine-rich DNA binding /
: / Formation of WDR5-containing histone-modifying complexes / minor groove of adenine-thymine-rich DNA binding /  脱分極 /
脱分極 /  MLL1 complex / negative regulation of fibroblast proliferation / homeostasis of number of cells within a tissue / spleen development / transcription initiation-coupled chromatin remodeling / cellular response to transforming growth factor beta stimulus /
MLL1 complex / negative regulation of fibroblast proliferation / homeostasis of number of cells within a tissue / spleen development / transcription initiation-coupled chromatin remodeling / cellular response to transforming growth factor beta stimulus /  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / post-embryonic development / circadian regulation of gene expression / protein modification process / lysine-acetylated histone binding / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function /
転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / post-embryonic development / circadian regulation of gene expression / protein modification process / lysine-acetylated histone binding / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function /  visual learning / PKMTs methylate histone lysines / Transcriptional regulation of granulopoiesis / RUNX1 regulates transcription of genes involved in differentiation of HSCs / fibroblast proliferation / protein-containing complex assembly / apoptotic process /
visual learning / PKMTs methylate histone lysines / Transcriptional regulation of granulopoiesis / RUNX1 regulates transcription of genes involved in differentiation of HSCs / fibroblast proliferation / protein-containing complex assembly / apoptotic process /  chromatin binding / positive regulation of DNA-templated transcription / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / zinc ion binding /
chromatin binding / positive regulation of DNA-templated transcription / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / zinc ion binding /  核質 / identical protein binding /
核質 / identical protein binding /  細胞核 /
細胞核 /  細胞質基質
細胞質基質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR /
溶液NMR /  SIMULATED ANNEALING, DISTANCE GEOMETRY
SIMULATED ANNEALING, DISTANCE GEOMETRY  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2010
ジャーナル: Nat.Struct.Mol.Biol. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2kkf.cif.gz
2kkf.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2kkf.ent.gz
pdb2kkf.ent.gz PDB形式
PDB形式 2kkf.json.gz
2kkf.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/kk/2kkf
https://data.pdbj.org/pub/pdb/validation_reports/kk/2kkf ftp://data.pdbj.org/pub/pdb/validation_reports/kk/2kkf
ftp://data.pdbj.org/pub/pdb/validation_reports/kk/2kkf リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: ALL1, CXXC7, HRX, HTRX, KMT2A, MLL, TRX1 / プラスミド: pGEX4-T2 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ALL1, CXXC7, HRX, HTRX, KMT2A, MLL, TRX1 / プラスミド: pGEX4-T2 / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) ヒストンメチルトランスフェラーゼ
ヒストンメチルトランスフェラーゼ 溶液NMR
溶液NMR 試料調製
試料調製 : INOVA / 磁場強度: 600 MHz
: INOVA / 磁場強度: 600 MHz 解析
解析 SIMULATED ANNEALING, DISTANCE GEOMETRY / ソフトェア番号: 1
SIMULATED ANNEALING, DISTANCE GEOMETRY / ソフトェア番号: 1  ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj








































