+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5bqb | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
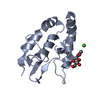
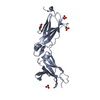
| タイトル | Crystal structure of Norrin, a Wnt signalling activator, Crystal Form III | ||||||||||||
 要素 要素 | Norrin | ||||||||||||
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  Wnt signalling pathway (Wntシグナル経路) / Wnt signalling pathway (Wntシグナル経路) /  Norrie disease protein / cystine-knot like growth factor / ligand for Frizzled 4 receptor Norrie disease protein / cystine-knot like growth factor / ligand for Frizzled 4 receptor | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報retina blood vessel maintenance / cone retinal bipolar cell differentiation / Norrin signaling pathway / extracellular matrix-cell signaling / re-entry into mitotic cell cycle / retinal blood vessel morphogenesis / retinal rod cell differentiation / ubiquitin-dependent endocytosis / retina layer formation / retinal pigment epithelium development ...retina blood vessel maintenance / cone retinal bipolar cell differentiation / Norrin signaling pathway / extracellular matrix-cell signaling / re-entry into mitotic cell cycle / retinal blood vessel morphogenesis / retinal rod cell differentiation / ubiquitin-dependent endocytosis / retina layer formation / retinal pigment epithelium development / establishment of blood-retinal barrier / microglial cell proliferation / glycine metabolic process / endothelial cell differentiation / dendritic spine development / establishment of blood-brain barrier / protein targeting to lysosome / vacuole organization / microglia differentiation /  frizzled binding / optic nerve development / retinal ganglion cell axon guidance / lens development in camera-type eye / exploration behavior / frizzled binding / optic nerve development / retinal ganglion cell axon guidance / lens development in camera-type eye / exploration behavior /  活動電位 / smoothened signaling pathway / 活動電位 / smoothened signaling pathway /  decidualization / canonical Wnt signaling pathway / response to axon injury / blood vessel remodeling / glutathione metabolic process / decidualization / canonical Wnt signaling pathway / response to axon injury / blood vessel remodeling / glutathione metabolic process /  クエン酸回路 / クエン酸回路 /  視覚 / transforming growth factor beta receptor signaling pathway / phosphatidylinositol 3-kinase/protein kinase B signal transduction / 視覚 / transforming growth factor beta receptor signaling pathway / phosphatidylinositol 3-kinase/protein kinase B signal transduction /  cytokine activity / mitotic cell cycle / cytokine activity / mitotic cell cycle /  nervous system development / cellular response to hypoxia / nervous system development / cellular response to hypoxia /  血管新生 / neuron apoptotic process / collagen-containing extracellular matrix / transcription by RNA polymerase II / protein ubiquitination / 血管新生 / neuron apoptotic process / collagen-containing extracellular matrix / transcription by RNA polymerase II / protein ubiquitination /  炎症 / positive regulation of DNA-templated transcription / 炎症 / positive regulation of DNA-templated transcription /  細胞膜 / protein homodimerization activity / 細胞膜 / protein homodimerization activity /  extracellular space extracellular space類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.3 Å 分子置換 / 解像度: 2.3 Å | ||||||||||||
 データ登録者 データ登録者 | Chang, T.-H. / Hsieh, F.-L. / Harlos, K. / Jones, E.Y. | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2015 ジャーナル: Elife / 年: 2015タイトル: Structure and functional properties of Norrin mimic Wnt for signalling with Frizzled4, Lrp5/6, and proteoglycan. 著者: Chang, T.H. / Hsieh, F.L. / Zebisch, M. / Harlos, K. / Elegheert, J. / Jones, E.Y. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5bqb.cif.gz 5bqb.cif.gz | 98.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5bqb.ent.gz pdb5bqb.ent.gz | 76.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5bqb.json.gz 5bqb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bq/5bqb https://data.pdbj.org/pub/pdb/validation_reports/bq/5bqb ftp://data.pdbj.org/pub/pdb/validation_reports/bq/5bqb ftp://data.pdbj.org/pub/pdb/validation_reports/bq/5bqb | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 |  / Norrie disease protein / X-linked exudative vitreoretinopathy 2 protein / Norrie disease protein / X-linked exudative vitreoretinopathy 2 protein分子量: 13614.706 Da / 分子数: 4 / 断片: residues 25-133 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: NDP, EVR2 / プラスミド: pHLIgK-STR-8H-SUMO-1D4 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: NDP, EVR2 / プラスミド: pHLIgK-STR-8H-SUMO-1D4 / 発現宿主:   Homo sapiens (ヒト) / 参照: UniProt: Q00604 Homo sapiens (ヒト) / 参照: UniProt: Q00604#2: 化合物 | ChemComp-CIT /  クエン酸 クエン酸#3: 化合物 |  塩化物 塩化物#4: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.68 Å3/Da / 溶媒含有率: 54.14 % |
|---|---|
結晶化 | 温度: 294 K / 手法: 蒸気拡散法, シッティングドロップ法 / 詳細: 0.1M citrate, pH 5.0, 30% PEG6000 |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I03 / 波長: 0.9796 Å / ビームライン: I03 / 波長: 0.9796 Å | |||||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2014年2月15日 | |||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.9796 Å / 相対比: 1 : 0.9796 Å / 相対比: 1 | |||||||||||||||||||||||||||
| 反射 | 解像度: 2.3→44.19 Å / Num. obs: 26073 / % possible obs: 99.2 % / 冗長度: 5.8 % / Biso Wilson estimate: 51.94 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.063 / Rpim(I) all: 0.028 / Net I/σ(I): 16.7 / Num. measured all: 151301 | |||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / Rejects: 0
|
-位相決定
位相決定 | 手法:  分子置換 分子置換 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR | Model details: Phaser MODE: MR_AUTO
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 5BPU 解像度: 2.3→44.19 Å / FOM work R set: 0.6505 / SU ML: 0.34 / 交差検証法: THROUGHOUT / σ(F): 1.36 / 位相誤差: 29.23 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 283.09 Å2 / Biso mean: 91.3 Å2 / Biso min: 24.76 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.3→44.19 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj





