-検索条件
-検索結果
検索 (著者・登録者: gros & p)の結果294件中、1から50件目までを表示しています

EMDB-18170: 
YPEL5-bound WDR26-CTLH E3 ligase - assembly I

EMDB-18171: 
YPEL5-bound WDR26-CTLH E3 ligase - assembly II

EMDB-18172: 
NMNAT1 core-bound RANBP9-TWA1-WDR26 module of WDR26-CTLH E3 ligase

EMDB-18173: 
NMNAT1 loop-bound RANBP9-TWA1-WDR26 module of WDR26-CTLH E3 ligase

EMDB-18174: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly I - class 1

EMDB-18175: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly I - class 2

EMDB-18176: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 1

EMDB-18177: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 2

EMDB-18178: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 3

EMDB-18316: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to YPEL5

EMDB-18345: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to NMNAT1 substrate
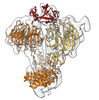
EMDB-19039: 
Map of YPEL5-bound WDR26 dimer obtained by focused refinement of the WDR26-CTLH subcomplex

EMDB-41816: 
Cryo-EM structure of the RAF1-HSP90-CDC37 complex in the closed state

EMDB-41817: 
Cryo-EM structure of the HSP90 dimer (NTD-MD) in the semi-open state

EMDB-41818: 
Cryo-EM structure of the cross-linked HSP90 dimer (NTD-MD) in the semi-open state

PDB-8u1l: 
Cryo-EM structure of the RAF1-HSP90-CDC37 complex in the closed state

PDB-8u1m: 
Cryo-EM structure of the HSP90 dimer (NTD-MD) in the semi-open state

PDB-8u1n: 
Cryo-EM structure of the cross-linked HSP90 dimer (NTD-MD) in the semi-open state

EMDB-18941: 
SARS-CoV-2 S (Spike) protein (BA.1) in complex with VHH Ma16B06 (sub-volume of two adjacent RBD-VHH modules)
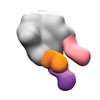
EMDB-40242: 
BG505 MD39 SOSIP in complex with Rh.NJ85 wk12 gp120GH, N611/FP, and base epitope polyclonal antibodies

EMDB-40243: 
BG505 MD39 SOSIP in complex with Rh.NJ86 wk12 V1V3, C3V5, N611/FP, and base epitope polyclonal antibodies

EMDB-40244: 
BG505 MD39 SOSIP in complex with Rh.NJ76 wk12 C3V5, N611/FP, and base epitope polyclonal antibodies
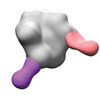
EMDB-40252: 
BG505 MD39 SOSIP in complex with Rh.NK04 wk12 gp120 glycan hole and base epitope polyclonal antibodies

EMDB-40254: 
BG505 MD39 SOSIP in complex with Rh.NJ75 wk12 gp120 glycan hole and base epitope polyclonal antibodies

EMDB-40255: 
BG505 MD39 SOSIP in complex with Rh.NJ87 wk12 C3V5 and base epitope polyclonal antibodies

EMDB-40256: 
BG505 MD39 SOSIP in complex with Rh.NJ84 wk12 V1V3, gp120 glycan hole and base epitope polyclonal antibodies

EMDB-40257: 
BG505 MD39 SOSIP in complex with Rh.NJ77 wk12 V1V3, C3V5, N611/FP and base epitope polyclonal

EMDB-29452: 
Structure Of Respiratory Syncytial Virus Polymerase with Novel Non-Nucleoside Inhibitor

PDB-8fu3: 
Structure Of Respiratory Syncytial Virus Polymerase with Novel Non-Nucleoside Inhibitor

EMDB-16730: 
Cryo-EM structure of complement C5 in complex with nanobodies UNbC5-1 and UNbC5-2

PDB-8cml: 
Cryo-EM structure of complement C5 in complex with nanobodies UNbC5-1 and UNbC5-2

EMDB-17369: 
A CHIMERA construct containing human SARM1 ARM and SAM domains and C. elegans TIR domain.

EMDB-17370: 
C. elegans TIR-1 protein.

PDB-8p2l: 
A CHIMERA construct containing human SARM1 ARM and SAM domains and C. elegans TIR domain.

PDB-8p2m: 
C. elegans TIR-1 protein.

EMDB-27973: 
Erwinia amylovora 70S Ribosome

EMDB-27993: 
Erwinia amlyavora 50S ribosome

EMDB-28003: 
Erwinia phage vB_EamM_RAY (RAY) Capsid Vertex

EMDB-28004: 
Erwinia phage vB_EamM_RAY (RAY) Capsid Collar

EMDB-28005: 
Erwinia phage vB_EamM_RAY (RAY) Tail Sheath

EMDB-28006: 
Erwinia phage vB_EamM_RAY (RAY) Baseplate

EMDB-28007: 
Erwinia phage vB_EamM_RAY (RAY) Chimallin

EMDB-28008: 
Erwinia phage vB_EamM_RAY (RAY) Putative PhuZ Filament

EMDB-28009: 
Pseudomonas chlororaphis phage 201phi2-1 PhuZ Filament

EMDB-28010: 
Pseudomonas phage phiKZ PhuZ Filament

EMDB-16480: 
Structure of the SARS-CoV-2 spike glycoprotein in complex with the 10D12 heavy-chain-only antibody (3 RBDs up)

EMDB-16481: 
Structure of the SARS-CoV-2 spike glycoprotein in complex with the 10D12 heavy-chain-only antibody (2 RBDs up)

EMDB-16490: 
Structure of the SARS-CoV-2 spike glycoprotein in complex with the 10D12 heavy-chain-only antibody (local refinement)

PDB-8c8p: 
Structure of the SARS-CoV-2 spike glycoprotein in complex with the 10D12 heavy-chain-only antibody (local refinement)

EMDB-13951: 
Inhibitor-induced hSARM1 duplex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
