7. メニュー操作
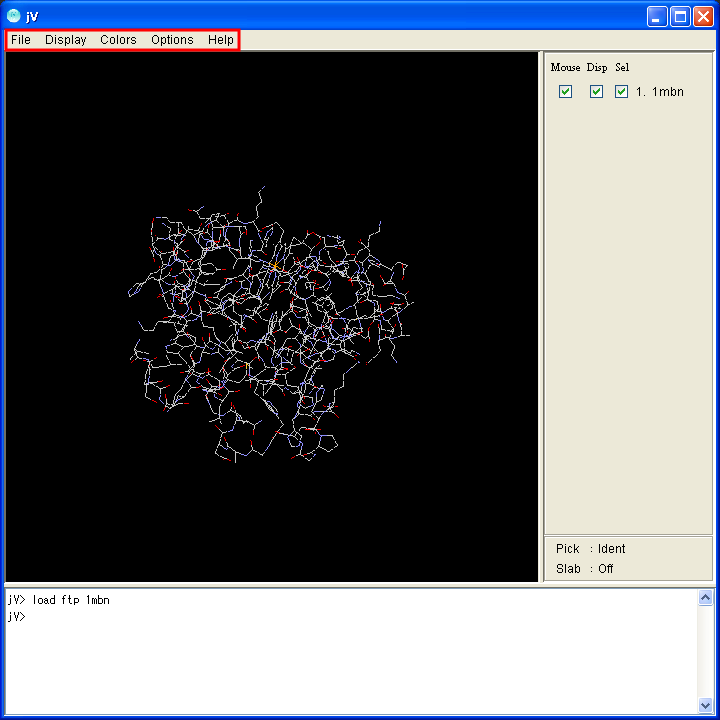
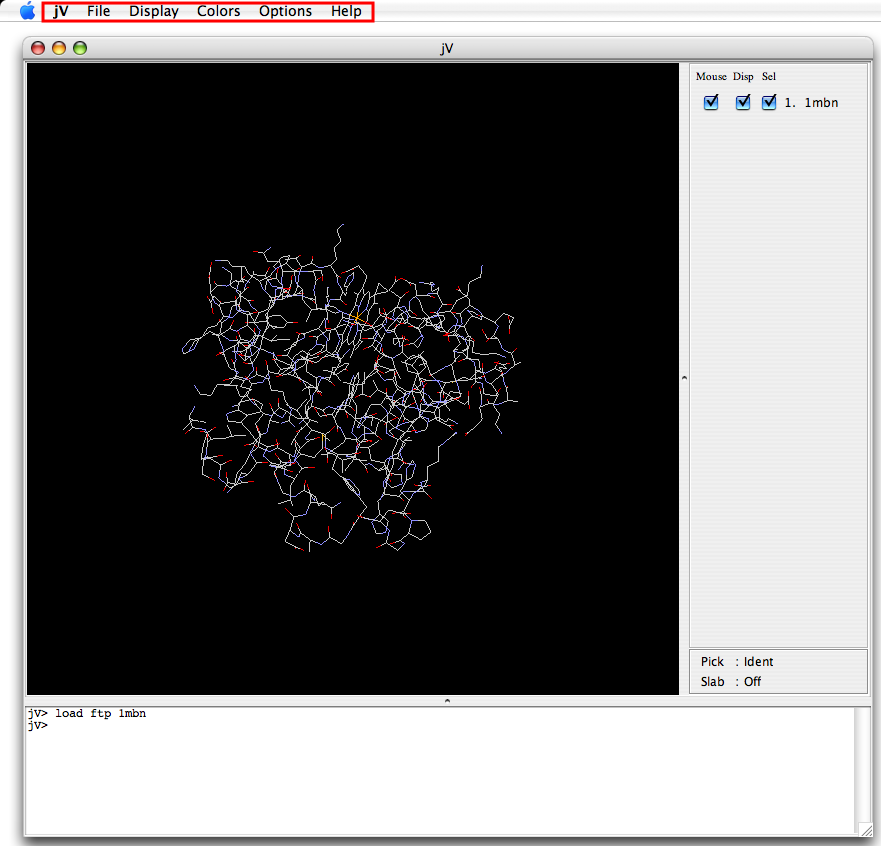
このページでは、スタンドアロン版のメニューバーにある各項目と、アップレット版の右クリックメニューの各項目について説明します。スタンドアロン(JARバイナリ)版の場合はアプリケーションのメニューを、アプレット版の場合は画面を構造表示エリアを右クリック(Macの場合はCommand + クリック)すると表示されるポップアップメニューを利用します。
 |
 |
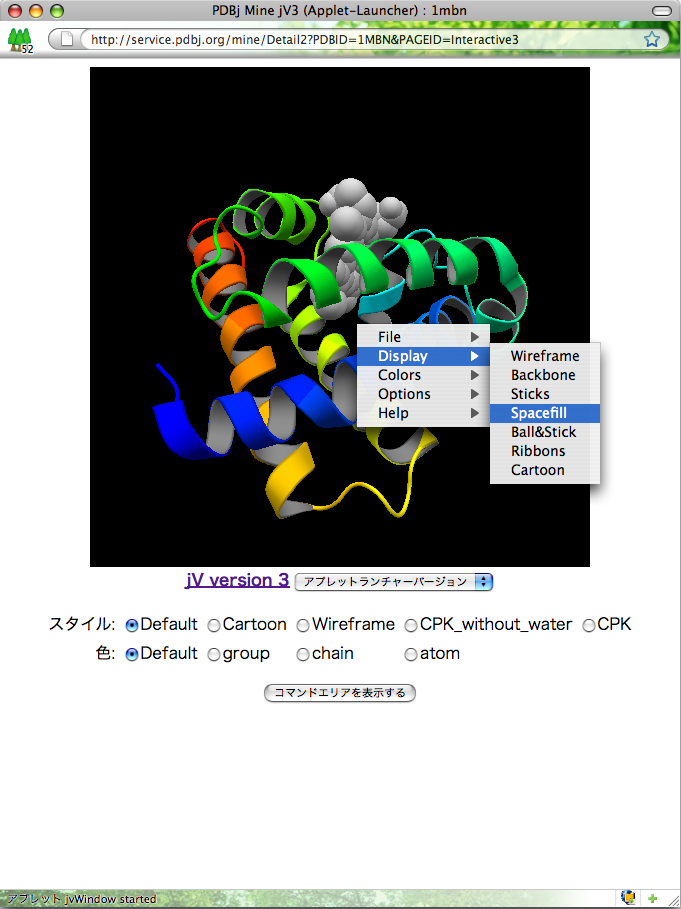 |
| スタンドアロン版(Windows) | スタンドアロン版(Mac) | アプレット版 |
7.1. ファイル(File)
| メニュー名 | 概要 |
|---|---|
| Open-Local | 7.1.1. ローカルファイルを開く |
| Open-Remote | 7.1.2. リモートファイルを開く |
| Information | 7.1.3. 情報表示 |
| Close | 7.1.4. 閉じる |
| Save | 7.1.5. 保存 |
| Exit | 7.1.6. 終了 |
| メニュー名 | 概要 |
|---|---|
| Select | 分子ファイル選択・選択解除→6. マウス操作のSelectのON/OFFに相当 |
| Display | 分子ファイル表示ON/OFF→6. マウス操作のDisplayのON/OFFに相当 |
| Information | 7.1.3. 情報表示 |
| Save | 7.1.5. 保存 |
7.1.1. ローカルファイルを開く([File][Open-Local])
![[File][Open-Local]](./images/jV_menu01.png)
jVを実行しているシステムからローカルパスで参照できるファイルを読み込みます。 選択できるファイル種は以下の通りです。 各サブサブメニュー項目をクリックすると、ファイルを選択するダイアログ画面が表示されます。
| サブメニューラベル | 備考 |
|---|---|
| mmCIF | PDBx/mmCIF形式のファイルを読み込みます。書式など詳細はPDBx/mmCIF 辞書関連情報をご覧下さい。 |
| PDBML | PDBML形式のファイルを読み込みます。 |
| PDB | PDB形式のファイルを読み込みます。 |
| Polygon | jV用ポリゴンXMLファイルを読み込みます。書式など詳細はポリゴンファイルについてをご覧下さい。 |
| Polygon CGO | eF-siteサービスで配布しているポリゴンCGO(PyMOL-specific Compiled Graphics Objects)ファイルを読み込みます。 |
| Script | jV用スクリプトファイル(一連の操作をjVのコマンドで記録したテキストファイル)を読み込みます。 |
| Animation | アニメーションファイル(複数のモデルを含むPDB形式のファイル)を読み込みます。 |
「PDBML」では、PDBML形式(XML形式のPDBデータファイル)のファイルを読み込みます。
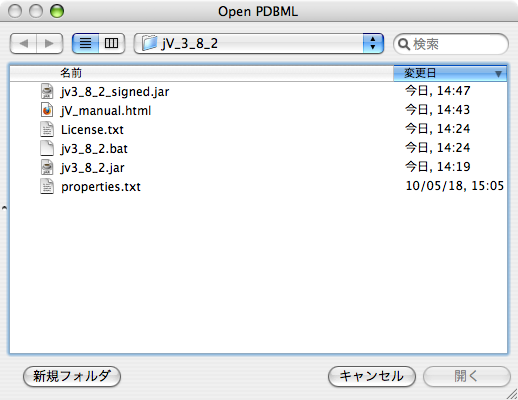
「PDB」ではPDB形式(固定幅テキスト形式のPDBフォーマット)のファイルを読み込みます。
「Polygon」ではポリゴンデータファイルを読み込みます。
「Script」ではjVの操作群を記録したスクリプトファイルを読み込みます。
「Animation」ではアニメーションファイルを読み込みます。
7.1.2. リモートファイルを開く(Open-Remote)
![[File][Open-Remote]](./images/jV_menu02.png)
このメニューでは、インターネット上にあるファイルの読み込みを指示します。 読み込むファイルの種類はローカルファイルの場合と同じです。 Open-Remoteメニューをポイントすると表示されるサブメニューから表示したいファイルの種類を選んでクリックして下さい。 いずれのサブメニューもリモートファイルを選択するダイアログ画面が表示され、そちらでURLを指定します。 なお、PDBやeF-siteからファイルを取得する場合、IDを指定するだけで読み込むことのできる便利なメニュー「PDB ID」「eF-site ID」もあります。
「PDBML」では、URLを指定してリモートサイトにあるPDBMLファイルを開きます。
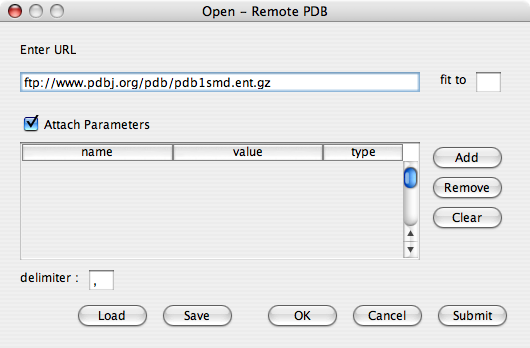
「Enter URL」の下にある入力欄に読み込みたいファイルのURLを入力します。 「fit to」には数値を指定します。 指定した値に対応したファイルID(読み込まれた順に1から割り振られる連番)が存在した場合、そのファイルに重なる位置に読み込まれます。 指定した値に対応したファイルIDが存在しなかった場合、または値を指定しなかった場合は、表示エリアの中央にファイルが読み込まれます。
読み込む際、パラメータを指定したい場合、「Attach Parameters」のチェックボックスをONにして下さい。 GETパラメータを含むURLを指定する場合、パラメータ部分はこちらで指定して下さい。 「Add」ボタンをクリックすると、ダイアログの中央にある表に新しいパラメータ行が1行追加されます。必要な数だけ追加することができます。 追加したパラメータを個別に消去するには、消去したい行をクリックして選択し、「Remove」ボタンをクリックして下さい。 全てのパラメータを一括消去するには「Clear」ボタンをクリックして下さい。パラメータの「型(type)」に指定できる値は以下の通りです。
| 型 (type) |
値 (values) |
マウス選択 (mouse pick) |
|---|---|---|
| text | 任意の値 | 不可 |
| atom ID | 原子ID | 可 |
| atom | 鎖ID、残基ID、原子IDの組み合わせ | 可 |
| residue | 鎖ID、残基IDの組み合わせ | 可 |
| orig_coords | 分子ファイルに書かれた原子座標 | 可 |
| curr_coords | 現在表示されている分子の原子座標 | 可 |
マウス選択が可となっているtext型以外については、既に表示されているファイルがある場合に立体画像表示エリアをクリックして情報を入力することができます。
「delimiter」には、原子座標のx,y,zの各値を区切るのに使われる記号を指定するのに使う文字列を指定しますが、初期値の「,」(コンマ)がjVでは使用されるため変更する必要はありません。
全ての指定を終えたら「OK」ボタンをクリックして下さい。ダイアログは閉じて、データがダウンロードされ、画像が表示されます。 連続して複数のファイルを読み込みたい場合は「Submit」ボタンを使って下さい。「OK」ボタンと同様にデータがダウンロードされて画像が表示されますが、ダイアログは閉じず続けて次の指定を行うことができます。 ダウンロードを中止する際は、「Cancel」ボタンをクリックして下さい。ダウンロードは行われずダイアログは閉じます。
「save」ボタンをクリックすると、ファイル選択ダイアログが表示されます。ファイル名と保存場所を指定して現在の設定を保存することができます。「load」ボタンをクリックすると、同様にファイル選択ダイアログが表示され、保存した設定を読み込むことができます。保存ファイルは下記のようなXML形式で記述されています。
<?xml version="1.0"?> <remote_file url="http://example.com"> <params attach="true" coordinates_delimiter=","> <param name="param1" value="value1" type="text"/> </params> </remote_file>
「PDB」「Polygon」「Script」「Animation」の場合も「PDBML」と同様です。
「PDB ID」では、次のようなダイアログが表示されます。
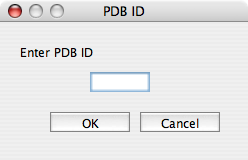
「Enter PDB ID」欄に情報を表示したい分子のPDB IDを入力して「OK」ボタンをクリックします。
この方法では、ダウンロードサイトを指定する必要はありません。
load ftp [PDB ID] コマンド、[File][Open-Remote][PDB]でURLを指定して開くのと結果は同じです。
「eF-site ID」では、次のようなダイアログが表示されます。
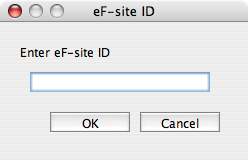
「PDB ID」の場合と同様にして、「Enter eF-site ID」欄に情報を表示したい分子のeF-site IDを入力して「OK」ボタンをクリックします。 この方法では、ダウンロードサイトを指定する必要はありません。 [File][Open-Remote][Polygon]でURLを指定して開くのと結果は同じです。
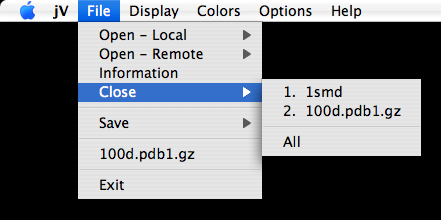
7.1.4. 閉じる(Close)
zapコマンドと同じ動作をします。
closeメニューをポイントすると、開かれているファイルのリストがサブメニューに表示されます。
閉じたいファイルをクリックすれば、そのファイルのみが閉じられます。
zapコマンドを、ファイルIDを指定して実行するのと同じです。
例えば、1番目のサブメニューをクリックするのと、引数をつけてzap 1を実行するのは同じです。
サブメニューの一番下に表示される「All」をクリックすると開かれている全てのファイルが閉じられます。引数を指定せずzapコマンドを実行するのと同じです。
なおこの「All」サブメニューは2つ以上のファイルを開いている時にしか表示されません。
7.1.5. 保存(Save)
「PDB」ではPDB format形式で、原子位置情報などを抽出したものがテキスト形式で保存されます。具体的に抽出される項目名は、HEADER, COMPND, EXPDTA, MODEL, ATOM, TER, HETATM, ENDMDL, ENDのようです。このメニューはスタンドアロン版のみ利用できます。
「Script」ではファイルの読み込み、分子の移動、表示方法の変更など一連の操作過程を記録した情報がスクリプトファイルとして保存されます。このメニューはスタンドアロン版のみ利用できます。
「PNG」「JPEG」ではメイン画面に表示されている内容を画像ファイルとして保存します。このメニューはスタンドアロン版およびアプレット版どちらでも利用できます(アプレット版はjV 3.8より対応)。
7.1.6. 終了(Exit)
jVを終了します。exit コマンドを実行するのと同じです。
7.2. 表示(Display)
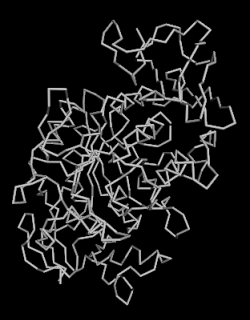
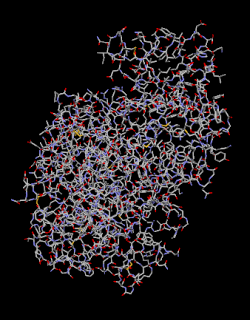
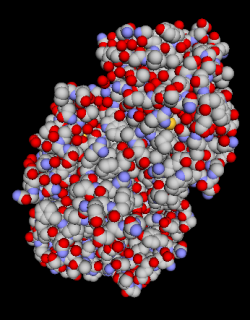
選択されている原子の表示方法を設定します。「Wireframe」(ワイヤーフレーム)「Backbone」(主鎖)「Sticks」(棒)「Spacefill」(空間充塡)「Ball&Stick」(球棒)「Ribbons」(リボン)「Cartoon」(太いリボン)の7種があります。
選択できるのはいずれか1つで、任意のメニューを選択すると、その表示方法に切り替わります。
原子の選択に関しては select コマンド参照のこと。
このメニューに関しては、スタンドアロン版メニューとアップレット版の右クリックメニューとで違いはありません。
なおこのメニューによる操作とコマンドによる操作(wireframe、backbone、spacefill (cpk)、cartoon)は似ていますが、メニューによる操作では選択範囲の表示が指定した表現に「変更される」のに対し、コマンドによる操作では既存の表示を残したまま指定した表現が「追記される」点が異なります。

|
|
|
| ワイヤーフレーム(Wireframe) | 主鎖(Backbone) | 棒(Sticks) |
|

|

|
| 空間充塡(Spacefill) | 球棒(Ball&Stick) | リボン(Ribbons) |
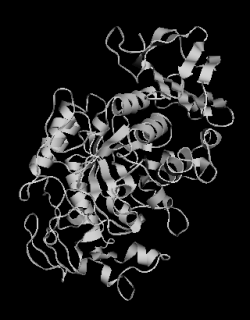
|
||
| 太いリボン(Cartoon) |
メニューによる表示操作では、既存表示を消去してから指定方法により描画されます。 コマンドによる動作はこれとは異なり、既存表示は残したまま指定方法により上書き描画されます。 従って、例えばワイヤーフレーム表示されている画像を太いリボン(Cartoon)表現に変更する際、メニューを使うと、全てCartoon表現になりますが、コマンドを使うと側鎖部分はワイヤーフレーム表現が残ったまま主鎖部分だけがCartoon表現で上書きされます。 必要に応じて使い分けて下さい。

ワイヤーフレーム表現の画像をメニューでCartoon表現に変更したもの。 |

ワイヤーフレーム表現の画像を |
7.3. 色(Colors)
選択されている原子を以下の配色規則に従って色分けします。
選択できるのはいずれか1つで、任意のメニューを選択すると、その表示方法に切り替わります。
原子の選択に関しては select コマンド参照。
配色の詳細については色表現の配色参照。
このメニューに関しては、スタンドアロン版メニューとアップレット版の右クリックメニューとで違いはありません。
| 配色指定 | 内容 |
|---|---|
| モノクロ(Monochrome) | 灰色単色で表示。 |
| CPK | 既定の原子色に従い原子種別に色分けして表示 (「色表現」のcpk参照)。 |
| 均整配色(Shapely) | アミノ酸や核酸の性質に従って色分けして表示 (「色表現」の均整配色(shapely)参照)。 |
| グループ(Group) | 各鎖をN末端側(核酸の場合は5´末端側)に向かうほど赤く、 C末端側(核酸の場合は3´末端側)に向かうほど青くなるよう段階的に変色させて表示。 |
| 鎖(Chain) | 鎖ごとに色分けして表示。 |
| 温度(Temperature) | 各原子の温度因子に従い、高い値ほど赤く低い値ほど青くなるよう段階的に変色させて表示。 |
| 構造(Structure) |
二次構造によって色分けして表示 (「色表現」の構造参照)。 |
| 電荷(Charge) | 原子の電荷が正であるほど青く、負であるほど赤くなるよう段階的に変色させて表示。 |
| アミノ酸(Amino) | 既定のアミノ酸配色に従いアミノ酸種別に色分けして表示(「色表現」のアミノ酸参照)。 |
7.4. オプション(Options)
最初のメニューグループでは、画像の表示方法を設定します。
「Hetero Atoms」「Hydrogens」「Slab」「Stereo」「Load To Center」の5項目(アップレット右クリックメニューについては「Load To Center」を除く4項目)があり、それぞれ独立にON/OFFすることができます。
メニューをクリックする度にON/OFF(stereoについては「ON(交差視点モード)」「ON(平行視点モード)」「OFF」の3状態)が切り替わり、ONの時はメニュー項目名の左にチェックマークがつきます。
| メニュー | 備考 |
|---|---|
| ヘテロ分子(Hetero Atoms) | このメニューをONにすると、 ヘテロ原子(PDBファイルで「HETATM」グループに登録されている原子)も含め現在読み込まれている全原子を選択します。 このメニューをOFFにすると、ヘテロ原子を除く全原子が選択されます(言い換えると、PDBファイルで「ATOM」グループに登録されている原子だけが選択対象となります)。 このメニューの動作は、どの原子が選択されていたかに依存しません(常に読み込まれている全原子が対象になります)。 |
| 水素(Hydrogens) | このメニューをONにすると、 水素原子も含め現在読み込まれている全原子を選択します。 このメニューをOFFにすると、水素原子を除く全原子が選択されます。 このメニューの動作は、どの原子が選択されていたかに依存しません(常に読み込まれている全原子が対象になります)。 |
| Slab |
分子をZ軸(奥行き方向)に垂直な平面で切断するかどうかを切り替えます。
メニューをクリックする度にON/OFFが切り替わります。
切断位置を設定したい場合は slab コマンドを使用して下さい。
|
| ステレオ(Stereo) |
ステレオ形式での表示設定を行います。
メニューをクリックする度に
「ステレオ表示OFF」「ステレオ表示ON 交差視点モード」「ステレオ表示ON 平行視点モード」
の順に切り替わります。
視点設定を行いたい場合は stereo コマンド使用して下さい。
|
| 中央に表示(Load To Center) |
これをONにすると、既に開いている全画像の中央を初期中心位置として新しいファイルを読み込みます。
メニューをクリックする度にON/OFFが切り替わります。
set loadcenter コマンドと同じ動作です。
このメニューはアップレット右クリックメニューにはありません。
|
2つ目のメニューグループでは、マウスピッキング(マウスクリックによる動作)の設定を行います。
メニューには「Pick Off」「Pick Ident」「Pick Coordinate」「Pick Distance」「Pick Center」「Pick Select」の6種があります。
動作は set picking コマンドと同じ動作です。詳細はコマンドの説明を参照して下さい。このメニューグループに関しては、スタンドアロン版メニューとアップレット版右クリックメニューとで違いはありません。
3つ目のメニューグループでは、アニメーションの設定を行います。「Animation」をクリックすると、次のような設定ダイアログが開きます。このメニューグループに関しても、スタンドアロン版メニューとアップレット版右クリックメニューとで違いはありません。
![jV menu [Options]-[Animation]](images/menu_op_anim.png)
アニメーションファイルが1つも開かれていない時は上図のようにSelect,Frame,Stepが操作できない状態になっています。アニメーションファイルを開くとこれらが操作できるようになります。動作についてはアニメーション関係コマンド参照。
7.5. ヘルプ(Help)
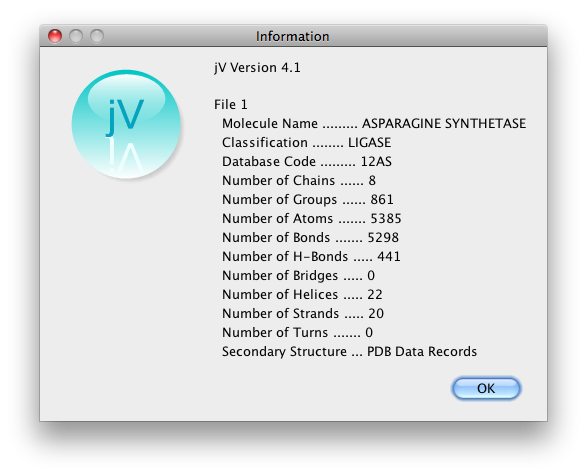
「About jV」メニューをクリックすると、次のようなダイアログが表示されます。 バージョン情報と、jVに関する概要が記されています。 内容はこのマニュアルの各ページ冒頭の記載事項と同様です。 スタンドアロン版メニューとアップレット版右クリックメニューに違いはありません。