| 登録情報 | データベース: PDB / ID: 7bqv
|
|---|
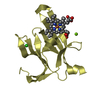
| タイトル | Cereblon in complex with SALL4 and (S)-5-hydroxythalidomide |
|---|
 要素 要素 | - Protein cereblon
- Sal-like protein 4
|
|---|
 キーワード キーワード | METAL BINDING PROTEIN /  ZINC FINGER (ジンクフィンガー) / ZINC FINGER (ジンクフィンガー) /  E3 UBIQUITIN LIGASE (ユビキチンリガーゼ) / E3 UBIQUITIN LIGASE (ユビキチンリガーゼ) /  COMPLEX / COMPLEX /  THALIDOMIDE (サリドマイド) THALIDOMIDE (サリドマイド) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
POU5F1 (OCT4), SOX2, NANOG activate genes related to proliferation / negative regulation of monoatomic ion transmembrane transport / Transcriptional regulation of pluripotent stem cells / embryonic limb morphogenesis / Cul4A-RING E3 ubiquitin ligase complex / inner cell mass cell proliferation / ventricular septum development / locomotory exploration behavior / somatic stem cell population maintenance / positive regulation of Wnt signaling pathway ...POU5F1 (OCT4), SOX2, NANOG activate genes related to proliferation / negative regulation of monoatomic ion transmembrane transport / Transcriptional regulation of pluripotent stem cells / embryonic limb morphogenesis / Cul4A-RING E3 ubiquitin ligase complex / inner cell mass cell proliferation / ventricular septum development / locomotory exploration behavior / somatic stem cell population maintenance / positive regulation of Wnt signaling pathway / negative regulation of protein-containing complex assembly /  ヘテロクロマチン / Regulation of PTEN gene transcription / neural tube closure / positive regulation of protein-containing complex assembly / proteasome-mediated ubiquitin-dependent protein catabolic process / Potential therapeutics for SARS / transmembrane transporter binding / protein ubiquitination / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / intracellular membrane-bounded organelle / regulation of transcription by RNA polymerase II / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / ヘテロクロマチン / Regulation of PTEN gene transcription / neural tube closure / positive regulation of protein-containing complex assembly / proteasome-mediated ubiquitin-dependent protein catabolic process / Potential therapeutics for SARS / transmembrane transporter binding / protein ubiquitination / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / intracellular membrane-bounded organelle / regulation of transcription by RNA polymerase II / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  核質 / 核質 /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 Yippee/Mis18/Cereblon / Yippee zinc-binding/DNA-binding /Mis18, centromere assembly /  CULT domain / CULT domain profile. / CULT domain / CULT domain profile. /  Lon protease, N-terminal domain superfamily / Lon N-terminal domain profile. / Lon protease, N-terminal domain superfamily / Lon N-terminal domain profile. /  Lon protease, N-terminal domain / ATP-dependent protease La (LON) substrate-binding domain / Found in ATP-dependent protease La (LON) / PUA-like superfamily ...Yippee/Mis18/Cereblon / Yippee zinc-binding/DNA-binding /Mis18, centromere assembly / Lon protease, N-terminal domain / ATP-dependent protease La (LON) substrate-binding domain / Found in ATP-dependent protease La (LON) / PUA-like superfamily ...Yippee/Mis18/Cereblon / Yippee zinc-binding/DNA-binding /Mis18, centromere assembly /  CULT domain / CULT domain profile. / CULT domain / CULT domain profile. /  Lon protease, N-terminal domain superfamily / Lon N-terminal domain profile. / Lon protease, N-terminal domain superfamily / Lon N-terminal domain profile. /  Lon protease, N-terminal domain / ATP-dependent protease La (LON) substrate-binding domain / Found in ATP-dependent protease La (LON) / PUA-like superfamily / Lon protease, N-terminal domain / ATP-dependent protease La (LON) substrate-binding domain / Found in ATP-dependent protease La (LON) / PUA-like superfamily /  Zinc finger, C2H2 type / Zinc finger, C2H2 type /  ジンクフィンガー / Zinc finger C2H2 type domain profile. / Zinc finger C2H2 superfamily / Zinc finger C2H2 type domain signature. / Zinc finger C2H2-type類似検索 - ドメイン・相同性 ジンクフィンガー / Zinc finger C2H2 type domain profile. / Zinc finger C2H2 superfamily / Zinc finger C2H2 type domain signature. / Zinc finger C2H2-type類似検索 - ドメイン・相同性 Chem-F4U / Protein cereblon / Sal-like protein 4類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.8 Å 分子置換 / 解像度: 1.8 Å |
|---|
 データ登録者 データ登録者 | Furihata, H. / Miyauchi, Y. / Asano, A. / Tanokura, M. / Miyakawa, T. |
|---|
| 資金援助 |  日本, 1件 日本, 1件 | 組織 | 認可番号 | 国 |
|---|
| Japan Agency for Medical Research and Development (AMED) | JP19am0101077 |  日本 日本 |
|
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020
タイトル: Structural bases of IMiD selectivity that emerges by 5-hydroxythalidomide.
著者: Furihata, H. / Yamanaka, S. / Honda, T. / Miyauchi, Y. / Asano, A. / Shibata, N. / Tanokura, M. / Sawasaki, T. / Miyakawa, T. |
|---|
| 履歴 | | 登録 | 2020年3月25日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2020年8月26日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年10月14日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.year / _citation_author.identifier_ORCID |
|---|
| 改定 1.2 | 2023年11月29日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード ZINC FINGER (ジンクフィンガー) /
ZINC FINGER (ジンクフィンガー) /  E3 UBIQUITIN LIGASE (ユビキチンリガーゼ) /
E3 UBIQUITIN LIGASE (ユビキチンリガーゼ) /  COMPLEX /
COMPLEX /  THALIDOMIDE (サリドマイド)
THALIDOMIDE (サリドマイド) 機能・相同性情報
機能・相同性情報 ヘテロクロマチン / Regulation of PTEN gene transcription / neural tube closure / positive regulation of protein-containing complex assembly / proteasome-mediated ubiquitin-dependent protein catabolic process / Potential therapeutics for SARS / transmembrane transporter binding / protein ubiquitination / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / intracellular membrane-bounded organelle / regulation of transcription by RNA polymerase II / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /
ヘテロクロマチン / Regulation of PTEN gene transcription / neural tube closure / positive regulation of protein-containing complex assembly / proteasome-mediated ubiquitin-dependent protein catabolic process / Potential therapeutics for SARS / transmembrane transporter binding / protein ubiquitination / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / intracellular membrane-bounded organelle / regulation of transcription by RNA polymerase II / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  核質 /
核質 /  生体膜 /
生体膜 /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.8 Å
分子置換 / 解像度: 1.8 Å  データ登録者
データ登録者 日本, 1件
日本, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2020
ジャーナル: Nat Commun / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7bqv.cif.gz
7bqv.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7bqv.ent.gz
pdb7bqv.ent.gz PDB形式
PDB形式 7bqv.json.gz
7bqv.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/bq/7bqv
https://data.pdbj.org/pub/pdb/validation_reports/bq/7bqv ftp://data.pdbj.org/pub/pdb/validation_reports/bq/7bqv
ftp://data.pdbj.org/pub/pdb/validation_reports/bq/7bqv リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: CRBN, AD-006 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CRBN, AD-006 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: Q96SW2
Escherichia coli (大腸菌) / 参照: UniProt: Q96SW2
 Homo sapiens (ヒト) / 遺伝子: SALL4, ZNF797 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: SALL4, ZNF797 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: Q9UJQ4
Escherichia coli (大腸菌) / 参照: UniProt: Q9UJQ4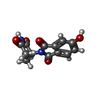






 硫酸塩
硫酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Photon Factory
Photon Factory  / ビームライン: AR-NE3A / 波長: 1 Å
/ ビームライン: AR-NE3A / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj








