+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6t0e | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The glucuronoyl esterase OtCE15A S267A variant from Opitutus terrae in complex with benzyl D-glucuronoate and D-glucuronate | |||||||||
 要素 要素 | glucuronoyl esterase OtCE15A | |||||||||
 キーワード キーワード |  HYDROLASE (加水分解酵素) / HYDROLASE (加水分解酵素) /  Esterase (エステラーゼ) / Esterase (エステラーゼ) /  Complex / Complex /  Biomass (バイオマス) Biomass (バイオマス) | |||||||||
| 機能・相同性 |  Alpha/Beta hydrolase fold / alpha-D-glucopyranuronic acid / benzyl alpha-D-glucopyranuronate / DI(HYDROXYETHYL)ETHER / Alpha/Beta hydrolase fold / alpha-D-glucopyranuronic acid / benzyl alpha-D-glucopyranuronate / DI(HYDROXYETHYL)ETHER /  TRIETHYLENE GLYCOL / TRIETHYLENE GLYCOL /  トリメチルアミン-N-オキシド / Putative acetyl xylan esterase トリメチルアミン-N-オキシド / Putative acetyl xylan esterase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Opitutus terrae (バクテリア) Opitutus terrae (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.89 Å 分子置換 / 解像度: 1.89 Å | |||||||||
 データ登録者 データ登録者 | Mazurkewich, S. / Navarro Poulsen, J.C. / Larsbrink, J. / Lo Leggio, L. | |||||||||
| 資金援助 |  スウェーデン, スウェーデン,  デンマーク, 2件 デンマーク, 2件
| |||||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2019 ジャーナル: J.Biol.Chem. / 年: 2019タイトル: Structural and biochemical studies of the glucuronoyl esteraseOtCE15A illuminate its interaction with lignocellulosic components. 著者: Mazurkewich, S. / Poulsen, J.N. / Lo Leggio, L. / Larsbrink, J. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6t0e.cif.gz 6t0e.cif.gz | 327.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6t0e.ent.gz pdb6t0e.ent.gz | 266 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6t0e.json.gz 6t0e.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/t0/6t0e https://data.pdbj.org/pub/pdb/validation_reports/t0/6t0e ftp://data.pdbj.org/pub/pdb/validation_reports/t0/6t0e ftp://data.pdbj.org/pub/pdb/validation_reports/t0/6t0e | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 46132.547 Da / 分子数: 2 / 変異: S267A / 由来タイプ: 組換発現 由来: (組換発現)   Opitutus terrae (strain DSM 11246 / JCM 15787 / PB90-1) (バクテリア) Opitutus terrae (strain DSM 11246 / JCM 15787 / PB90-1) (バクテリア)株: DSM 11246 / JCM 15787 / PB90-1 / 遺伝子: Oter_0116 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: B1ZMF4 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: B1ZMF4 |
|---|
-糖 , 2種, 3分子 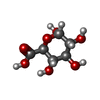
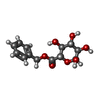

| #2: 糖 |  グルクロン酸 グルクロン酸#4: 糖 | ChemComp-M55 / | |
|---|
-非ポリマー , 9種, 472分子 





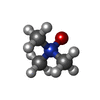










| #3: 化合物 | ChemComp-CL /  塩化物 塩化物 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| #5: 化合物 | ChemComp-PG4 /  ポリエチレングリコール ポリエチレングリコール | ||||||||||||
| #6: 化合物 | ChemComp-EDO /  エチレングリコール エチレングリコール#7: 化合物 | ChemComp-PEG /  ジエチレングリコール ジエチレングリコール#8: 化合物 | ChemComp-PGE /  ポリエチレングリコール ポリエチレングリコール#9: 化合物 | ChemComp-MG / | #10: 化合物 | ChemComp-TMO /  トリメチルアミン-N-オキシド トリメチルアミン-N-オキシド#11: 化合物 | ChemComp-DMS /  ジメチルスルホキシド ジメチルスルホキシド#12: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.14 Å3/Da / 溶媒含有率: 42.39 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: Enzyme mixed 50/50 with reservoir solution containing JCSG+ screen solution G4: 0.2 M Trimethylamine N-oxide, 0.1 M Tris pH 8.5, and 20 % w/v PEG 2000 MME |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: MASSIF-3 / 波長: 0.9677 Å / ビームライン: MASSIF-3 / 波長: 0.9677 Å |
| 検出器 | タイプ: DECTRIS EIGER X 4M / 検出器: PIXEL / 日付: 2018年12月9日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9677 Å / 相対比: 1 : 0.9677 Å / 相対比: 1 |
| 反射 | 解像度: 1.89→44.45 Å / Num. obs: 63514 / % possible obs: 98.98 % / 冗長度: 13.6 % / Biso Wilson estimate: 33.05 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.1589 / Rpim(I) all: 0.04436 / Rrim(I) all: 0.1651 / Net I/σ(I): 12.25 |
| 反射 シェル | 解像度: 1.89→1.96 Å / 冗長度: 13.4 % / Mean I/σ(I) obs: 0.84 / Num. unique obs: 5944 / CC1/2: 0.403 / Rpim(I) all: 0.6206 / % possible all: 92 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 6gs0 解像度: 1.89→44.45 Å / SU ML: 0.2534 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 22.5575
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 39.74 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.89→44.45 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj









