+ データを開く
データを開く
- 基本情報
基本情報
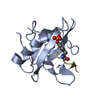
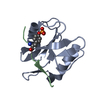
| 登録情報 | データベース: PDB / ID: 6hpj | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of human SRSF1 RRM1 bound to AACAAA RNA | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  RNA BINDING PROTEIN (RNA結合タンパク質) / RNA BINDING PROTEIN (RNA結合タンパク質) /  splicing / splicing /  SR protein (SRタンパク質) / SRSF1 / RRM / SR protein (SRタンパク質) / SRSF1 / RRM /  spinal muscular atrophy (脊髄性筋萎縮症) / SMA / spinal muscular atrophy (脊髄性筋萎縮症) / SMA /  RNA (リボ核酸) RNA (リボ核酸) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 DNA topoisomerase binding / DNA topoisomerase binding /  protein kinase B binding / alternative mRNA splicing, via spliceosome / mRNA splice site recognition / mRNA 3'-end processing / Transport of Mature mRNA derived from an Intron-Containing Transcript / RNA Polymerase II Transcription Termination / IgG binding / mRNA Splicing - Minor Pathway / oligodendrocyte differentiation ... protein kinase B binding / alternative mRNA splicing, via spliceosome / mRNA splice site recognition / mRNA 3'-end processing / Transport of Mature mRNA derived from an Intron-Containing Transcript / RNA Polymerase II Transcription Termination / IgG binding / mRNA Splicing - Minor Pathway / oligodendrocyte differentiation ... DNA topoisomerase binding / DNA topoisomerase binding /  protein kinase B binding / alternative mRNA splicing, via spliceosome / mRNA splice site recognition / mRNA 3'-end processing / Transport of Mature mRNA derived from an Intron-Containing Transcript / RNA Polymerase II Transcription Termination / IgG binding / mRNA Splicing - Minor Pathway / oligodendrocyte differentiation / protein kinase B binding / alternative mRNA splicing, via spliceosome / mRNA splice site recognition / mRNA 3'-end processing / Transport of Mature mRNA derived from an Intron-Containing Transcript / RNA Polymerase II Transcription Termination / IgG binding / mRNA Splicing - Minor Pathway / oligodendrocyte differentiation /  regulation of RNA splicing / mRNA 5'-splice site recognition / Processing of Capped Intron-Containing Pre-mRNA / mRNA transport / catalytic step 2 spliceosome / mRNA Splicing - Major Pathway / positive regulation of RNA splicing / regulation of RNA splicing / mRNA 5'-splice site recognition / Processing of Capped Intron-Containing Pre-mRNA / mRNA transport / catalytic step 2 spliceosome / mRNA Splicing - Major Pathway / positive regulation of RNA splicing /  liver regeneration / liver regeneration /  転写後修飾 / 転写後修飾 /  mRNA splicing, via spliceosome / nuclear speck / mRNA splicing, via spliceosome / nuclear speck /  mRNA binding / mRNA binding /  RNA binding / extracellular region / RNA binding / extracellular region /  核質 / 核質 /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |  Streptococcus sp. group G (バクテリア) Streptococcus sp. group G (バクテリア)  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing | ||||||
 データ登録者 データ登録者 | Allain, F.T.H. / Clery, A. | ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure of SRSF1 RRM1 bound to RNA reveals an unexpected bimodal mode of interaction and explains its involvement in SMN1 exon7 splicing. 著者: Clery, A. / Krepl, M. / Nguyen, C.K.X. / Moursy, A. / Jorjani, H. / Katsantoni, M. / Okoniewski, M. / Mittal, N. / Zavolan, M. / Sponer, J. / Allain, F.H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6hpj.cif.gz 6hpj.cif.gz | 597.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6hpj.ent.gz pdb6hpj.ent.gz | 495.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6hpj.json.gz 6hpj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hp/6hpj https://data.pdbj.org/pub/pdb/validation_reports/hp/6hpj ftp://data.pdbj.org/pub/pdb/validation_reports/hp/6hpj ftp://data.pdbj.org/pub/pdb/validation_reports/hp/6hpj | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: 抗体 | 分子量: 17956.693 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: GB1 tag followed by 6His tag and SRSF1 RRM1 from amino acid 1 to 97,GB1 tag followed by 6His tag and SRSF1 RRM1 from amino acid 1 to 97 由来: (組換発現)  Streptococcus sp. group G (バクテリア), (組換発現) Streptococcus sp. group G (バクテリア), (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: spg, SRSF1, ASF, SF2, SF2P33, SFRS1, OK/SW-cl.3 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P19909, UniProt: Q07955 Escherichia coli (大腸菌) / 参照: UniProt: P19909, UniProt: Q07955 |
|---|---|
| #2: RNA鎖 | 分子量: 1906.253 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: RNA molecule / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||
| 試料状態 | 詳細: 20mM NaHPO4 pH7, 50mM Arg, 50mM Glu, 0.05% beta-mercaptoethanol イオン強度: 120 mM / Label: sample / pH: 7 Not defined / 圧: atmospheric atm / 温度: 313 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法:  simulated annealing / ソフトェア番号: 1 simulated annealing / ソフトェア番号: 1 | |||||||||||||||
| 代表構造 | 選択基準: lowest energy | |||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




