| 登録情報 | データベース: PDB / ID: 6ed9
|
|---|
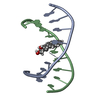
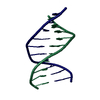
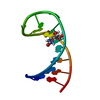
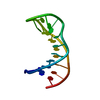
| タイトル | NMR structure for Sp1 transcription factor duplex 5'-d(TGGGCGGGA) |
|---|
 要素 要素 | - DNA (5'-D(*TP*CP*CP*CP*GP*CP*CP*CP*A)-3')
- DNA (5'-D(*TP*GP*GP*GP*CP*GP*GP*GP*A)-3')
|
|---|
 キーワード キーワード |  DNA (デオキシリボ核酸) / Sp1 Consensus Sequence DNA (デオキシリボ核酸) / Sp1 Consensus Sequence |
|---|
| 機能・相同性 |  デオキシリボ核酸 デオキシリボ核酸 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing |
|---|
 データ登録者 データ登録者 | Davis, E.V. / Hennig, M. / Arya, D.P. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Human Genome Research Institute (NIH/NHGRI) | GM097917 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: An NMR Structure Determination and Analysis of Four Sp1 Consensus Sequences
著者: Davis, E.V. / Hennig, M. / Arya, D.P. |
|---|
| 履歴 | | 登録 | 2018年8月9日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2019年8月14日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年12月18日 | Group: Author supporting evidence / Data collection / カテゴリ: pdbx_audit_support / pdbx_nmr_spectrometer
Item: _pdbx_audit_support.funding_organization / _pdbx_nmr_spectrometer.model |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Database references / Other / カテゴリ: database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード DNA (デオキシリボ核酸) / Sp1 Consensus Sequence
DNA (デオキシリボ核酸) / Sp1 Consensus Sequence デオキシリボ核酸
デオキシリボ核酸 機能・相同性情報
機能・相同性情報
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR /
溶液NMR /  simulated annealing
simulated annealing  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6ed9.cif.gz
6ed9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6ed9.ent.gz
pdb6ed9.ent.gz PDB形式
PDB形式 6ed9.json.gz
6ed9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ed/6ed9
https://data.pdbj.org/pub/pdb/validation_reports/ed/6ed9 ftp://data.pdbj.org/pub/pdb/validation_reports/ed/6ed9
ftp://data.pdbj.org/pub/pdb/validation_reports/ed/6ed9

 リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 simulated annealing / ソフトェア番号: 2
simulated annealing / ソフトェア番号: 2  ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj

