| 登録情報 | データベース: PDB / ID: 5xa1
|
|---|
| タイトル | Crystal structure of inositol 1,4,5-trisphosphate receptor cytosolic domain with inositol 1,4,5-trisphosphate |
|---|
 要素 要素 | Inositol 1,4,5-trisphosphate receptor type 1 |
|---|
 キーワード キーワード | METAL TRANSPORT / receptor channel calcium |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cGMP effects / Elevation of cytosolic Ca2+ levels / Effects of PIP2 hydrolysis / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /  calcineurin complex / platelet dense granule membrane / epithelial fluid transport ...cGMP effects / Elevation of cytosolic Ca2+ levels / Effects of PIP2 hydrolysis / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling / calcineurin complex / platelet dense granule membrane / epithelial fluid transport ...cGMP effects / Elevation of cytosolic Ca2+ levels / Effects of PIP2 hydrolysis / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /  calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / voluntary musculoskeletal movement / Ion homeostasis / calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / voluntary musculoskeletal movement / Ion homeostasis /  inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / GABA-ergic synapse / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol / inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / GABA-ergic synapse / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol /  phosphatidylinositol binding / post-embryonic development / secretory granule membrane / phosphatidylinositol binding / post-embryonic development / secretory granule membrane /  筋小胞体 / 筋小胞体 /  synaptic membrane / synaptic membrane /  liver regeneration / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of neuron projection development / positive regulation of insulin secretion / calcium ion transport / presynapse / liver regeneration / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of neuron projection development / positive regulation of insulin secretion / calcium ion transport / presynapse /  核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / postsynapse / 核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / postsynapse /  protein phosphatase binding / transmembrane transporter binding / protein phosphatase binding / transmembrane transporter binding /  postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding / postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding /  樹状突起 / neuronal cell body / 樹状突起 / neuronal cell body /  シナプス / シナプス /  calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane / calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane /  核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm / 核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm /  小胞体 / protein-containing complex / 小胞体 / protein-containing complex /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 Inositol 1,4,5-trisphosphate receptor / RyR/IP3 receptor binding core, RIH domain superfamily / : / RyR/IP3R Homology associated domain / Inositol 1,4,5-trisphosphate/ryanodine receptor / RIH domain / RyR and IP3R Homology associated / Inositol 1,4,5-trisphosphate/ryanodine receptor / RIH domain / MIR motif ...Inositol 1,4,5-trisphosphate receptor / RyR/IP3 receptor binding core, RIH domain superfamily / : / RyR/IP3R Homology associated domain / Inositol 1,4,5-trisphosphate/ryanodine receptor / RIH domain / RyR and IP3R Homology associated / Inositol 1,4,5-trisphosphate/ryanodine receptor / RIH domain / MIR motif / MIR domain / MIR domain profile. /  Domain in ryanodine and inositol trisphosphate receptors and protein O-mannosyltransferases / Mir domain superfamily / Domain in ryanodine and inositol trisphosphate receptors and protein O-mannosyltransferases / Mir domain superfamily /  Ion transport domain / Ion transport domain /  Ion transport protein類似検索 - ドメイン・相同性 Ion transport protein類似検索 - ドメイン・相同性 D-MYO-INOSITOL-1,4,5-TRIPHOSPHATE / Inositol 1,4,5-trisphosphate receptor type 1類似検索 - 構成要素 |
|---|
| 生物種 |    Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 6.204 Å 分子置換 / 解像度: 6.204 Å |
|---|
 データ登録者 データ登録者 | Hamada, K. / Miyatake, H. / Terauchi, A. / Mikoshiba, K. |
|---|
| 資金援助 |  日本, 1件 日本, 1件 | 組織 | 認可番号 | 国 |
|---|
| JSPS | 15K06791 |  日本 日本 |
|
|---|
 引用 引用 |  ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017
タイトル: IP3-mediated gating mechanism of the IP3 receptor revealed by mutagenesis and X-ray crystallography
著者: Hamada, K. / Miyatake, H. / Terauchi, A. / Mikoshiba, K. |
|---|
| 履歴 | | 登録 | 2017年3月10日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2017年4月19日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年5月17日 | Group: Database references |
|---|
| 改定 1.2 | 2023年11月22日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 calcineurin complex / platelet dense granule membrane / epithelial fluid transport ...cGMP effects / Elevation of cytosolic Ca2+ levels / Effects of PIP2 hydrolysis / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /
calcineurin complex / platelet dense granule membrane / epithelial fluid transport ...cGMP effects / Elevation of cytosolic Ca2+ levels / Effects of PIP2 hydrolysis / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /  calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / voluntary musculoskeletal movement / Ion homeostasis /
calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / voluntary musculoskeletal movement / Ion homeostasis /  inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / GABA-ergic synapse / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol /
inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / GABA-ergic synapse / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol /  phosphatidylinositol binding / post-embryonic development / secretory granule membrane /
phosphatidylinositol binding / post-embryonic development / secretory granule membrane /  筋小胞体 /
筋小胞体 /  synaptic membrane /
synaptic membrane /  liver regeneration / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of neuron projection development / positive regulation of insulin secretion / calcium ion transport / presynapse /
liver regeneration / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of neuron projection development / positive regulation of insulin secretion / calcium ion transport / presynapse /  核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / postsynapse /
核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / postsynapse /  protein phosphatase binding / transmembrane transporter binding /
protein phosphatase binding / transmembrane transporter binding /  postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding /
postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding /  樹状突起 / neuronal cell body /
樹状突起 / neuronal cell body /  シナプス /
シナプス /  calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane /
calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane /  核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm /
核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm /  小胞体 / protein-containing complex /
小胞体 / protein-containing complex /  生体膜 / identical protein binding /
生体膜 / identical protein binding /  細胞膜 /
細胞膜 /  細胞質
細胞質
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 6.204 Å
分子置換 / 解像度: 6.204 Å  データ登録者
データ登録者 日本, 1件
日本, 1件  引用
引用 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017
ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5xa1.cif.gz
5xa1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5xa1.ent.gz
pdb5xa1.ent.gz PDB形式
PDB形式 5xa1.json.gz
5xa1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/xa/5xa1
https://data.pdbj.org/pub/pdb/validation_reports/xa/5xa1 ftp://data.pdbj.org/pub/pdb/validation_reports/xa/5xa1
ftp://data.pdbj.org/pub/pdb/validation_reports/xa/5xa1 リンク
リンク 集合体
集合体


 要素
要素
 Mus musculus (ハツカネズミ) / 遺伝子: Itpr1, Insp3r, Pcd6, Pcp1 / 発現宿主:
Mus musculus (ハツカネズミ) / 遺伝子: Itpr1, Insp3r, Pcd6, Pcp1 / 発現宿主: 
 Spodoptera (蝶・蛾) / 参照: UniProt: P11881
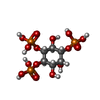
Spodoptera (蝶・蛾) / 参照: UniProt: P11881 イノシトールトリスリン酸
イノシトールトリスリン酸 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL26B2 / 波長: 1 Å
/ ビームライン: BL26B2 / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj