+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5x07 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of FOXA2 DNA binding domain bound to a full consensus DNA site | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA / FOXA2 /  Forkhead domain (フォークヘッドボックスタンパク質) / Consensus binding site / Isothermal Titration Calorimetry / ITC / DNA BINDING PROTEIN-DNA complex Forkhead domain (フォークヘッドボックスタンパク質) / Consensus binding site / Isothermal Titration Calorimetry / ITC / DNA BINDING PROTEIN-DNA complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of transcription from RNA polymerase II promoter by glucose / negative regulation of glucokinase activity / positive regulation of cell-cell adhesion mediated by cadherin / primitive streak formation / positive regulation of embryonic development / positive regulation of transcription from RNA polymerase II promoter by glucose / response to interleukin-6 / endocrine pancreas development / regulation of insulin secretion involved in cellular response to glucose stimulus / negative regulation of epithelial to mesenchymal transition ...negative regulation of transcription from RNA polymerase II promoter by glucose / negative regulation of glucokinase activity / positive regulation of cell-cell adhesion mediated by cadherin / primitive streak formation / positive regulation of embryonic development / positive regulation of transcription from RNA polymerase II promoter by glucose / response to interleukin-6 / endocrine pancreas development / regulation of insulin secretion involved in cellular response to glucose stimulus / negative regulation of epithelial to mesenchymal transition / Formation of definitive endoderm / positive regulation of gastrulation / Formation of axial mesoderm / dopaminergic neuron differentiation / cell fate specification /  regulation of blood coagulation / regulation of blood coagulation /  Regulation of gene expression in beta cells / anatomical structure morphogenesis / adult locomotory behavior / negative regulation of DNA-binding transcription factor activity / sequence-specific double-stranded DNA binding / Regulation of gene expression in beta cells / anatomical structure morphogenesis / adult locomotory behavior / negative regulation of DNA-binding transcription factor activity / sequence-specific double-stranded DNA binding /  細胞結合 / chromatin organization / 細胞結合 / chromatin organization /  細胞分化 / 細胞分化 /  nucleic acid binding / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein domain specific binding / nucleic acid binding / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein domain specific binding /  クロマチン / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / クロマチン / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II /  DNA binding / DNA binding /  核質 / 核質 /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.796 Å 分子置換 / 解像度: 2.796 Å | ||||||||||||
 データ登録者 データ登録者 | Li, J. / Guo, M. / Zhou, Z. / Jiang, L. / Chen, X. / Qu, L. / Wu, D. / Chen, Z. / Chen, L. / Chen, Y. | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2017 ジャーナル: Biochemistry / 年: 2017タイトル: Structure of the Forkhead Domain of FOXA2 Bound to a Complete DNA Consensus Site 著者: Li, J. / Machado, A.C.D. / Guo, M. / Sagendorf, J.M. / Zhou, Z. / Jiang, L. / Chen, X. / Wu, D. / Qu, L. / Chen, Z. / Chen, L. / Rohs, R. / Chen, Y. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5x07.cif.gz 5x07.cif.gz | 278.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5x07.ent.gz pdb5x07.ent.gz | 232.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5x07.json.gz 5x07.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/x0/5x07 https://data.pdbj.org/pub/pdb/validation_reports/x0/5x07 ftp://data.pdbj.org/pub/pdb/validation_reports/x0/5x07 ftp://data.pdbj.org/pub/pdb/validation_reports/x0/5x07 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 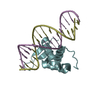
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 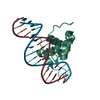
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 4860.159 Da / 分子数: 4 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト)#2: DNA鎖 | 分子量: 4932.272 Da / 分子数: 4 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト)#3: タンパク質 | 分子量: 12392.217 Da / 分子数: 4 / Fragment: UNP residues 157-258 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: FOXA2, HNF3B, TCF3B / 発現宿主: Homo sapiens (ヒト) / 遺伝子: FOXA2, HNF3B, TCF3B / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: Q9Y261 Escherichia coli (大腸菌) / 参照: UniProt: Q9Y261 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.78 Å3/Da / 溶媒含有率: 55.73 % |
|---|---|
結晶化 | 温度: 277 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 80mM Mg (OAc)2, 50mM MES buffer, 16%-20% PEG4K, pH 6.5 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRF SSRF  / ビームライン: BL17U / 波長: 0.9789 Å / ビームライン: BL17U / 波長: 0.9789 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2016年6月1日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9789 Å / 相対比: 1 : 0.9789 Å / 相対比: 1 |
| 反射 | 解像度: 2.796→33.64 Å / Num. obs: 178752 / % possible obs: 96.8 % / 冗長度: 7.5 % / Net I/σ(I): 19.47 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 2.796→33.637 Å / SU ML: 0.49 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 32.22 分子置換 / 解像度: 2.796→33.637 Å / SU ML: 0.49 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 32.22
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.796→33.637 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: 178.8931 Å / Origin y: 40.393 Å / Origin z: 185.4466 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: all |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj









































