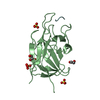
登録情報 データベース : PDB / ID : 5dn2タイトル Human NRP2 b1 domain in complex with the peptide corresponding to the C-terminus of VEGF-A Neuropilin-2 Vascular endothelial growth factor A キーワード / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 1.95 Å データ登録者 Tsai, Y.C.I. / Frankel, P. / Fotinou, C. / Rana, R. / Zachary, I. / Djordjevic, S. ジャーナル : Febs J. / 年 : 2016タイトル : Structural studies of neuropilin-2 reveal a zinc ion binding site remote from the vascular endothelial growth factor binding pocket.著者 : Tsai, Y.C. / Fotinou, C. / Rana, R. / Yelland, T. / Frankel, P. / Zachary, I. / Djordjevic, S. 履歴 登録 2015年9月9日 登録サイト / 処理サイト 改定 1.0 2016年7月20日 Provider / タイプ 改定 1.1 2024年1月10日 Group / Database references / Refinement descriptionカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / diffrn_radiation_wavelength / pdbx_initial_refinement_model / struct_ncs_dom_lim Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ncs_dom_lim.beg_auth_comp_id / _struct_ncs_dom_lim.beg_label_asym_id / _struct_ncs_dom_lim.beg_label_comp_id / _struct_ncs_dom_lim.beg_label_seq_id / _struct_ncs_dom_lim.end_auth_comp_id / _struct_ncs_dom_lim.end_label_asym_id / _struct_ncs_dom_lim.end_label_comp_id / _struct_ncs_dom_lim.end_label_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード SIGNALING PROTEIN /
SIGNALING PROTEIN /  Neuropilin-2 / VEGFA /
Neuropilin-2 / VEGFA /  Angiogenesis (血管新生) /
Angiogenesis (血管新生) /  heparin (ヘパリン) /
heparin (ヘパリン) /  receptor (受容体)
receptor (受容体) 機能・相同性情報
機能・相同性情報 VEGF-A complex / Signaling by VEGF ...vestibulocochlear nerve structural organization / dorsal root ganglion morphogenesis / ventral trunk neural crest cell migration / sympathetic neuron projection guidance / facioacoustic ganglion development / basophil chemotaxis / trigeminal ganglion development / cellular stress response to acid chemical /
VEGF-A complex / Signaling by VEGF ...vestibulocochlear nerve structural organization / dorsal root ganglion morphogenesis / ventral trunk neural crest cell migration / sympathetic neuron projection guidance / facioacoustic ganglion development / basophil chemotaxis / trigeminal ganglion development / cellular stress response to acid chemical /  VEGF-A complex / Signaling by VEGF / lymph vessel morphogenesis / trigeminal nerve structural organization / sensory neuron axon guidance / facial nerve structural organization / gonadotrophin-releasing hormone neuronal migration to the hypothalamus /
VEGF-A complex / Signaling by VEGF / lymph vessel morphogenesis / trigeminal nerve structural organization / sensory neuron axon guidance / facial nerve structural organization / gonadotrophin-releasing hormone neuronal migration to the hypothalamus /  vascular endothelial growth factor receptor 1 binding / negative regulation of establishment of endothelial barrier / branchiomotor neuron axon guidance /
vascular endothelial growth factor receptor 1 binding / negative regulation of establishment of endothelial barrier / branchiomotor neuron axon guidance /  vascular endothelial growth factor receptor binding / VEGF ligand-receptor interactions / positive regulation of mast cell chemotaxis / positive regulation of peptidyl-tyrosine autophosphorylation / negative regulation of adherens junction organization / post-embryonic camera-type eye development / primitive erythrocyte differentiation / positive regulation of protein kinase C signaling / negative regulation of blood-brain barrier permeability / positive regulation of cell proliferation by VEGF-activated platelet derived growth factor receptor signaling pathway / axon extension involved in axon guidance / VEGF-activated neuropilin signaling pathway / positive regulation of vascular endothelial growth factor signaling pathway / coronary vein morphogenesis / cardiac vascular smooth muscle cell development / sympathetic neuron projection extension / NrCAM interactions / Neurophilin interactions with VEGF and VEGFR / positive regulation of trophoblast cell migration / endothelial cell chemotaxis / motor neuron migration / positive regulation of endothelial cell chemotaxis by VEGF-activated vascular endothelial growth factor receptor signaling pathway / eye photoreceptor cell development / sympathetic ganglion development / vascular endothelial growth factor receptor-2 signaling pathway / VEGF binds to VEGFR leading to receptor dimerization / regulation of nitric oxide mediated signal transduction / neural crest cell migration involved in autonomic nervous system development / positive regulation of axon extension involved in axon guidance / positive regulation of cell migration by vascular endothelial growth factor signaling pathway / positive regulation of branching involved in ureteric bud morphogenesis / positive regulation of protein localization to early endosome / vascular wound healing /
vascular endothelial growth factor receptor binding / VEGF ligand-receptor interactions / positive regulation of mast cell chemotaxis / positive regulation of peptidyl-tyrosine autophosphorylation / negative regulation of adherens junction organization / post-embryonic camera-type eye development / primitive erythrocyte differentiation / positive regulation of protein kinase C signaling / negative regulation of blood-brain barrier permeability / positive regulation of cell proliferation by VEGF-activated platelet derived growth factor receptor signaling pathway / axon extension involved in axon guidance / VEGF-activated neuropilin signaling pathway / positive regulation of vascular endothelial growth factor signaling pathway / coronary vein morphogenesis / cardiac vascular smooth muscle cell development / sympathetic neuron projection extension / NrCAM interactions / Neurophilin interactions with VEGF and VEGFR / positive regulation of trophoblast cell migration / endothelial cell chemotaxis / motor neuron migration / positive regulation of endothelial cell chemotaxis by VEGF-activated vascular endothelial growth factor receptor signaling pathway / eye photoreceptor cell development / sympathetic ganglion development / vascular endothelial growth factor receptor-2 signaling pathway / VEGF binds to VEGFR leading to receptor dimerization / regulation of nitric oxide mediated signal transduction / neural crest cell migration involved in autonomic nervous system development / positive regulation of axon extension involved in axon guidance / positive regulation of cell migration by vascular endothelial growth factor signaling pathway / positive regulation of branching involved in ureteric bud morphogenesis / positive regulation of protein localization to early endosome / vascular wound healing /  vascular endothelial growth factor receptor activity / positive regulation of blood vessel endothelial cell proliferation involved in sprouting angiogenesis / nerve development / semaphorin receptor complex / tube formation / camera-type eye morphogenesis / positive regulation of epithelial tube formation /
vascular endothelial growth factor receptor activity / positive regulation of blood vessel endothelial cell proliferation involved in sprouting angiogenesis / nerve development / semaphorin receptor complex / tube formation / camera-type eye morphogenesis / positive regulation of epithelial tube formation /  neuropilin binding / induction of positive chemotaxis / coronary artery morphogenesis / negative regulation of cell-cell adhesion mediated by cadherin /
neuropilin binding / induction of positive chemotaxis / coronary artery morphogenesis / negative regulation of cell-cell adhesion mediated by cadherin /  vascular endothelial growth factor receptor 2 binding / outflow tract septum morphogenesis / semaphorin receptor activity / commissural neuron axon guidance / dopaminergic neuron differentiation / positive regulation of vascular permeability / surfactant homeostasis / positive regulation of endothelial cell chemotaxis /
vascular endothelial growth factor receptor 2 binding / outflow tract septum morphogenesis / semaphorin receptor activity / commissural neuron axon guidance / dopaminergic neuron differentiation / positive regulation of vascular permeability / surfactant homeostasis / positive regulation of endothelial cell chemotaxis /  platelet-derived growth factor receptor binding / cell migration involved in sprouting angiogenesis /
platelet-derived growth factor receptor binding / cell migration involved in sprouting angiogenesis /  extracellular matrix binding / retinal ganglion cell axon guidance / cardiac muscle cell development /
extracellular matrix binding / retinal ganglion cell axon guidance / cardiac muscle cell development /  血管新生 / positive regulation of positive chemotaxis / regulation of postsynapse organization / Regulation of gene expression by Hypoxia-inducible Factor / vascular endothelial growth factor signaling pathway / positive regulation of leukocyte migration / positive regulation of p38MAPK cascade / artery morphogenesis / positive regulation of DNA biosynthetic process / negative chemotaxis / branching involved in blood vessel morphogenesis / positive regulation of neuroblast proliferation / positive regulation of cell migration involved in sprouting angiogenesis / positive chemotaxis /
血管新生 / positive regulation of positive chemotaxis / regulation of postsynapse organization / Regulation of gene expression by Hypoxia-inducible Factor / vascular endothelial growth factor signaling pathway / positive regulation of leukocyte migration / positive regulation of p38MAPK cascade / artery morphogenesis / positive regulation of DNA biosynthetic process / negative chemotaxis / branching involved in blood vessel morphogenesis / positive regulation of neuroblast proliferation / positive regulation of cell migration involved in sprouting angiogenesis / positive chemotaxis /  transmembrane receptor protein tyrosine kinase activator activity /
transmembrane receptor protein tyrosine kinase activator activity /  cytokine binding / positive regulation of sprouting angiogenesis / outflow tract morphogenesis /
cytokine binding / positive regulation of sprouting angiogenesis / outflow tract morphogenesis /  chemoattractant activity / activation of protein kinase activity /
chemoattractant activity / activation of protein kinase activity /  growth factor binding / positive regulation of focal adhesion assembly / mesoderm development / monocyte differentiation / positive regulation of receptor internalization
growth factor binding / positive regulation of focal adhesion assembly / mesoderm development / monocyte differentiation / positive regulation of receptor internalization
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.95 Å
分子置換 / 解像度: 1.95 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Febs J. / 年: 2016
ジャーナル: Febs J. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5dn2.cif.gz
5dn2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5dn2.ent.gz
pdb5dn2.ent.gz PDB形式
PDB形式 5dn2.json.gz
5dn2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/dn/5dn2
https://data.pdbj.org/pub/pdb/validation_reports/dn/5dn2 ftp://data.pdbj.org/pub/pdb/validation_reports/dn/5dn2
ftp://data.pdbj.org/pub/pdb/validation_reports/dn/5dn2
 リンク
リンク 集合体
集合体



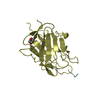
 要素
要素 Neuropilin / Vascular endothelial cell growth factor 165 receptor 2
Neuropilin / Vascular endothelial cell growth factor 165 receptor 2
 Homo sapiens (ヒト) / 遺伝子: NRP2, VEGF165R2 / プラスミド: pET15b-TEV / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: NRP2, VEGF165R2 / プラスミド: pET15b-TEV / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): Rosetta-gami 2(DE3)pLysS / 参照: UniProt: O60462
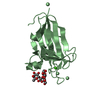
Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): Rosetta-gami 2(DE3)pLysS / 参照: UniProt: O60462 / VEGF-A / Vascular permeability factor / VPF
/ VEGF-A / Vascular permeability factor / VPF
 Homo sapiens (ヒト) / 遺伝子: VEGFA, VEGF / プラスミド: pET14b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: VEGFA, VEGF / プラスミド: pET14b / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): Shuffle / 参照: UniProt: P15692
Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): Shuffle / 参照: UniProt: P15692 1,4-ジオキサン
1,4-ジオキサン グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.97625 Å
/ ビームライン: I03 / 波長: 0.97625 Å : 0.97625 Å / 相対比: 1
: 0.97625 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


















