+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4qwx | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | yCP in complex with the epoxyketone inhibitor ONX 0914 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR /  Cancer (悪性腫瘍) / Cancer (悪性腫瘍) /  Proteasome (プロテアソーム) / Proteasome (プロテアソーム) /  Bortezomib (ボルテゾミブ) / Bortezomib (ボルテゾミブ) /  Drug Resistance (薬剤耐性) / Binding Analysis / HYDROLASE-HYDROLASE INHIBITOR complex Drug Resistance (薬剤耐性) / Binding Analysis / HYDROLASE-HYDROLASE INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proteasome core complex assembly / nuclear outer membrane-endoplasmic reticulum membrane network / Cross-presentation of soluble exogenous antigens (endosomes) / TNFR2 non-canonical NF-kB pathway / proteasomal ubiquitin-independent protein catabolic process / Ub-specific processing proteases / proteasome storage granule / endopeptidase activator activity /  proteasome assembly / proteasome assembly /  proteasome endopeptidase complex ...proteasome core complex assembly / nuclear outer membrane-endoplasmic reticulum membrane network / Cross-presentation of soluble exogenous antigens (endosomes) / TNFR2 non-canonical NF-kB pathway / proteasomal ubiquitin-independent protein catabolic process / Ub-specific processing proteases / proteasome storage granule / endopeptidase activator activity / proteasome endopeptidase complex ...proteasome core complex assembly / nuclear outer membrane-endoplasmic reticulum membrane network / Cross-presentation of soluble exogenous antigens (endosomes) / TNFR2 non-canonical NF-kB pathway / proteasomal ubiquitin-independent protein catabolic process / Ub-specific processing proteases / proteasome storage granule / endopeptidase activator activity /  proteasome assembly / proteasome assembly /  proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome core complex, alpha-subunit complex / threonine-type endopeptidase activity / Neutrophil degranulation / proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome core complex, alpha-subunit complex / threonine-type endopeptidase activity / Neutrophil degranulation /  proteasome complex / proteasomal protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process / proteasome complex / proteasomal protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process /  endopeptidase activity / endopeptidase activity /  mRNA binding / endoplasmic reticulum membrane / mRNA binding / endoplasmic reticulum membrane /  ミトコンドリア / ミトコンドリア /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.9 Å 分子置換 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Huber, E.M. / Heinemeyer, W. / Groll, M. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2015 ジャーナル: Structure / 年: 2015タイトル: Bortezomib-Resistant Mutant Proteasomes: Structural and Biochemical Evaluation with Carfilzomib and ONX 0914. 著者: Huber, E.M. / Heinemeyer, W. / Groll, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4qwx.cif.gz 4qwx.cif.gz | 2.4 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4qwx.ent.gz pdb4qwx.ent.gz | 2 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4qwx.json.gz 4qwx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qw/4qwx https://data.pdbj.org/pub/pdb/validation_reports/qw/4qwx ftp://data.pdbj.org/pub/pdb/validation_reports/qw/4qwx ftp://data.pdbj.org/pub/pdb/validation_reports/qw/4qwx | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  4quxC  4quyC  4qv0C  4qv1C  4qv3C  4qv4C  4qv5C  4qv6C  4qv7C  4qv8C  4qv9C  4qvlC  4qvmC  4qvnC  4qvpC  4qvqC  4qvvC  4qvwC  4qvyC  4qw0C  4qw1C  4qw3C  4qw4C  4qw5C  4qw6C  4qw7C  4qwfC  4qwgC  4qwiC  4qwjC  4qwkC  4qwlC  4qwrC  4qwsC  4qwuC  4qxjC  4qz0C  4qz1C  4qz2C  4qz3C  4qz4C  4qz5C  4qz6C  4qz7C  4qzwC  4qzxC  4qzzC  4r00C  1rypS C: 同じ文献を引用 ( S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
| ||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
-PROTEASOME SUBUNIT ALPHA TYPE- ... , 6種, 12分子 AOBPCQDRESGU
| #1: タンパク質 |  プロテアソーム / MACROPAIN SUBUNIT Y7 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y7 / PROTEASOME COMPONENT Y7 / ...MACROPAIN SUBUNIT Y7 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y7 / PROTEASOME COMPONENT Y7 / PROTEINASE YSCE SUBUNIT 7 プロテアソーム / MACROPAIN SUBUNIT Y7 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y7 / PROTEASOME COMPONENT Y7 / ...MACROPAIN SUBUNIT Y7 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y7 / PROTEASOME COMPONENT Y7 / PROTEINASE YSCE SUBUNIT 7分子量: 27191.828 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P23639, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P23639,  proteasome endopeptidase complex proteasome endopeptidase complex#2: タンパク質 |  プロテアソーム / MACROPAIN SUBUNIT Y13 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y13 / PROTEASOME COMPONENT Y13 ...MACROPAIN SUBUNIT Y13 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y13 / PROTEASOME COMPONENT Y13 / PROTEINASE YSCE SUBUNIT 13 プロテアソーム / MACROPAIN SUBUNIT Y13 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y13 / PROTEASOME COMPONENT Y13 ...MACROPAIN SUBUNIT Y13 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT Y13 / PROTEASOME COMPONENT Y13 / PROTEINASE YSCE SUBUNIT 13分子量: 28748.230 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P23638, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P23638,  proteasome endopeptidase complex proteasome endopeptidase complex#3: タンパク質 |  プロテアソーム / MACROPAIN SUBUNIT PRE6 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE6 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE6 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE6 / PROTEASOME COMPONENT PRE6 / PROTEINASE YSCE SUBUNIT PRE6 プロテアソーム / MACROPAIN SUBUNIT PRE6 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE6 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE6 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE6 / PROTEASOME COMPONENT PRE6 / PROTEINASE YSCE SUBUNIT PRE6分子量: 28478.111 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P40303, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P40303,  proteasome endopeptidase complex proteasome endopeptidase complex#4: タンパク質 |  プロテアソーム / MACROPAIN SUBUNIT PUP2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP2 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PUP2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP2 / PROTEASOME COMPONENT PUP2 / PROTEINASE YSCE SUBUNIT PUP2 プロテアソーム / MACROPAIN SUBUNIT PUP2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP2 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PUP2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP2 / PROTEASOME COMPONENT PUP2 / PROTEINASE YSCE SUBUNIT PUP2分子量: 28649.086 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P32379, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P32379,  proteasome endopeptidase complex proteasome endopeptidase complex#5: タンパク質 |  プロテアソーム / MACROPAIN SUBUNIT PRE5 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE5 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE5 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE5 / PROTEASOME COMPONENT PRE5 / PROTEINASE YSCE SUBUNIT PRE5 プロテアソーム / MACROPAIN SUBUNIT PRE5 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE5 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE5 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE5 / PROTEASOME COMPONENT PRE5 / PROTEINASE YSCE SUBUNIT PRE5分子量: 25634.000 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P40302, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P40302,  proteasome endopeptidase complex proteasome endopeptidase complex#7: タンパク質 |  プロテアソーム / MACROPAIN SUBUNIT C7-ALPHA / MULTICATALYTIC ENDOPEPTIDASE COMPLEX C7 / PROTEASOME COMPONENT C7- ...MACROPAIN SUBUNIT C7-ALPHA / MULTICATALYTIC ENDOPEPTIDASE COMPLEX C7 / PROTEASOME COMPONENT C7-ALPHA / PROTEASOME COMPONENT Y8 / PROTEINASE YSCE SUBUNIT 7 / SCL1 SUPPRESSOR PROTEIN プロテアソーム / MACROPAIN SUBUNIT C7-ALPHA / MULTICATALYTIC ENDOPEPTIDASE COMPLEX C7 / PROTEASOME COMPONENT C7- ...MACROPAIN SUBUNIT C7-ALPHA / MULTICATALYTIC ENDOPEPTIDASE COMPLEX C7 / PROTEASOME COMPONENT C7-ALPHA / PROTEASOME COMPONENT Y8 / PROTEINASE YSCE SUBUNIT 7 / SCL1 SUPPRESSOR PROTEIN分子量: 28033.830 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P21243, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P21243,  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|
-タンパク質 , 1種, 2分子 FT
| #6: タンパク質 | 分子量: 31575.068 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P21242, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P21242,  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|
-PROTEASOME SUBUNIT BETA TYPE- ... , 7種, 14分子 HVIWJXKYLZMaNb
| #8: タンパク質 |  PSMB2 / MACROPAIN SUBUNIT PUP1 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP1 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PUP1 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP1 / PROTEASOME COMPONENT PUP1 / PROTEINASE YSCE SUBUNIT PUP1 PSMB2 / MACROPAIN SUBUNIT PUP1 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP1 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PUP1 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP1 / PROTEASOME COMPONENT PUP1 / PROTEINASE YSCE SUBUNIT PUP1分子量: 25114.459 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P25043, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P25043,  proteasome endopeptidase complex proteasome endopeptidase complex#9: タンパク質 |  PSMB3 / MACROPAIN SUBUNIT PUP3 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP3 / PROTEASOME COMPONENT PUP3 PSMB3 / MACROPAIN SUBUNIT PUP3 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PUP3 / PROTEASOME COMPONENT PUP3分子量: 22627.842 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P25451, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P25451,  proteasome endopeptidase complex proteasome endopeptidase complex#10: タンパク質 |  PSMB4 / MACROPAIN SUBUNIT C11 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT C11 / PROTEASOME COMPONENT C11 ...MACROPAIN SUBUNIT C11 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT C11 / PROTEASOME COMPONENT C11 / PROTEINASE YSCE SUBUNIT 11 PSMB4 / MACROPAIN SUBUNIT C11 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT C11 / PROTEASOME COMPONENT C11 ...MACROPAIN SUBUNIT C11 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT C11 / PROTEASOME COMPONENT C11 / PROTEINASE YSCE SUBUNIT 11分子量: 22545.676 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P22141, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P22141,  proteasome endopeptidase complex proteasome endopeptidase complex#11: タンパク質 |  PSMB5 / MACROPAIN SUBUNIT PRE2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE2 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE2 / PROTEASOME COMPONENT PRE2 / PROTEINASE YSCE SUBUNIT PRE2 PSMB5 / MACROPAIN SUBUNIT PRE2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE2 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE2 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE2 / PROTEASOME COMPONENT PRE2 / PROTEINASE YSCE SUBUNIT PRE2分子量: 23325.248 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)株: ATCC 204508 / S288c / 遺伝子: PRE2, DOA3, PRG1, YPR103W, P8283.10 / 発現宿主:   SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): 4932 / 参照: UniProt: P30656, SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): 4932 / 参照: UniProt: P30656,  proteasome endopeptidase complex proteasome endopeptidase complex#12: タンパク質 |  プロテアソーム / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT C5 / PROTEASOME COMPONENT C5 プロテアソーム / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT C5 / PROTEASOME COMPONENT C5分子量: 24883.928 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P23724, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P23724,  proteasome endopeptidase complex proteasome endopeptidase complex#13: タンパク質 |  プロテアソーム / MACROPAIN SUBUNIT PRE4 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE4 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE4 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE4 / PROTEASOME COMPONENT PRE4 / PROTEINASE YSCE SUBUNIT PRE4 プロテアソーム / MACROPAIN SUBUNIT PRE4 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE4 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE4 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE4 / PROTEASOME COMPONENT PRE4 / PROTEINASE YSCE SUBUNIT PRE4分子量: 27200.893 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P30657, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P30657,  proteasome endopeptidase complex proteasome endopeptidase complex#14: タンパク質 |  PSMB1 / MACROPAIN SUBUNIT PRE3 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE3 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE3 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE3 / PROTEASOME COMPONENT PRE3 / PROTEINASE YSCE SUBUNIT PRE3 PSMB1 / MACROPAIN SUBUNIT PRE3 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE3 / PROTEASOME COMPONENT ...MACROPAIN SUBUNIT PRE3 / MULTICATALYTIC ENDOPEPTIDASE COMPLEX SUBUNIT PRE3 / PROTEASOME COMPONENT PRE3 / PROTEINASE YSCE SUBUNIT PRE3分子量: 21517.186 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P38624, Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c / 参照: UniProt: P38624,  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|
-非ポリマー , 6種, 228分子 

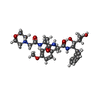








| #15: 化合物 | ChemComp-MG / #16: 化合物 |  塩化物 塩化物#17: 化合物 | ChemComp-04C / #18: 化合物 | ChemComp-MES /  MES (緩衝剤) MES (緩衝剤)#19: 化合物 | #20: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.67 Å3/Da / 溶媒含有率: 66.52 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.8 詳細: 20 MM MGAC2, 13% MPD, VAPOR DIFFUSION, HANGING DROP, TEMPERATURE 293K, pH 6.8 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06SA / 波長: 1 / 波長: 1 Å / ビームライン: X06SA / 波長: 1 / 波長: 1 Å |
| 検出器 | タイプ: PSI PILATUS 6M / 検出器: PIXEL / 日付: 2013年9月7日 |
| 放射 | モノクロメーター: LN2 COOLED FIXED-EXIT. SI(111) MONOCHROMATOR プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.9→30 Å / Num. all: 236478 / Num. obs: 226546 / % possible obs: 95.8 % / Observed criterion σ(F): 2 / Observed criterion σ(I): 2 / Rmerge(I) obs: 0.087 / Net I/σ(I): 9.8 |
| 反射 シェル | 解像度: 2.9→3 Å / Rmerge(I) obs: 0.539 / Mean I/σ(I) obs: 2.1 / % possible all: 97.2 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 1RYP 解像度: 2.9→15 Å / Cor.coef. Fo:Fc: 0.96 / Cor.coef. Fo:Fc free: 0.938 / SU B: 31.248 / SU ML: 0.249 / 交差検証法: THROUGHOUT / ESU R Free: 0.305 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 75.235 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.9→15 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.9→2.972 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー
























 PDBj
PDBj

















