登録情報 データベース : PDB / ID : 4k9bタイトル Structure of Ternary Complex of cGAS with dsDNA and Bound c[G(2 ,5 )pA(3 ,5 )p] Cyclic GMP-AMP synthase DNA-F DNA-R キーワード / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Mus musculus (ハツカネズミ)手法 / / / 解像度 : 2.26 Å データ登録者 Gao, P. / Wu, Y. / Patel, D.J. ジャーナル : Cell(Cambridge,Mass.) / 年 : 2013タイトル : Cyclic [G(2',5')pA(3',5')p] is the metazoan second messenger produced by DNA-activated cyclic GMP-AMP synthase.著者 : Gao, P. / Ascano, M. / Wu, Y. / Barchet, W. / Gaffney, B.L. / Zillinger, T. / Serganov, A.A. / Liu, Y. / Jones, R.A. / Hartmann, G. / Tuschl, T. / Patel, D.J. 履歴 登録 2013年4月19日 登録サイト / 処理サイト 改定 1.0 2013年5月15日 Provider / タイプ 改定 1.1 2013年8月28日 Group 改定 1.2 2024年2月28日 Group / Database references / Derived calculationsカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / struct_conn / struct_conn_type / struct_ref_seq_dif / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn_type.id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity /
cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity /  傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / negative regulation of DNA repair / cGAS/STING signaling pathway / regulation of immunoglobulin production /
傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / negative regulation of DNA repair / cGAS/STING signaling pathway / regulation of immunoglobulin production /  regulation of T cell activation / pattern recognition receptor signaling pathway ...regulation of type I interferon production /
regulation of T cell activation / pattern recognition receptor signaling pathway ...regulation of type I interferon production /  cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity /
cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity /  傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / negative regulation of DNA repair / cGAS/STING signaling pathway / regulation of immunoglobulin production /
傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / negative regulation of DNA repair / cGAS/STING signaling pathway / regulation of immunoglobulin production /  regulation of T cell activation / pattern recognition receptor signaling pathway / negative regulation of double-strand break repair via homologous recombination / negative regulation of cGAS/STING signaling pathway / cellular response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / cGMP-mediated signaling / cAMP-mediated signaling /
regulation of T cell activation / pattern recognition receptor signaling pathway / negative regulation of double-strand break repair via homologous recombination / negative regulation of cGAS/STING signaling pathway / cellular response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / cGMP-mediated signaling / cAMP-mediated signaling /  nucleosome binding / positive regulation of type I interferon production /
nucleosome binding / positive regulation of type I interferon production /  regulation of immune response / positive regulation of defense response to virus by host /
regulation of immune response / positive regulation of defense response to virus by host /  phosphatidylinositol-4,5-bisphosphate binding / activation of innate immune response / molecular condensate scaffold activity / determination of adult lifespan / positive regulation of cellular senescence / site of double-strand break /
phosphatidylinositol-4,5-bisphosphate binding / activation of innate immune response / molecular condensate scaffold activity / determination of adult lifespan / positive regulation of cellular senescence / site of double-strand break /  double-stranded DNA binding / defense response to virus /
double-stranded DNA binding / defense response to virus /  nuclear body /
nuclear body /  DNA修復 /
DNA修復 /  自然免疫系 / DNA damage response /
自然免疫系 / DNA damage response /  chromatin binding / GTP binding / protein homodimerization activity /
chromatin binding / GTP binding / protein homodimerization activity /  DNA binding /
DNA binding /  核質 /
核質 /  ATP binding /
ATP binding /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.26 Å
分子置換 / 解像度: 2.26 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell(Cambridge,Mass.) / 年: 2013
ジャーナル: Cell(Cambridge,Mass.) / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4k9b.cif.gz
4k9b.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4k9b.ent.gz
pdb4k9b.ent.gz PDB形式
PDB形式 4k9b.json.gz
4k9b.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/k9/4k9b
https://data.pdbj.org/pub/pdb/validation_reports/k9/4k9b ftp://data.pdbj.org/pub/pdb/validation_reports/k9/4k9b
ftp://data.pdbj.org/pub/pdb/validation_reports/k9/4k9b リンク
リンク 集合体
集合体
 要素
要素 / cGAMP synthase / cGAS / m-cGAS / Mab-21 domain-containing protein 1
/ cGAMP synthase / cGAS / m-cGAS / Mab-21 domain-containing protein 1
 Mus musculus (ハツカネズミ) / 遺伝子: Mb21d1 / 発現宿主:
Mus musculus (ハツカネズミ) / 遺伝子: Mb21d1 / 発現宿主: 
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) 転移酵素; リンを含む基を移すもの; 核酸を移すもの
転移酵素; リンを含む基を移すもの; 核酸を移すもの

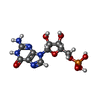
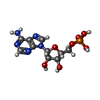





 リン酸塩
リン酸塩 グアニル酸
グアニル酸 アデニル酸
アデニル酸 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 24-ID-E / 波長: 0.9792 Å
/ ビームライン: 24-ID-E / 波長: 0.9792 Å : 0.9792 Å / 相対比: 1
: 0.9792 Å / 相対比: 1  解析
解析 :
:  分子置換 / 解像度: 2.26→42.656 Å / SU ML: 0.26 / σ(F): 1.34 / 位相誤差: 22.77 / 立体化学のターゲット値: ML
分子置換 / 解像度: 2.26→42.656 Å / SU ML: 0.26 / σ(F): 1.34 / 位相誤差: 22.77 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj












































