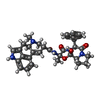
| 登録情報 | データベース: PDB / ID: 4iar
|
|---|
| タイトル | Crystal structure of the chimeric protein of 5-HT1B-BRIL in complex with ergotamine (PSI Community Target) |
|---|
 要素 要素 | Chimera protein of human 5-hydroxytryptamine receptor 1B and E. Coli soluble cytochrome b562 |
|---|
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  ELECTRON TRANSPORT (電子伝達系) / ELECTRON TRANSPORT (電子伝達系) /  ergotamine (エルゴタミン) / Novel protein engineering / GPCR Network / ergotamine (エルゴタミン) / Novel protein engineering / GPCR Network /  Membrane protein (膜タンパク質) / PSI-Biology / Membrane protein (膜タンパク質) / PSI-Biology /  Structural Genomics (構造ゲノミクス) / Structural Genomics (構造ゲノミクス) /  GPCR (Gタンパク質共役受容体) / GPCR Dock GPCR (Gタンパク質共役受容体) / GPCR Dock |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
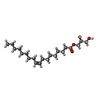
voltage-gated calcium channel activity involved in regulation of presynaptic cytosolic calcium levels / adenylate cyclase-inhibiting serotonin receptor signaling pathway / negative regulation of gamma-aminobutyric acid secretion / G protein-coupled serotonin receptor complex / serotonergic synapse /  regulation of behavior / regulation of behavior /  セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion ...voltage-gated calcium channel activity involved in regulation of presynaptic cytosolic calcium levels / adenylate cyclase-inhibiting serotonin receptor signaling pathway / negative regulation of gamma-aminobutyric acid secretion / G protein-coupled serotonin receptor complex / serotonergic synapse / セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion ...voltage-gated calcium channel activity involved in regulation of presynaptic cytosolic calcium levels / adenylate cyclase-inhibiting serotonin receptor signaling pathway / negative regulation of gamma-aminobutyric acid secretion / G protein-coupled serotonin receptor complex / serotonergic synapse /  regulation of behavior / regulation of behavior /  セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion / セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion /  急性アルコール中毒 / cellular response to temperature stimulus / 急性アルコール中毒 / cellular response to temperature stimulus /  serotonin binding / serotonin binding /  bone remodeling / negative regulation of synaptic transmission, glutamatergic / G protein-coupled serotonin receptor activity / bone remodeling / negative regulation of synaptic transmission, glutamatergic / G protein-coupled serotonin receptor activity /  血管収縮 / protein kinase C-activating G protein-coupled receptor signaling pathway / 血管収縮 / protein kinase C-activating G protein-coupled receptor signaling pathway /  neurotransmitter receptor activity / G protein-coupled receptor internalization / regulation of synaptic vesicle exocytosis / regulation of dopamine secretion / neurotransmitter receptor activity / G protein-coupled receptor internalization / regulation of synaptic vesicle exocytosis / regulation of dopamine secretion /  heterocyclic compound binding / cellular response to alkaloid / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / heterocyclic compound binding / cellular response to alkaloid / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger /  ヘルト萼状シナプス / positive regulation of vascular associated smooth muscle cell proliferation / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / presynaptic modulation of chemical synaptic transmission / response to cocaine / cellular response to xenobiotic stimulus / ヘルト萼状シナプス / positive regulation of vascular associated smooth muscle cell proliferation / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / presynaptic modulation of chemical synaptic transmission / response to cocaine / cellular response to xenobiotic stimulus /  presynaptic membrane / G alpha (i) signalling events / chemical synaptic transmission / response to ethanol / presynaptic membrane / G alpha (i) signalling events / chemical synaptic transmission / response to ethanol /  electron transfer activity / electron transfer activity /  ペリプラズム / iron ion binding / ペリプラズム / iron ion binding /  樹状突起 / 樹状突起 /  heme binding / heme binding /  小胞体 / 小胞体 /  細胞膜類似検索 - 分子機能 細胞膜類似検索 - 分子機能 5-Hydroxytryptamine 1B receptor /  5-hydroxytryptamine receptor family / Cytochrome c/b562 / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / Cytochrome b562 / Cytochrome b562 / Cytochrome c/b562 / Serpentine type 7TM GPCR chemoreceptor Srsx / Four Helix Bundle (Hemerythrin (Met), subunit A) ...5-Hydroxytryptamine 1B receptor / 5-hydroxytryptamine receptor family / Cytochrome c/b562 / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / Cytochrome b562 / Cytochrome b562 / Cytochrome c/b562 / Serpentine type 7TM GPCR chemoreceptor Srsx / Four Helix Bundle (Hemerythrin (Met), subunit A) ...5-Hydroxytryptamine 1B receptor /  5-hydroxytryptamine receptor family / Cytochrome c/b562 / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / Cytochrome b562 / Cytochrome b562 / Cytochrome c/b562 / Serpentine type 7TM GPCR chemoreceptor Srsx / Four Helix Bundle (Hemerythrin (Met), subunit A) / G-protein coupled receptors family 1 signature. / G protein-coupled receptor, rhodopsin-like / 5-hydroxytryptamine receptor family / Cytochrome c/b562 / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / Cytochrome b562 / Cytochrome b562 / Cytochrome c/b562 / Serpentine type 7TM GPCR chemoreceptor Srsx / Four Helix Bundle (Hemerythrin (Met), subunit A) / G-protein coupled receptors family 1 signature. / G protein-coupled receptor, rhodopsin-like /  GPCR, rhodopsin-like, 7TM / G-protein coupled receptors family 1 profile. / 7 transmembrane receptor (rhodopsin family) / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 GPCR, rhodopsin-like, 7TM / G-protein coupled receptors family 1 profile. / 7 transmembrane receptor (rhodopsin family) / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性  エルゴタミン / (2S)-2,3-dihydroxypropyl (9Z)-octadec-9-enoate / Soluble cytochrome b562 / 5-hydroxytryptamine receptor 1B類似検索 - 構成要素 エルゴタミン / (2S)-2,3-dihydroxypropyl (9Z)-octadec-9-enoate / Soluble cytochrome b562 / 5-hydroxytryptamine receptor 1B類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)
   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å |
|---|
 データ登録者 データ登録者 | Wang, C. / Jiang, Y. / Ma, J. / Wu, H. / Wacker, D. / Katritch, V. / Han, G.W. / Liu, W. / Huang, X. / Vardy, E. ...Wang, C. / Jiang, Y. / Ma, J. / Wu, H. / Wacker, D. / Katritch, V. / Han, G.W. / Liu, W. / Huang, X. / Vardy, E. / McCorvy, J.D. / Gao, X. / Zhou, E.X. / Melcher, K. / Zhang, C. / Bai, F. / Yang, H. / Yang, L. / Jiang, H. / Roth, B.L. / Cherezov, V. / Stevens, R.C. / Xu, H.E. / GPCR Network (GPCR) |
|---|
 引用 引用 |  ジャーナル: Science / 年: 2013 ジャーナル: Science / 年: 2013
タイトル: Structural basis for molecular recognition at serotonin receptors.
著者: Wang, C. / Jiang, Y. / Ma, J. / Wu, H. / Wacker, D. / Katritch, V. / Han, G.W. / Liu, W. / Huang, X.P. / Vardy, E. / McCorvy, J.D. / Gao, X. / Zhou, X.E. / Melcher, K. / Zhang, C. / Bai, F. / ...著者: Wang, C. / Jiang, Y. / Ma, J. / Wu, H. / Wacker, D. / Katritch, V. / Han, G.W. / Liu, W. / Huang, X.P. / Vardy, E. / McCorvy, J.D. / Gao, X. / Zhou, X.E. / Melcher, K. / Zhang, C. / Bai, F. / Yang, H. / Yang, L. / Jiang, H. / Roth, B.L. / Cherezov, V. / Stevens, R.C. / Xu, H.E. |
|---|
| 履歴 | | 登録 | 2012年12月7日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2013年3月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2013年4月10日 | Group: Database references |
|---|
| 改定 1.2 | 2013年5月15日 | Group: Database references |
|---|
| 改定 1.3 | 2013年12月18日 | Group: Derived calculations |
|---|
| 改定 1.4 | 2017年6月7日 | Group: Database references / Structure summary |
|---|
| 改定 1.5 | 2017年11月15日 | Group: Refinement description / カテゴリ: software / Item: _software.name |
|---|
| 改定 1.6 | 2022年2月2日 | Group: Database references / Derived calculations
カテゴリ: database_2 / struct_ref_seq ...database_2 / struct_ref_seq / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq.pdbx_auth_seq_align_end / _struct_ref_seq.seq_align_end / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.7 | 2023年9月20日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素
 キーワード
キーワード SIGNALING PROTEIN /
SIGNALING PROTEIN /  ELECTRON TRANSPORT (電子伝達系) /
ELECTRON TRANSPORT (電子伝達系) /  ergotamine (エルゴタミン) / Novel protein engineering / GPCR Network /
ergotamine (エルゴタミン) / Novel protein engineering / GPCR Network /  Membrane protein (膜タンパク質) / PSI-Biology /
Membrane protein (膜タンパク質) / PSI-Biology /  Structural Genomics (構造ゲノミクス) /
Structural Genomics (構造ゲノミクス) /  GPCR (Gタンパク質共役受容体) / GPCR Dock
GPCR (Gタンパク質共役受容体) / GPCR Dock 機能・相同性情報
機能・相同性情報 regulation of behavior /
regulation of behavior /  セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion ...voltage-gated calcium channel activity involved in regulation of presynaptic cytosolic calcium levels / adenylate cyclase-inhibiting serotonin receptor signaling pathway / negative regulation of gamma-aminobutyric acid secretion / G protein-coupled serotonin receptor complex / serotonergic synapse /
セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion ...voltage-gated calcium channel activity involved in regulation of presynaptic cytosolic calcium levels / adenylate cyclase-inhibiting serotonin receptor signaling pathway / negative regulation of gamma-aminobutyric acid secretion / G protein-coupled serotonin receptor complex / serotonergic synapse /  regulation of behavior /
regulation of behavior /  セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion /
セロトニン受容体 / negative regulation of synaptic transmission, GABAergic / response to mineralocorticoid / negative regulation of serotonin secretion /  急性アルコール中毒 / cellular response to temperature stimulus /
急性アルコール中毒 / cellular response to temperature stimulus /  serotonin binding /
serotonin binding /  bone remodeling / negative regulation of synaptic transmission, glutamatergic / G protein-coupled serotonin receptor activity /
bone remodeling / negative regulation of synaptic transmission, glutamatergic / G protein-coupled serotonin receptor activity /  血管収縮 / protein kinase C-activating G protein-coupled receptor signaling pathway /
血管収縮 / protein kinase C-activating G protein-coupled receptor signaling pathway /  neurotransmitter receptor activity / G protein-coupled receptor internalization / regulation of synaptic vesicle exocytosis / regulation of dopamine secretion /
neurotransmitter receptor activity / G protein-coupled receptor internalization / regulation of synaptic vesicle exocytosis / regulation of dopamine secretion /  heterocyclic compound binding / cellular response to alkaloid / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger /
heterocyclic compound binding / cellular response to alkaloid / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger /  ヘルト萼状シナプス / positive regulation of vascular associated smooth muscle cell proliferation / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / presynaptic modulation of chemical synaptic transmission / response to cocaine / cellular response to xenobiotic stimulus /
ヘルト萼状シナプス / positive regulation of vascular associated smooth muscle cell proliferation / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / presynaptic modulation of chemical synaptic transmission / response to cocaine / cellular response to xenobiotic stimulus /  presynaptic membrane / G alpha (i) signalling events / chemical synaptic transmission / response to ethanol /
presynaptic membrane / G alpha (i) signalling events / chemical synaptic transmission / response to ethanol /  electron transfer activity /
electron transfer activity /  ペリプラズム / iron ion binding /
ペリプラズム / iron ion binding /  樹状突起 /
樹状突起 /  heme binding /
heme binding /  小胞体 /
小胞体 /  細胞膜
細胞膜
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.7 Å
分子置換 / 解像度: 2.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Science / 年: 2013
ジャーナル: Science / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4iar.cif.gz
4iar.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4iar.ent.gz
pdb4iar.ent.gz PDB形式
PDB形式 4iar.json.gz
4iar.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ia/4iar
https://data.pdbj.org/pub/pdb/validation_reports/ia/4iar ftp://data.pdbj.org/pub/pdb/validation_reports/ia/4iar
ftp://data.pdbj.org/pub/pdb/validation_reports/ia/4iar リンク
リンク 集合体
集合体
 要素
要素

 Homo sapiens (ヒト), (組換発現)
Homo sapiens (ヒト), (組換発現) 
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Spodoptera frugiperda (ツマジロクサヨトウ)
Spodoptera frugiperda (ツマジロクサヨトウ) エルゴタミン
エルゴタミン 水
水 X線回折 / 使用した結晶の数: 47
X線回折 / 使用した結晶の数: 47  試料調製
試料調製
 : 1.033 Å / 相対比: 1
: 1.033 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj