+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4i6s | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of RSL mutant W76A in complex with L-fucose | ||||||
 要素 要素 | Putative fucose-binding lectin protein | ||||||
 キーワード キーワード | SUGAR BINDING PROTEIN /  lectin (レクチン) / lectin (レクチン) /  beta-propeller (Βプロペラドメイン) / beta-propeller (Βプロペラドメイン) /  L-fucose (フコース) / L-fucose (フコース) /  multivalency (原子価) / Trivalent Fucose binding lectin / Fucosylated oligosaccharides binding / multivalency (原子価) / Trivalent Fucose binding lectin / Fucosylated oligosaccharides binding /  Soluble (溶解度) Soluble (溶解度) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |   Ralstonia solanacearum (青枯病菌) Ralstonia solanacearum (青枯病菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.54 Å 分子置換 / 解像度: 1.54 Å | ||||||
 データ登録者 データ登録者 | Audfray, A. / Arnaud, J. / Varrot, A. / Imberty, A. | ||||||
 引用 引用 |  ジャーナル: Acs Chem.Biol. / 年: 2013 ジャーナル: Acs Chem.Biol. / 年: 2013タイトル: Reduction of lectin valency drastically changes glycolipid dynamics in membranes but not surface avidity 著者: Arnaud, J. / Claudinon, J. / Trondle, K. / Trovaslet, M. / Larson, G. / Thomas, A. / Varrot, A. / Romer, W. / Imberty, A. / Audfray, A. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET DETERMINATION METHOD: AUTHOR DETERMINED |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4i6s.cif.gz 4i6s.cif.gz | 122.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4i6s.ent.gz pdb4i6s.ent.gz | 95.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4i6s.json.gz 4i6s.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i6/4i6s https://data.pdbj.org/pub/pdb/validation_reports/i6/4i6s ftp://data.pdbj.org/pub/pdb/validation_reports/i6/4i6s ftp://data.pdbj.org/pub/pdb/validation_reports/i6/4i6s | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||
| Components on special symmetry positions |
| ||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
-タンパク質 , 1種, 3分子 ABC
| #1: タンパク質 | 分子量: 9749.626 Da / 分子数: 3 / 変異: W76A / 由来タイプ: 組換発現 由来: (組換発現)   Ralstonia solanacearum (青枯病菌) Ralstonia solanacearum (青枯病菌)株: ATCC 11696 / 遺伝子: CMR15_11270, rsc2107 / プラスミド: pET25 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): Tuner / 参照: UniProt: D8NA05 Escherichia coli (大腸菌) / 株 (発現宿主): Tuner / 参照: UniProt: D8NA05 |
|---|
-糖 , 2種, 9分子 
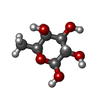

| #2: 糖 | ChemComp-FUC /  フコース フコース#3: 糖 | ChemComp-FUL /  フコース フコース |
|---|
-非ポリマー , 3種, 288分子 




| #4: 化合物 | ChemComp-EDO /  エチレングリコール エチレングリコール | ||
|---|---|---|---|
| #5: 化合物 |  ポリエチレングリコール ポリエチレングリコール#6: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 非ポリマーの詳細 | HETEROGEN FUC(102 CHAIN A) AND FUL(103 CHAIN A) ARE IN ALTERNATE CONFORMATIONS OF EACH OTHER. ...HETEROGEN FUC(102 CHAIN A) AND FUL(103 CHAIN A) ARE IN ALTERNATE CONFORMATI |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.32 Å3/Da / 溶媒含有率: 46.98 % |
|---|---|
結晶化 | 温度: 292 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 詳細: 0.1M Tris/Bicine, 0.12M Monosaccharides, 30% P550MME/P20K, pH 8.5, VAPOR DIFFUSION, HANGING DROP, temperature 292.0K |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SOLEIL SOLEIL  / ビームライン: PROXIMA 1 / 波長: 0.9817 Å / ビームライン: PROXIMA 1 / 波長: 0.9817 Å | |||||||||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2012年5月4日 詳細: KIRKPATRICK-BAEZ PAIR OF BI-MORPH MIRRORS PLUS CHANNEL CUT CRYOGENICALLY COOLED MONOCHROMATOR CRYSTAL | |||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||
| 放射波長 | 波長 : 0.9817 Å / 相対比: 1 : 0.9817 Å / 相対比: 1 | |||||||||||||||||||||
| 反射 | 解像度: 1.54→51.88 Å / Num. obs: 41015 / % possible obs: 99.8 % / Observed criterion σ(F): 2 / Observed criterion σ(I): 2 / 冗長度: 9 % / Biso Wilson estimate: 20 Å2 / Rmerge(I) obs: 0.042 / Rsym value: 0.042 / Net I/σ(I): 24.5 | |||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 2BT9 解像度: 1.54→51.88 Å / Cor.coef. Fo:Fc: 0.973 / Cor.coef. Fo:Fc free: 0.969 / SU B: 2.104 / SU ML: 0.041 / 交差検証法: THROUGHOUT / σ(F): 2 / σ(I): 2 / ESU R: 0.067 / ESU R Free: 0.067 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 24.716 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.54→51.88 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.54→1.576 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




