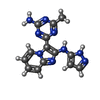
登録情報 データベース : PDB / ID : 4fhjタイトル Crystal Structure of PI3K-gamma in Complex with Imidazopyridine 2 Phosphatidylinositol 4,5-bisphosphate 3-kinase catalytic subunit gamma isoform キーワード / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 2.6 Å データ登録者 Shaffer, P.L. / Tang, J. / Yakowec, P. ジャーナル : Bioorg.Med.Chem.Lett. / 年 : 2012タイトル : Discovery and optimization of potent and selective imidazopyridine and imidazopyridazine mTOR inhibitors.著者: Peterson, E.A. / Boezio, A.A. / Andrews, P.S. / Boezio, C.M. / Bush, T.L. / Cheng, A.C. / Choquette, D. / Coats, J.R. / Colletti, A.E. / Copeland, K.W. / Dupont, M. / Graceffa, R. / ... 著者 : Peterson, E.A. / Boezio, A.A. / Andrews, P.S. / Boezio, C.M. / Bush, T.L. / Cheng, A.C. / Choquette, D. / Coats, J.R. / Colletti, A.E. / Copeland, K.W. / Dupont, M. / Graceffa, R. / Grubinska, B. / Kim, J.L. / Lewis, R.T. / Liu, J. / Mullady, E.L. / Potashman, M.H. / Romero, K. / Shaffer, P.L. / Stanton, M.K. / Stellwagen, J.C. / Teffera, Y. / Yi, S. / Cai, T. / La, D.S. 履歴 登録 2012年6月6日 登録サイト / 処理サイト 改定 1.0 2012年7月18日 Provider / タイプ 改定 1.1 2012年8月29日 Group 改定 1.2 2017年11月15日 Group / カテゴリ / Item 改定 1.3 2023年9月13日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ref_seq_dif / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード inhibitor (酵素阻害剤) / p110 /
inhibitor (酵素阻害剤) / p110 /  kinase (キナーゼ) /
kinase (キナーゼ) /  transferase (転移酵素) / ATP-binding / p84 / p101 / Transferase-Inhibitor complex
transferase (転移酵素) / ATP-binding / p84 / p101 / Transferase-Inhibitor complex 機能・相同性情報
機能・相同性情報 phosphatidylinositol-4-phosphate 3-kinase / regulation of calcium ion transmembrane transport / positive regulation of acute inflammatory response / respiratory burst involved in defense response / negative regulation of cardiac muscle contraction / T cell chemotaxis ...secretory granule localization / negative regulation of triglyceride catabolic process / natural killer cell chemotaxis / neutrophil extravasation /
phosphatidylinositol-4-phosphate 3-kinase / regulation of calcium ion transmembrane transport / positive regulation of acute inflammatory response / respiratory burst involved in defense response / negative regulation of cardiac muscle contraction / T cell chemotaxis ...secretory granule localization / negative regulation of triglyceride catabolic process / natural killer cell chemotaxis / neutrophil extravasation /  phosphatidylinositol-4-phosphate 3-kinase / regulation of calcium ion transmembrane transport / positive regulation of acute inflammatory response / respiratory burst involved in defense response / negative regulation of cardiac muscle contraction / T cell chemotaxis / negative regulation of fibroblast apoptotic process / phosphatidylinositol 3-kinase complex, class IB / sphingosine-1-phosphate receptor signaling pathway / dendritic cell chemotaxis / 1-phosphatidylinositol-4-phosphate 3-kinase activity / 1-phosphatidylinositol-4,5-bisphosphate 3-kinase activity /
phosphatidylinositol-4-phosphate 3-kinase / regulation of calcium ion transmembrane transport / positive regulation of acute inflammatory response / respiratory burst involved in defense response / negative regulation of cardiac muscle contraction / T cell chemotaxis / negative regulation of fibroblast apoptotic process / phosphatidylinositol 3-kinase complex, class IB / sphingosine-1-phosphate receptor signaling pathway / dendritic cell chemotaxis / 1-phosphatidylinositol-4-phosphate 3-kinase activity / 1-phosphatidylinositol-4,5-bisphosphate 3-kinase activity /  phosphatidylinositol-4,5-bisphosphate 3-kinase / phosphatidylinositol 3-kinase complex, class IA /
phosphatidylinositol-4,5-bisphosphate 3-kinase / phosphatidylinositol 3-kinase complex, class IA /  PI3キナーゼ / phosphatidylinositol-3-phosphate biosynthetic process / 1-phosphatidylinositol-3-kinase activity / mast cell degranulation / Erythropoietin activates Phosphoinositide-3-kinase (PI3K) / hepatocyte apoptotic process / positive regulation of Rac protein signal transduction / regulation of cell adhesion mediated by integrin / Synthesis of PIPs at the plasma membrane / phosphatidylinositol phosphate biosynthetic process /
PI3キナーゼ / phosphatidylinositol-3-phosphate biosynthetic process / 1-phosphatidylinositol-3-kinase activity / mast cell degranulation / Erythropoietin activates Phosphoinositide-3-kinase (PI3K) / hepatocyte apoptotic process / positive regulation of Rac protein signal transduction / regulation of cell adhesion mediated by integrin / Synthesis of PIPs at the plasma membrane / phosphatidylinositol phosphate biosynthetic process /  regulation of angiogenesis / T cell proliferation / cellular response to cAMP / GPVI-mediated activation cascade /
regulation of angiogenesis / T cell proliferation / cellular response to cAMP / GPVI-mediated activation cascade /  ephrin receptor binding /
ephrin receptor binding /  T細胞 /
T細胞 /  好中球 / positive regulation of endothelial cell migration / phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of cytokine production / positive regulation of MAP kinase activity /
好中球 / positive regulation of endothelial cell migration / phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of cytokine production / positive regulation of MAP kinase activity /  血小板 /
血小板 /  エンドサイトーシス / G beta:gamma signalling through PI3Kgamma / Signaling by CSF1 (M-CSF) in myeloid cells /
エンドサイトーシス / G beta:gamma signalling through PI3Kgamma / Signaling by CSF1 (M-CSF) in myeloid cells /  kinase activity / positive regulation of cytosolic calcium ion concentration /
kinase activity / positive regulation of cytosolic calcium ion concentration /  血管新生 /
血管新生 /  獲得免疫系 / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /
獲得免疫系 / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  non-specific serine/threonine protein kinase /
non-specific serine/threonine protein kinase /  protein kinase activity /
protein kinase activity /  免疫応答 /
免疫応答 /  炎症 / G protein-coupled receptor signaling pathway /
炎症 / G protein-coupled receptor signaling pathway /  リン酸化 / protein serine kinase activity /
リン酸化 / protein serine kinase activity /  自然免疫系 / protein serine/threonine kinase activity /
自然免疫系 / protein serine/threonine kinase activity /  ATP binding /
ATP binding /  生体膜 / identical protein binding /
生体膜 / identical protein binding /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.6 Å
分子置換 / 解像度: 2.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2012
ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2012 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4fhj.cif.gz
4fhj.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4fhj.ent.gz
pdb4fhj.ent.gz PDB形式
PDB形式 4fhj.json.gz
4fhj.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/fh/4fhj
https://data.pdbj.org/pub/pdb/validation_reports/fh/4fhj ftp://data.pdbj.org/pub/pdb/validation_reports/fh/4fhj
ftp://data.pdbj.org/pub/pdb/validation_reports/fh/4fhj リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: PIK3CG
Homo sapiens (ヒト) / 遺伝子: PIK3CG
 Spodoptera frugiperda (ツマジロクサヨトウ)
Spodoptera frugiperda (ツマジロクサヨトウ) phosphatidylinositol-4,5-bisphosphate 3-kinase,
phosphatidylinositol-4,5-bisphosphate 3-kinase,  non-specific serine/threonine protein kinase
non-specific serine/threonine protein kinase 硫酸塩
硫酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.2 / 波長: 1 Å
/ ビームライン: 5.0.2 / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj