登録情報 データベース : PDB / ID : 4cffタイトル Structure of full length human AMPK in complex with a small molecule activator, a thienopyridone derivative (A-769662) (5'-AMP-ACTIVATED PROTEIN KINASE SUBUNIT ...) x 2 5'-AMP-ACTIVATED PROTEIN KINASE CATALYTIC SUBUNIT ALPHA-2 キーワード / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 HOMO SAPIENS (ヒト)手法 / / / 解像度 : 3.924 Å データ登録者 Xiao, B. / Sanders, M.J. / Carmena, D. / Bright, N.J. / Haire, L.F. / Underwood, E. / Patel, B.R. / Heath, R.B. / Walker, P.A. / Hallen, S. ...Xiao, B. / Sanders, M.J. / Carmena, D. / Bright, N.J. / Haire, L.F. / Underwood, E. / Patel, B.R. / Heath, R.B. / Walker, P.A. / Hallen, S. / Giordanetto, F. / Martin, S.R. / Carling, D. / Gamblin, S.J. ジャーナル : Nat.Commun. / 年 : 2013タイトル : Structural Basis of Ampk Regulation by Small Molecule Activators.著者 : Xiao, B. / Sanders, M.J. / Carmena, D. / Bright, N.J. / Haire, L.F. / Underwood, E. / Patel, B.R. / Heath, R.B. / Walker, P.A. / Hallen, S. / Giordanetto, F. / Martin, S.R. / Carling, D. / Gamblin, S.J. 履歴 登録 2013年11月14日 登録サイト / 処理サイト 改定 1.0 2013年12月25日 Provider / タイプ 改定 1.1 2014年1月8日 Group 改定 1.2 2014年2月12日 Group / Other / Refinement description改定 1.3 2019年5月8日 Group Data collection / Derived calculations ... Data collection / Derived calculations / Experimental preparation / Other カテゴリ database_PDB_rev / database_PDB_rev_record ... database_PDB_rev / database_PDB_rev_record / exptl_crystal_grow / pdbx_database_proc / pdbx_database_status / struct_conn Item / _pdbx_database_status.recvd_author_approval / _struct_conn.pdbx_leaving_atom_flag改定 1.4 2023年12月20日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Other / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_initial_refinement_model / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSFERASE (転移酵素) / NUCLEOTIDE-BINDING / STAUROSPORINE-BINDING /
TRANSFERASE (転移酵素) / NUCLEOTIDE-BINDING / STAUROSPORINE-BINDING /  SERINE/THREONINE-PROTEIN KINASE / ACTIVATOR / CARBOHYDRATE BINDING MODULE (CBM)
SERINE/THREONINE-PROTEIN KINASE / ACTIVATOR / CARBOHYDRATE BINDING MODULE (CBM) 機能・相同性情報
機能・相同性情報 regulation of stress granule assembly / histone H2BS36 kinase activity / AMPK inhibits chREBP transcriptional activation activity / positive regulation of peptidyl-lysine acetylation / lipid droplet disassembly / Lipophagy ...[hydroxymethylglutaryl-CoA reductase (NADPH)] kinase / nail development / [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase activity / : /
regulation of stress granule assembly / histone H2BS36 kinase activity / AMPK inhibits chREBP transcriptional activation activity / positive regulation of peptidyl-lysine acetylation / lipid droplet disassembly / Lipophagy ...[hydroxymethylglutaryl-CoA reductase (NADPH)] kinase / nail development / [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase activity / : /  regulation of stress granule assembly / histone H2BS36 kinase activity / AMPK inhibits chREBP transcriptional activation activity / positive regulation of peptidyl-lysine acetylation / lipid droplet disassembly / Lipophagy / import into nucleus /
regulation of stress granule assembly / histone H2BS36 kinase activity / AMPK inhibits chREBP transcriptional activation activity / positive regulation of peptidyl-lysine acetylation / lipid droplet disassembly / Lipophagy / import into nucleus /  cAMP-dependent protein kinase regulator activity / protein kinase regulator activity / Energy dependent regulation of mTOR by LKB1-AMPK / negative regulation of TOR signaling / Carnitine metabolism / negative regulation of hepatocyte apoptotic process / nucleotide-activated protein kinase complex / protein localization to lipid droplet / response to muscle activity / Activation of PPARGC1A (PGC-1alpha) by phosphorylation / Nuclear events mediated by NFE2L2 / regulation of glycolytic process / lipid biosynthetic process /
cAMP-dependent protein kinase regulator activity / protein kinase regulator activity / Energy dependent regulation of mTOR by LKB1-AMPK / negative regulation of TOR signaling / Carnitine metabolism / negative regulation of hepatocyte apoptotic process / nucleotide-activated protein kinase complex / protein localization to lipid droplet / response to muscle activity / Activation of PPARGC1A (PGC-1alpha) by phosphorylation / Nuclear events mediated by NFE2L2 / regulation of glycolytic process / lipid biosynthetic process /  cAMP-dependent protein kinase activity / negative regulation of tubulin deacetylation /
cAMP-dependent protein kinase activity / negative regulation of tubulin deacetylation /  オートファジー / AMP-activated protein kinase activity / positive regulation of protein localization / cholesterol biosynthetic process / AMP binding / cellular response to nutrient levels / positive regulation of macroautophagy /
オートファジー / AMP-activated protein kinase activity / positive regulation of protein localization / cholesterol biosynthetic process / AMP binding / cellular response to nutrient levels / positive regulation of macroautophagy /  regulation of macroautophagy / positive regulation of protein kinase activity / cellular response to glucose starvation / fatty acid homeostasis / positive regulation of autophagy / Activation of AMPK downstream of NMDARs / regulation of microtubule cytoskeleton organization /
regulation of macroautophagy / positive regulation of protein kinase activity / cellular response to glucose starvation / fatty acid homeostasis / positive regulation of autophagy / Activation of AMPK downstream of NMDARs / regulation of microtubule cytoskeleton organization /  energy homeostasis / negative regulation of TORC1 signaling / cellular response to calcium ion / protein serine/threonine/tyrosine kinase activity / positive regulation of glycolytic process / Translocation of SLC2A4 (GLUT4) to the plasma membrane /
energy homeostasis / negative regulation of TORC1 signaling / cellular response to calcium ion / protein serine/threonine/tyrosine kinase activity / positive regulation of glycolytic process / Translocation of SLC2A4 (GLUT4) to the plasma membrane /  ADP binding / cellular response to glucose stimulus / TP53 Regulates Metabolic Genes /
ADP binding / cellular response to glucose stimulus / TP53 Regulates Metabolic Genes /  regulation of circadian rhythm /
regulation of circadian rhythm /  Wntシグナル経路 /
Wntシグナル経路 /  オートファジー / cytoplasmic stress granule / cellular response to prostaglandin E stimulus / fatty acid biosynthetic process / rhythmic process / cellular response to xenobiotic stimulus /
オートファジー / cytoplasmic stress granule / cellular response to prostaglandin E stimulus / fatty acid biosynthetic process / rhythmic process / cellular response to xenobiotic stimulus /  glucose homeostasis / cellular response to oxidative stress / positive regulation of cold-induced thermogenesis /
glucose homeostasis / cellular response to oxidative stress / positive regulation of cold-induced thermogenesis /  精子形成 / Regulation of TP53 Activity through Phosphorylation /
精子形成 / Regulation of TP53 Activity through Phosphorylation /  non-specific serine/threonine protein kinase /
non-specific serine/threonine protein kinase /  protein kinase activity / intracellular signal transduction / nuclear speck /
protein kinase activity / intracellular signal transduction / nuclear speck /  神経繊維 /
神経繊維 /  protein phosphorylation / negative regulation of gene expression / protein serine kinase activity / protein serine/threonine kinase activity / neuronal cell body /
protein phosphorylation / negative regulation of gene expression / protein serine kinase activity / protein serine/threonine kinase activity / neuronal cell body /  樹状突起 /
樹状突起 /  chromatin binding / positive regulation of gene expression / negative regulation of apoptotic process /
chromatin binding / positive regulation of gene expression / negative regulation of apoptotic process /  protein kinase binding /
protein kinase binding /  ゴルジ体 /
ゴルジ体 /  シグナル伝達 /
シグナル伝達 /  核質 /
核質 /  ATP binding /
ATP binding /  生体膜 /
生体膜 /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3.924 Å
分子置換 / 解像度: 3.924 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Commun. / 年: 2013
ジャーナル: Nat.Commun. / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4cff.cif.gz
4cff.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4cff.ent.gz
pdb4cff.ent.gz PDB形式
PDB形式 4cff.json.gz
4cff.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/cf/4cff
https://data.pdbj.org/pub/pdb/validation_reports/cf/4cff ftp://data.pdbj.org/pub/pdb/validation_reports/cf/4cff
ftp://data.pdbj.org/pub/pdb/validation_reports/cf/4cff リンク
リンク 集合体
集合体


 要素
要素
 HOMO SAPIENS (ヒト) / 発現宿主:
HOMO SAPIENS (ヒト) / 発現宿主: 
 ESCHERICHIA COLI (大腸菌)
ESCHERICHIA COLI (大腸菌) non-specific serine/threonine protein kinase, [acetyl-CoA carboxylase] kinase, [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase
non-specific serine/threonine protein kinase, [acetyl-CoA carboxylase] kinase, [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase
 HOMO SAPIENS (ヒト) / 発現宿主:
HOMO SAPIENS (ヒト) / 発現宿主: 
 ESCHERICHIA COLI (大腸菌) / 参照: UniProt: Q9Y478
ESCHERICHIA COLI (大腸菌) / 参照: UniProt: Q9Y478
 HOMO SAPIENS (ヒト) / 発現宿主:
HOMO SAPIENS (ヒト) / 発現宿主: 
 ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P54619
ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P54619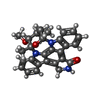
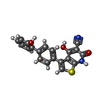



 スタウロスポリン
スタウロスポリン アデニル酸
アデニル酸 X線回折
X線回折 試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I04-1 / 波長: 0.92
/ ビームライン: I04-1 / 波長: 0.92  : 0.92 Å / 相対比: 1
: 0.92 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj













