+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4bzj | ||||||
|---|---|---|---|---|---|---|---|
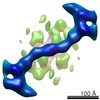
| タイトル | The structure of the COPII coat assembled on membranes | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  PROTEIN TRANSPORT / PROTEIN TRANSPORT /  SECRETION (分泌) / TRAFFICKING SECRETION (分泌) / TRAFFICKING | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Seh1-associated complex / positive regulation of ER to Golgi vesicle-mediated transport / protein exit from endoplasmic reticulum / COPII-coated vesicle budding / COPII-mediated vesicle transport / nuclear pore localization / regulation of TORC1 signaling / COPII-coated vesicle cargo loading / nuclear pore outer ring / COPII vesicle coat ...Seh1-associated complex / positive regulation of ER to Golgi vesicle-mediated transport / protein exit from endoplasmic reticulum / COPII-coated vesicle budding / COPII-mediated vesicle transport / nuclear pore localization / regulation of TORC1 signaling / COPII-coated vesicle cargo loading / nuclear pore outer ring / COPII vesicle coat / positive regulation of protein exit from endoplasmic reticulum / mating projection tip / endoplasmic reticulum organization / nucleocytoplasmic transport / vacuolar membrane / positive regulation of TOR signaling / mRNA transport / endoplasmic reticulum exit site /  核膜孔 / 核膜孔 /  : / positive regulation of TORC1 signaling / cell periphery / : / positive regulation of TORC1 signaling / cell periphery /  intracellular protein transport / protein import into nucleus / intracellular protein transport / protein import into nucleus /  核膜 / endoplasmic reticulum membrane / structural molecule activity / positive regulation of DNA-templated transcription / 核膜 / endoplasmic reticulum membrane / structural molecule activity / positive regulation of DNA-templated transcription /  小胞体 小胞体類似検索 - 分子機能 | ||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||
| 手法 |  電子顕微鏡法 / サブトモグラム平均法 / 電子顕微鏡法 / サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 40 Å クライオ電子顕微鏡法 / 解像度: 40 Å | ||||||
 データ登録者 データ登録者 | Zanetti, G. / Prinz, S. / Daum, S. / Meister, A. / Schekman, R. / Bacia, K. / Briggs, J.A.G. | ||||||
 引用 引用 |  ジャーナル: Elife / 年: 2013 ジャーナル: Elife / 年: 2013タイトル: The structure of the COPII transport-vesicle coat assembled on membranes. 著者: Giulia Zanetti / Simone Prinz / Sebastian Daum / Annette Meister / Randy Schekman / Kirsten Bacia / John A G Briggs /  要旨: Coat protein complex II (COPII) mediates formation of the membrane vesicles that export newly synthesised proteins from the endoplasmic reticulum. The inner COPII proteins bind to cargo and membrane, ...Coat protein complex II (COPII) mediates formation of the membrane vesicles that export newly synthesised proteins from the endoplasmic reticulum. The inner COPII proteins bind to cargo and membrane, linking them to the outer COPII components that form a cage around the vesicle. Regulated flexibility in coat architecture is essential for transport of a variety of differently sized cargoes, but structural data on the assembled coat has not been available. We have used cryo-electron tomography and subtomogram averaging to determine the structure of the complete, membrane-assembled COPII coat. We describe a novel arrangement of the outer coat and find that the inner coat can assemble into regular lattices. The data reveal how coat subunits interact with one another and with the membrane, suggesting how coordinated assembly of inner and outer coats can mediate and regulate packaging of vesicles ranging from small spheres to large tubular carriers. DOI:http://dx.doi.org/10.7554/eLife.00951.001. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4bzj.cif.gz 4bzj.cif.gz | 406.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4bzj.ent.gz pdb4bzj.ent.gz | 324.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4bzj.json.gz 4bzj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bz/4bzj https://data.pdbj.org/pub/pdb/validation_reports/bz/4bzj ftp://data.pdbj.org/pub/pdb/validation_reports/bz/4bzj ftp://data.pdbj.org/pub/pdb/validation_reports/bz/4bzj | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  Protein targeting / Protein WEB1 Protein targeting / Protein WEB1分子量: 138833.422 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: SEC31, WEB1, YDL195W, D1229 / プラスミド: PNS3141 (6H31/CK1313) / 発現宿主:   SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): RSY1112 / 参照: UniProt: P38968 SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): RSY1112 / 参照: UniProt: P38968#2: タンパク質 | |  Protein targeting Protein targeting分子量: 32400.205 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: SEC13, ANU3, YLR208W, L8167.4 / プラスミド: PNS3141 (6H31/CK1313) / 発現宿主:   SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): RSY1112 / 参照: UniProt: Q04491 SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): RSY1112 / 参照: UniProt: Q04491#3: タンパク質 | |  Protein targeting Protein targeting分子量: 32508.428 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: SEC13, ANU3, YLR208W, L8167.4 / プラスミド: PNS3141 (6H31/CK1313) / 発現宿主:   SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): RSY1112 / 参照: UniProt: Q04491 SACCHAROMYCES CEREVISIAE (パン酵母) / 株 (発現宿主): RSY1112 / 参照: UniProt: Q04491#4: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: サブトモグラム平均法 |
- 試料調製
試料調製
| 構成要素 | 名称: COPII coat assembled on membranes / タイプ: COMPLEX |
|---|---|
| 緩衝液 | 名称: HEPES, 50 MM KOAC, 1.2 MM MGCL2 / pH: 6.8 / 詳細: HEPES, 50 MM KOAC, 1.2 MM MGCL2 / pH: 6.8 / 詳細: HEPES, 50 MM KOAC, 1.2 MM MGCL2 |
| 試料 | 濃度: 0.03 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | 詳細: C-FLAT GRIDS / グリッドのタイプ: C-flat |
急速凍結 | 装置: HOMEMADE PLUNGER / 凍結剤: ETHANE / 詳細: ETHANE; HOMEMADE PLUNGER |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS / 日付: 2012年9月18日 |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 19500 X / 最大 デフォーカス(公称値): 3200 nm / 最小 デフォーカス(公称値): 2000 nm / Cs Bright-field microscopy / 倍率(公称値): 19500 X / 最大 デフォーカス(公称値): 3200 nm / 最小 デフォーカス(公称値): 2000 nm / Cs : 2.7 mm : 2.7 mm |
| 試料ホルダ | 傾斜角・最大: 60 ° / 傾斜角・最小: -60 ° |
| 撮影 | 電子線照射量: 80 e/Å2 / フィルム・検出器のモデル: GATAN MULTISCAN |
| 画像スキャン | デジタル画像の数: 26 |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | 詳細: EACH TILTED IMAGE WITHIN TOMOGRAM | |||||||||||||||
| 対称性 | 点対称性 : C2 (2回回転対称 : C2 (2回回転対称 ) ) | |||||||||||||||
3次元再構成 | 手法: SUBTOMOGRAM ALIGNMENT AND AVERAGING / 解像度: 40 Å / 粒子像の数: 182 / ピクセルサイズ(公称値): 4.3 Å / ピクセルサイズ(実測値): 4.3 Å 詳細: SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-2430. (DEPOSITION ID: 11862). 対称性のタイプ: POINT | |||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL / Target criteria: Cross-correlation coefficient / 詳細: METHOD--RIGID BODY REFINEMENT PROTOCOL--X-RAY | |||||||||||||||
| 原子モデル構築 |
| |||||||||||||||
| 精密化 | 最高解像度: 40 Å | |||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 40 Å
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj



