+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3obs | ||||||
|---|---|---|---|---|---|---|---|
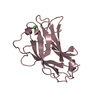
| タイトル | Crystal structure of Tsg101 UEV domain | ||||||
 要素 要素 | Tumor susceptibility gene 101 protein TSG101 TSG101 | ||||||
 キーワード キーワード |  PROTEIN TRANSPORT / Protein Transprot / PROTEIN TRANSPORT / Protein Transprot /  Ubiquitin Binding (ユビキチン) Ubiquitin Binding (ユビキチン) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of viral budding via host ESCRT complex / positive regulation of ubiquitin-dependent endocytosis / extracellular transport /  ESCRT I complex / negative regulation of epidermal growth factor-activated receptor activity / regulation of extracellular exosome assembly / ESCRT I complex / negative regulation of epidermal growth factor-activated receptor activity / regulation of extracellular exosome assembly /  ウイルス排出 / ウイルス排出 /  regulation of MAP kinase activity / exosomal secretion / protein transport to vacuole involved in ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway ...positive regulation of viral budding via host ESCRT complex / positive regulation of ubiquitin-dependent endocytosis / extracellular transport / regulation of MAP kinase activity / exosomal secretion / protein transport to vacuole involved in ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway ...positive regulation of viral budding via host ESCRT complex / positive regulation of ubiquitin-dependent endocytosis / extracellular transport /  ESCRT I complex / negative regulation of epidermal growth factor-activated receptor activity / regulation of extracellular exosome assembly / ESCRT I complex / negative regulation of epidermal growth factor-activated receptor activity / regulation of extracellular exosome assembly /  ウイルス排出 / ウイルス排出 /  regulation of MAP kinase activity / exosomal secretion / protein transport to vacuole involved in ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway / membrane fission / positive regulation of exosomal secretion / ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway / regulation of MAP kinase activity / exosomal secretion / protein transport to vacuole involved in ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway / membrane fission / positive regulation of exosomal secretion / ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway /  multivesicular body assembly / Flemming body / multivesicular body assembly / Flemming body /  virion binding / endosome to lysosome transport / negative regulation of epidermal growth factor receptor signaling pathway / viral budding via host ESCRT complex / autophagosome maturation / viral release from host cell / keratinocyte differentiation / Membrane binding and targetting of GAG proteins / Endosomal Sorting Complex Required For Transport (ESCRT) / virion binding / endosome to lysosome transport / negative regulation of epidermal growth factor receptor signaling pathway / viral budding via host ESCRT complex / autophagosome maturation / viral release from host cell / keratinocyte differentiation / Membrane binding and targetting of GAG proteins / Endosomal Sorting Complex Required For Transport (ESCRT) /  エンドソーム / HCMV Late Events / エンドソーム / HCMV Late Events /  ubiquitin binding / ubiquitin binding /  regulation of cell growth / regulation of cell growth /  オートファジー / Late endosomal microautophagy / protein modification process / Budding and maturation of HIV virion / calcium-dependent protein binding / transcription corepressor activity / late endosome / late endosome membrane / early endosome membrane / オートファジー / Late endosomal microautophagy / protein modification process / Budding and maturation of HIV virion / calcium-dependent protein binding / transcription corepressor activity / late endosome / late endosome membrane / early endosome membrane /  エンドソーム / endosome membrane / エンドソーム / endosome membrane /  regulation of cell cycle / regulation of cell cycle /  エンドソーム / エンドソーム /  細胞周期 / 細胞周期 /  細胞分裂 / negative regulation of cell population proliferation / 細胞分裂 / negative regulation of cell population proliferation /  中心体 / 中心体 /  ubiquitin protein ligase binding / protein-containing complex binding / ubiquitin protein ligase binding / protein-containing complex binding /  核小体 / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / 核小体 / negative regulation of transcription by RNA polymerase II / protein homodimerization activity /  DNA binding / extracellular exosome / DNA binding / extracellular exosome /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.5 Å 分子置換 / 解像度: 1.5 Å | ||||||
 データ登録者 データ登録者 | Im, Y.J. / Hurley, J.H. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2010 ジャーナル: Structure / 年: 2010タイトル: Crystallographic and Functional Analysis of the ESCRT-I /HIV-1 Gag PTAP Interaction. 著者: Im, Y.J. / Kuo, L. / Ren, X. / Burgos, P.V. / Zhao, X.Z. / Liu, F. / Burke, T.R. / Bonifacino, J.S. / Freed, E.O. / Hurley, J.H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3obs.cif.gz 3obs.cif.gz | 46.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3obs.ent.gz pdb3obs.ent.gz | 31 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3obs.json.gz 3obs.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ob/3obs https://data.pdbj.org/pub/pdb/validation_reports/ob/3obs ftp://data.pdbj.org/pub/pdb/validation_reports/ob/3obs ftp://data.pdbj.org/pub/pdb/validation_reports/ob/3obs | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 |  TSG101 / ESCRT-I complex subunit TSG101 TSG101 / ESCRT-I complex subunit TSG101分子量: 16501.195 Da / 分子数: 1 / 断片: N-terminal UEV domain (UNP residues 2 to 145) / 変異: 43VFNDGS48 -> GTG / 由来タイプ: 組換発現 詳細: to improve the crystallization properies, residues 43-49 in the beta1-beta2 loop were mutated to Gly-Thr-Gly 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: TSG101, Tsg101 / プラスミド: pGST2 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TSG101, Tsg101 / プラスミド: pGST2 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) Star / 参照: UniProt: Q99816 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) Star / 参照: UniProt: Q99816 |
|---|---|
| #2: 水 | ChemComp-HOH /  水 水 |
| 配列の詳細 | ENGINEERED |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.21 Å3/Da / 溶媒含有率: 44.26 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 0.1M HEPES-NaOH (pH 7.5), 25% PEG 3350, 0.2M Sodium Nitrate , VAPOR DIFFUSION, HANGING DROP, temperature 298K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 22-ID / 波長: 1 Å / ビームライン: 22-ID / 波長: 1 Å |
| 検出器 | タイプ: MARMOSAIC 300 mm CCD / 検出器: CCD / 日付: 2009年12月9日 / 詳細: mirrors |
| 放射 | モノクロメーター: Si 111 CHANNEL / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 1.5→50 Å / Num. all: 22608 / Num. obs: 22570 / % possible obs: 97.1 % / Observed criterion σ(F): 3 / Observed criterion σ(I): 3 / 冗長度: 3.6 % / Biso Wilson estimate: 12.4 Å2 / Rmerge(I) obs: 0.039 / Net I/σ(I): 47.5 |
| 反射 シェル | 解像度: 1.5→1.53 Å / 冗長度: 3.7 % / Rmerge(I) obs: 0.092 / Mean I/σ(I) obs: 25.3 / Num. unique all: 1130 / % possible all: 96.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 2F0R 解像度: 1.5→32.92 Å / Rfactor Rfree error: 0.007 / Data cutoff high absF: 600106.44 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 39.2413 Å2 / ksol: 0.381544 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 15.2 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.5→32.92 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: NONE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.5→1.59 Å / Rfactor Rfree error: 0.017 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj





