+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3lo2 | ||||||
|---|---|---|---|---|---|---|---|
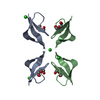
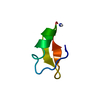
| タイトル | Crystal structure of human alpha-defensin 1 (Y21A mutant) | ||||||
 要素 要素 | Neutrophil defensin 1 | ||||||
 キーワード キーワード |  ANTIMICROBIAL PROTEIN (抗微生物ペプチド) / ANTIMICROBIAL PROTEIN (抗微生物ペプチド) /  ANTIMICROBIAL PEPTIDE (抗微生物ペプチド) / ANTIMICROBIAL PEPTIDE (抗微生物ペプチド) /  HUMAN ALPHA DEFENSIN 1 / HUMAN NEUTROPHIL PEPTIDE 1 / HUMAN ALPHA DEFENSIN 1 / HUMAN NEUTROPHIL PEPTIDE 1 /  HNP1 / HNP1 /  ANTIBIOTIC (抗生物質) / ANTIBIOTIC (抗生物質) /  ANTIMICROBIAL / Antiviral defense / ANTIMICROBIAL / Antiviral defense /  Defensin (ディフェンシン) / Defensin (ディフェンシン) /  Disulfide bond (ジスルフィド) / Disulfide bond (ジスルフィド) /  Fungicide (殺菌剤 (農薬その他)) / Fungicide (殺菌剤 (農薬その他)) /  Phosphoprotein / Phosphoprotein /  Secreted (分泌) Secreted (分泌) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pore-forming activity /  ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus / ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus /  innate immune response in mucosa ...pore-forming activity / innate immune response in mucosa ...pore-forming activity /  ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus / ディフェンシン / disruption of plasma membrane integrity in another organism / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / intracellular estrogen receptor signaling pathway / defense response to fungus /  innate immune response in mucosa / Golgi lumen / innate immune response in mucosa / Golgi lumen /  走化性 / azurophil granule lumen / antimicrobial humoral immune response mediated by antimicrobial peptide / antibacterial humoral response / collagen-containing extracellular matrix / defense response to virus / killing of cells of another organism / cellular response to lipopolysaccharide / defense response to Gram-negative bacterium / defense response to Gram-positive bacterium / 走化性 / azurophil granule lumen / antimicrobial humoral immune response mediated by antimicrobial peptide / antibacterial humoral response / collagen-containing extracellular matrix / defense response to virus / killing of cells of another organism / cellular response to lipopolysaccharide / defense response to Gram-negative bacterium / defense response to Gram-positive bacterium /  免疫応答 / Neutrophil degranulation / 免疫応答 / Neutrophil degranulation /  extracellular space / extracellular exosome / extracellular region extracellular space / extracellular exosome / extracellular region類似検索 - 分子機能 | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.56 Å 分子置換 / 解像度: 1.56 Å | ||||||
 データ登録者 データ登録者 | Pazgier, M. / Lu, W. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2010 ジャーナル: J.Biol.Chem. / 年: 2010タイトル: Trp-26 imparts functional versatility to human alpha-defensin HNP1. 著者: Wei, G. / Pazgier, M. / de Leeuw, E. / Rajabi, M. / Li, J. / Zou, G. / Jung, G. / Yuan, W. / Lu, W.Y. / Lehrer, R.I. / Lu, W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3lo2.cif.gz 3lo2.cif.gz | 27.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3lo2.ent.gz pdb3lo2.ent.gz | 17.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3lo2.json.gz 3lo2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lo/3lo2 https://data.pdbj.org/pub/pdb/validation_reports/lo/3lo2 ftp://data.pdbj.org/pub/pdb/validation_reports/lo/3lo2 ftp://data.pdbj.org/pub/pdb/validation_reports/lo/3lo2 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||
| 2 | 
| ||||||||||||||||||
| 3 |
| ||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||
| Components on special symmetry positions |
| ||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Ens-ID: 1 / Beg auth comp-ID: ALA / Beg label comp-ID: ALA / End auth comp-ID: CYS / End label comp-ID: CYS / Refine code: 6 / Auth seq-ID: 1 - 30 / Label seq-ID: 1 - 30
| ||||||||||||||||||
| 詳細 | biological unit is half of asymmetric unit |
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 3360.015 Da / 分子数: 2 / 断片: UNP residues 65-94 / 変異: Y21A / 由来タイプ: 合成 / 詳細: Protein is naturally found in HUMAN / 参照: UniProt: P59665 #2: 化合物 |  2-Methyl-2,4-pentanediol 2-Methyl-2,4-pentanediol#3: 化合物 | ChemComp-TFA / |  トリフルオロ酢酸 トリフルオロ酢酸#4: 化合物 | ChemComp-SO4 / |  硫酸塩 硫酸塩#5: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.02 Å3/Da / 溶媒含有率: 39.03 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 30% PEG 8,000; 0.2 M ammonium sulfate, pH pH 8, VAPOR DIFFUSION, HANGING DROP, temperature 298K PH範囲: pH 8 |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.54 Å 回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.54 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: RIGAKU RAXIS IV++ / 検出器: IMAGE PLATE / 日付: 2008年3月18日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 1.54 Å / 相対比: 1 : 1.54 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | 冗長度: 3.1 % / 数: 23252 / Rmerge(I) obs: 0.107 / Χ2: 1.2 / D res high: 1.56 Å / D res low: 50 Å / Num. obs: 7547 / % possible obs: 92 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Diffraction reflection shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.56→41.552 Å / Num. all: 8139 / Num. obs: 7534 / % possible obs: 93 % / Observed criterion σ(I): 2 / 冗長度: 3.1 % / Rmerge(I) obs: 0.107 / Rsym value: 0.157 / Net I/σ(I): 10.3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | 解像度: 1.56→1.59 Å / 冗長度: 2.9 % / Rmerge(I) obs: 0.106 / Mean I/σ(I) obs: 2.9 / Rsym value: 0.11 / % possible all: 80.3 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 3GNY 解像度: 1.56→20 Å / Cor.coef. Fo:Fc: 0.961 / Cor.coef. Fo:Fc free: 0.96 / Occupancy max: 1 / Occupancy min: 0.5 / SU B: 2.754 / SU ML: 0.048 / 交差検証法: THROUGHOUT / ESU R: 0.095 / ESU R Free: 0.091 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: U VALUES : RESIDUAL ONLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: BABINET MODEL WITH MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 13.388 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.56→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Dom-ID: 1 / Auth asym-ID: A / Ens-ID: 1 / 数: 231 / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.562→1.602 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj