| 登録情報 | データベース: PDB / ID: 3gpq
|
|---|
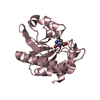
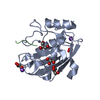
| タイトル | Crystal structure of macro domain of Chikungunya virus in complex with RNA |
|---|
 要素 要素 | - Non-structural protein 3
- RNA (5'-R(*AP*AP*A)-3')
|
|---|
 キーワード キーワード |  VIRAL PROTEIN/RNA (ウイルス性) / VIRAL PROTEIN/RNA (ウイルス性) /  macro domain / X domain / macro domain / X domain /  Chikungunya (チクングニア熱) / Chikungunya (チクングニア熱) /  alphavirus / alphavirus /  virus (ウイルス) / VIZIER. Viral enzymes involved in replication / virus (ウイルス) / VIZIER. Viral enzymes involved in replication /  RNA (リボ核酸) / ATP-binding / RNA (リボ核酸) / ATP-binding /  Cell membrane (細胞膜) / Cell membrane (細胞膜) /  Endosome (エンドソーム) / Endosome (エンドソーム) /  Helicase (ヘリカーゼ) / Helicase (ヘリカーゼ) /  Hydrolase (加水分解酵素) / Hydrolase (加水分解酵素) /  Lipoprotein (リポタンパク質) / Lipoprotein (リポタンパク質) /  Lysosome (リソソーム) / Lysosome (リソソーム) /  Membrane (生体膜) / Membrane (生体膜) /  Methyltransferase (メチルトランスフェラーゼ) / mRNA capping / Methyltransferase (メチルトランスフェラーゼ) / mRNA capping /  mRNA processing (転写後修飾) / Multifunctional enzyme / Nucleotide-binding / mRNA processing (転写後修飾) / Multifunctional enzyme / Nucleotide-binding /  Nucleotidyltransferase / Nucleotidyltransferase /  Nucleus (Nucleus) / Nucleus (Nucleus) /  Palmitate (パルミチン酸) / Palmitate (パルミチン酸) /  Phosphoprotein / Phosphoprotein /  Protease (プロテアーゼ) / Protease (プロテアーゼ) /  RNA replication (RNA依存性RNAポリメラーゼ) / RNA-binding / RNA replication (RNA依存性RNAポリメラーゼ) / RNA-binding /  RNA-directed RNA polymerase (RNA依存性RNAポリメラーゼ) / RNA-directed RNA polymerase (RNA依存性RNAポリメラーゼ) /  Thiol protease (システインプロテアーゼ) / Thiol protease (システインプロテアーゼ) /  Transferase (転移酵素) / Transferase (転移酵素) /  VIRAL PROTEIN-RNA complex (ウイルス性) VIRAL PROTEIN-RNA complex (ウイルス性) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
host cell filopodium /  ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase / ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity / polynucleotide 5'-phosphatase activity /  polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity ...host cell filopodium / polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity ...host cell filopodium /  ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase / ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity / polynucleotide 5'-phosphatase activity /  polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / cysteine-type peptidase activity / polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / cysteine-type peptidase activity /  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / host cell cytoplasmic vesicle membrane / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / host cell cytoplasmic vesicle membrane /  転移酵素; リンを含む基を移すもの; 核酸を移すもの / nucleoside-triphosphate phosphatase / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / nucleoside-triphosphate phosphatase /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ /  RNA helicase activity / RNA helicase activity /  ヘリカーゼ / ヘリカーゼ /  RNA依存性RNAポリメラーゼ / viral RNA genome replication / RNA依存性RNAポリメラーゼ / viral RNA genome replication /  RNA-dependent RNA polymerase activity / DNA-templated transcription / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / GTP binding / host cell plasma membrane / RNA-dependent RNA polymerase activity / DNA-templated transcription / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / GTP binding / host cell plasma membrane /  ATP hydrolysis activity / ATP hydrolysis activity /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  生体膜 / 生体膜 /  metal ion binding類似検索 - 分子機能 metal ion binding類似検索 - 分子機能  : / : /  : / : /  : / Non-structural protein 3, zinc-binding domain / Tomato mosaic virus helicase, N-terminal domain / Alphavirus nsP2 protease domain superfamily / Alphavirus nsp2 protease (nsp2pro) domain / Peptidase family C9 / Alphavirus nsp2 protease (nsp2pro) domain profile. / Viral methyltransferase ... : / Non-structural protein 3, zinc-binding domain / Tomato mosaic virus helicase, N-terminal domain / Alphavirus nsP2 protease domain superfamily / Alphavirus nsp2 protease (nsp2pro) domain / Peptidase family C9 / Alphavirus nsp2 protease (nsp2pro) domain profile. / Viral methyltransferase ... : / : /  : / : /  : / Non-structural protein 3, zinc-binding domain / Tomato mosaic virus helicase, N-terminal domain / Alphavirus nsP2 protease domain superfamily / Alphavirus nsp2 protease (nsp2pro) domain / Peptidase family C9 / Alphavirus nsp2 protease (nsp2pro) domain profile. / Viral methyltransferase / Alphavirus-like methyltransferase (MT) domain / Alphavirus-like methyltransferase (MT) domain profile. / : / Non-structural protein 3, zinc-binding domain / Tomato mosaic virus helicase, N-terminal domain / Alphavirus nsP2 protease domain superfamily / Alphavirus nsp2 protease (nsp2pro) domain / Peptidase family C9 / Alphavirus nsp2 protease (nsp2pro) domain profile. / Viral methyltransferase / Alphavirus-like methyltransferase (MT) domain / Alphavirus-like methyltransferase (MT) domain profile. /  Tymovirus, RNA-dependent RNA polymerase / Tymovirus, RNA-dependent RNA polymerase /  RNA依存性RNAポリメラーゼ / RNA依存性RNAポリメラーゼ /  Leucine Aminopeptidase, subunit E, domain 1 / Leucine Aminopeptidase, subunit E, domain 1 /  Leucine Aminopeptidase, subunit E; domain 1 / Viral (Superfamily 1) RNA helicase / (+) RNA virus helicase core domain / (+)RNA virus helicase core domain profile. / Non-structural protein 3, X-domain-like / Leucine Aminopeptidase, subunit E; domain 1 / Viral (Superfamily 1) RNA helicase / (+) RNA virus helicase core domain / (+)RNA virus helicase core domain profile. / Non-structural protein 3, X-domain-like /  Macro domain / Appr-1"-p processing enzyme / Macro domain / Appr-1"-p processing enzyme /  Macro domain / Macro domain profile. / Macro domain / Macro domain profile. /  Macro domain-like / Macro domain-like /  RNA-directed RNA polymerase, catalytic domain / RdRp of positive ssRNA viruses catalytic domain profile. / S-adenosyl-L-methionine-dependent methyltransferase superfamily / DNA/RNA polymerase superfamily / P-loop containing nucleoside triphosphate hydrolase / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 RNA-directed RNA polymerase, catalytic domain / RdRp of positive ssRNA viruses catalytic domain profile. / S-adenosyl-L-methionine-dependent methyltransferase superfamily / DNA/RNA polymerase superfamily / P-loop containing nucleoside triphosphate hydrolase / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |    Chikungunya virus (チクングニヤウイルス) Chikungunya virus (チクングニヤウイルス) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2 Å シンクロトロン / 解像度: 2 Å |
|---|
 データ登録者 データ登録者 | Malet, H. / Jamal, S. / Coutard, B. / Canard, B. |
|---|
 引用 引用 |  ジャーナル: J.Virol. / 年: 2009 ジャーナル: J.Virol. / 年: 2009
タイトル: The crystal structures of Chikungunya and Venezuelan equine encephalitis virus nsP3 macro domains define a conserved adenosine binding pocket
著者: Malet, H. / Coutard, B. / Jamal, S. / Dutartre, H. / Papageorgiou, N. / Neuvonen, M. / Ahola, T. / Forrester, N. / Gould, E.A. / Lafitte, D. / Ferron, F. / Lescar, J. / Gorbalenya, A.E. / de ...著者: Malet, H. / Coutard, B. / Jamal, S. / Dutartre, H. / Papageorgiou, N. / Neuvonen, M. / Ahola, T. / Forrester, N. / Gould, E.A. / Lafitte, D. / Ferron, F. / Lescar, J. / Gorbalenya, A.E. / de Lamballerie, X. / Canard, B. |
|---|
| 履歴 | | 登録 | 2009年3月23日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2009年7月21日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Advisory / Refinement description / Version format compliance |
|---|
| 改定 1.2 | 2023年9月6日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード VIRAL PROTEIN/RNA (ウイルス性) /
VIRAL PROTEIN/RNA (ウイルス性) /  macro domain / X domain /
macro domain / X domain /  Chikungunya (チクングニア熱) /
Chikungunya (チクングニア熱) /  alphavirus /
alphavirus /  virus (ウイルス) / VIZIER. Viral enzymes involved in replication /
virus (ウイルス) / VIZIER. Viral enzymes involved in replication /  RNA (リボ核酸) / ATP-binding /
RNA (リボ核酸) / ATP-binding /  Cell membrane (細胞膜) /
Cell membrane (細胞膜) /  Endosome (エンドソーム) /
Endosome (エンドソーム) /  Helicase (ヘリカーゼ) /
Helicase (ヘリカーゼ) /  Hydrolase (加水分解酵素) /
Hydrolase (加水分解酵素) /  Lipoprotein (リポタンパク質) /
Lipoprotein (リポタンパク質) /  Lysosome (リソソーム) /
Lysosome (リソソーム) /  Membrane (生体膜) /
Membrane (生体膜) /  Methyltransferase (メチルトランスフェラーゼ) / mRNA capping /
Methyltransferase (メチルトランスフェラーゼ) / mRNA capping /  mRNA processing (転写後修飾) / Multifunctional enzyme / Nucleotide-binding /
mRNA processing (転写後修飾) / Multifunctional enzyme / Nucleotide-binding /  Nucleotidyltransferase /
Nucleotidyltransferase /  Nucleus (Nucleus) /
Nucleus (Nucleus) /  Palmitate (パルミチン酸) /
Palmitate (パルミチン酸) /  Phosphoprotein /
Phosphoprotein /  Protease (プロテアーゼ) /
Protease (プロテアーゼ) /  RNA replication (RNA依存性RNAポリメラーゼ) / RNA-binding /
RNA replication (RNA依存性RNAポリメラーゼ) / RNA-binding /  RNA-directed RNA polymerase (RNA依存性RNAポリメラーゼ) /
RNA-directed RNA polymerase (RNA依存性RNAポリメラーゼ) /  Thiol protease (システインプロテアーゼ) /
Thiol protease (システインプロテアーゼ) /  Transferase (転移酵素) /
Transferase (転移酵素) /  VIRAL PROTEIN-RNA complex (ウイルス性)
VIRAL PROTEIN-RNA complex (ウイルス性) 機能・相同性情報
機能・相同性情報 ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /
ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity /
polynucleotide 5'-phosphatase activity /  polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity ...host cell filopodium /
polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity ...host cell filopodium /  ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /
ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity /
polynucleotide 5'-phosphatase activity /  polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / cysteine-type peptidase activity /
polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / cysteine-type peptidase activity /  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / host cell cytoplasmic vesicle membrane /
転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / host cell cytoplasmic vesicle membrane /  転移酵素; リンを含む基を移すもの; 核酸を移すもの / nucleoside-triphosphate phosphatase /
転移酵素; リンを含む基を移すもの; 核酸を移すもの / nucleoside-triphosphate phosphatase /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ /
加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ /  RNA helicase activity /
RNA helicase activity /  ヘリカーゼ /
ヘリカーゼ /  RNA依存性RNAポリメラーゼ / viral RNA genome replication /
RNA依存性RNAポリメラーゼ / viral RNA genome replication /  RNA-dependent RNA polymerase activity / DNA-templated transcription / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / GTP binding / host cell plasma membrane /
RNA-dependent RNA polymerase activity / DNA-templated transcription / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / GTP binding / host cell plasma membrane /  ATP hydrolysis activity /
ATP hydrolysis activity /  タンパク質分解 /
タンパク質分解 /  RNA binding /
RNA binding /  ATP binding /
ATP binding /  生体膜 /
生体膜 /  metal ion binding
metal ion binding

 Chikungunya virus (チクングニヤウイルス)
Chikungunya virus (チクングニヤウイルス) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2 Å
シンクロトロン / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Virol. / 年: 2009
ジャーナル: J.Virol. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3gpq.cif.gz
3gpq.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3gpq.ent.gz
pdb3gpq.ent.gz PDB形式
PDB形式 3gpq.json.gz
3gpq.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/gp/3gpq
https://data.pdbj.org/pub/pdb/validation_reports/gp/3gpq ftp://data.pdbj.org/pub/pdb/validation_reports/gp/3gpq
ftp://data.pdbj.org/pub/pdb/validation_reports/gp/3gpq リンク
リンク 集合体
集合体




 要素
要素

 Chikungunya virus (チクングニヤウイルス)
Chikungunya virus (チクングニヤウイルス)
 Escherichia coli (大腸菌) / 株 (発現宿主): Rosetta (DE3) pLysS / 参照: UniProt: Q8JUX6
Escherichia coli (大腸菌) / 株 (発現宿主): Rosetta (DE3) pLysS / 参照: UniProt: Q8JUX6 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-2 / 波長: 0.933 Å
/ ビームライン: ID14-2 / 波長: 0.933 Å : 0.933 Å / 相対比: 1
: 0.933 Å / 相対比: 1  解析
解析 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj


