| 登録情報 | データベース: PDB / ID: 2n0z
|
|---|
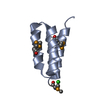
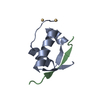
| タイトル | Solution structure of MyUb (1080-1122) of human Myosin VI |
|---|
 要素 要素 | Unconventional myosin-VI |
|---|
 キーワード キーワード |  MOTOR PROTEIN (モータータンパク質) / MOTOR PROTEIN (モータータンパク質) /  PROTEIN TRANSPORT PROTEIN TRANSPORT |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
 regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle ... regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle ... regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle / microfilament motor activity / inner ear morphogenesis / filamentous actin / regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle / microfilament motor activity / inner ear morphogenesis / filamentous actin /  微絨毛 / cytoskeletal motor activity / RHOU GTPase cycle / endocytic vesicle / DNA damage response, signal transduction by p53 class mediator / 微絨毛 / cytoskeletal motor activity / RHOU GTPase cycle / endocytic vesicle / DNA damage response, signal transduction by p53 class mediator /  オートファゴソーム / RHOBTB2 GTPase cycle / オートファゴソーム / RHOBTB2 GTPase cycle /  クラスリン / ruffle / クラスリン / ruffle /  filopodium / actin filament organization / filopodium / actin filament organization /  マイクロフィラメント / マイクロフィラメント /  ADP binding / sensory perception of sound / ADP binding / sensory perception of sound /  intracellular protein transport / ruffle membrane / intracellular protein transport / ruffle membrane /  エンドサイトーシス / エンドサイトーシス /  actin filament binding / actin filament binding /  マイクロフィラメント / apical part of cell / マイクロフィラメント / apical part of cell /  actin binding / actin binding /  細胞皮質 / cytoplasmic vesicle / 細胞皮質 / cytoplasmic vesicle /  核膜 / 核膜 /  calmodulin binding / calmodulin binding /  エンドソーム / response to xenobiotic stimulus / lysosomal membrane / perinuclear region of cytoplasm / エンドソーム / response to xenobiotic stimulus / lysosomal membrane / perinuclear region of cytoplasm /  ゴルジ体 / extracellular exosome / ゴルジ体 / extracellular exosome /  核質 / 核質 /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 : / : /  Myosin VI, lever arm / Myosin VI, lever arm /  Myosin VI, cargo binding domain / Class VI myosin, motor domain / Myosin VI cargo binding domain / Myosin S1 fragment, N-terminal / Myosin VI, cargo binding domain / Class VI myosin, motor domain / Myosin VI cargo binding domain / Myosin S1 fragment, N-terminal /  Myosin, N-terminal, SH3-like / Myosin N-terminal SH3-like domain profile. / Myosin, N-terminal, SH3-like / Myosin N-terminal SH3-like domain profile. /  Myosin head, motor domain / Myosin head (motor domain) ... Myosin head, motor domain / Myosin head (motor domain) ... : / : /  Myosin VI, lever arm / Myosin VI, lever arm /  Myosin VI, cargo binding domain / Class VI myosin, motor domain / Myosin VI cargo binding domain / Myosin S1 fragment, N-terminal / Myosin VI, cargo binding domain / Class VI myosin, motor domain / Myosin VI cargo binding domain / Myosin S1 fragment, N-terminal /  Myosin, N-terminal, SH3-like / Myosin N-terminal SH3-like domain profile. / Myosin, N-terminal, SH3-like / Myosin N-terminal SH3-like domain profile. /  Myosin head, motor domain / Myosin head (motor domain) / Myosin motor domain profile. / Myosin. Large ATPases. / Kinesin motor domain superfamily / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 Myosin head, motor domain / Myosin head (motor domain) / Myosin motor domain profile. / Myosin. Large ATPases. / Kinesin motor domain superfamily / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 Unconventional myosin-VI / Unconventional myosin-VI /  Unconventional myosin-VI類似検索 - 構成要素 Unconventional myosin-VI類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing |
|---|
| Model details | lowest energy, model1 |
|---|
 データ登録者 データ登録者 | He, F. / Walters, K. |
|---|
| 資金援助 |  米国, 2件 米国, 2件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Cancer Institute (NIH/NCI) | CA136472 |  米国 米国 | | National Institutes of Health/National Cancer Institute (NIH/NCI) | 1ZIABC011627 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Cell Rep / 年: 2016 ジャーナル: Cell Rep / 年: 2016
タイトル: Myosin VI Contains a Compact Structural Motif that Binds to Ubiquitin Chains.
著者: He, F. / Wollscheid, H.P. / Nowicka, U. / Biancospino, M. / Valentini, E. / Ehlinger, A. / Acconcia, F. / Magistrati, E. / Polo, S. / Walters, K.J. |
|---|
| 履歴 | | 登録 | 2015年3月20日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年3月9日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2016年3月30日 | Group: Database references |
|---|
| 改定 1.2 | 2016年4月6日 | Group: Database references |
|---|
| 改定 1.3 | 2016年4月27日 | Group: Structure summary |
|---|
| 改定 1.4 | 2021年10月13日 | Group: Data collection / Database references
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details |
|---|
| 改定 2.0 | 2021年10月20日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Author supporting evidence / Data collection / Database references / Derived calculations / Experimental preparation / Other / Refinement description / Source and taxonomy / Structure summary
カテゴリ: atom_site / entity_name_com ...atom_site / entity_name_com / entity_src_gen / pdbx_audit_support / pdbx_database_related / pdbx_database_status / pdbx_nmr_ensemble / pdbx_nmr_exptl / pdbx_nmr_exptl_sample / pdbx_nmr_exptl_sample_conditions / pdbx_nmr_refine / pdbx_nmr_sample_details / pdbx_nmr_software / pdbx_nmr_spectrometer / pdbx_poly_seq_scheme / pdbx_unobs_or_zero_occ_residues / pdbx_validate_close_contact / pdbx_validate_torsion / struct / struct_conf / struct_ref / struct_ref_seq / struct_ref_seq_dif
Item: _entity_src_gen.gene_src_common_name / _entity_src_gen.pdbx_beg_seq_num ..._entity_src_gen.gene_src_common_name / _entity_src_gen.pdbx_beg_seq_num / _entity_src_gen.pdbx_end_seq_num / _entity_src_gen.pdbx_gene_src_gene / _entity_src_gen.pdbx_seq_type / _pdbx_database_status.SG_entry / _pdbx_database_status.deposit_site / _pdbx_nmr_ensemble.conformer_selection_criteria / _pdbx_nmr_ensemble.conformers_calculated_total_number / _pdbx_nmr_exptl.sample_state / _pdbx_nmr_exptl.spectrometer_id / _pdbx_nmr_exptl_sample.component / _pdbx_nmr_exptl_sample_conditions.ionic_strength_units / _pdbx_nmr_exptl_sample_conditions.label / _pdbx_nmr_exptl_sample_conditions.pH_units / _pdbx_nmr_refine.software_ordinal / _pdbx_nmr_sample_details.contents / _pdbx_nmr_sample_details.label / _pdbx_nmr_sample_details.type / _pdbx_nmr_software.authors / _pdbx_nmr_software.classification / _pdbx_nmr_software.name / _pdbx_nmr_software.ordinal / _pdbx_nmr_software.version / _pdbx_nmr_spectrometer.type / _pdbx_poly_seq_scheme.auth_mon_id / _pdbx_poly_seq_scheme.auth_seq_num / _pdbx_poly_seq_scheme.pdb_mon_id / _struct.pdbx_CASP_flag / _struct_conf.beg_auth_comp_id / _struct_conf.beg_auth_seq_id / _struct_conf.beg_label_comp_id / _struct_conf.beg_label_seq_id / _struct_conf.end_auth_comp_id / _struct_conf.end_auth_seq_id / _struct_conf.end_label_comp_id / _struct_conf.end_label_seq_id / _struct_conf.pdbx_PDB_helix_id / _struct_conf.pdbx_PDB_helix_length / _struct_ref.db_code / _struct_ref.pdbx_align_begin / _struct_ref.pdbx_db_accession / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end / _struct_ref_seq.pdbx_db_accession / _struct_ref_seq_dif.pdbx_seq_db_accession_code
解説: Atoms with unrealistic or zero occupancies / Provider: author / タイプ: Coordinate replacement |
|---|
| 改定 2.1 | 2023年6月14日 | Group: Other / カテゴリ: pdbx_database_status / Item: _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 2.2 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素
 キーワード
キーワード MOTOR PROTEIN (モータータンパク質) /
MOTOR PROTEIN (モータータンパク質) /  PROTEIN TRANSPORT
PROTEIN TRANSPORT 機能・相同性情報
機能・相同性情報 regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle ...
regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle ... regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle / microfilament motor activity / inner ear morphogenesis / filamentous actin /
regulation of secretion / minus-end directed microfilament motor activity / unconventional myosin complex / actin filament-based movement / clathrin-coated vesicle membrane / inner ear auditory receptor cell differentiation / Gap junction degradation / vesicle transport along actin filament / Trafficking of AMPA receptors / RHOBTB1 GTPase cycle / microfilament motor activity / inner ear morphogenesis / filamentous actin /  微絨毛 / cytoskeletal motor activity / RHOU GTPase cycle / endocytic vesicle / DNA damage response, signal transduction by p53 class mediator /
微絨毛 / cytoskeletal motor activity / RHOU GTPase cycle / endocytic vesicle / DNA damage response, signal transduction by p53 class mediator /  オートファゴソーム / RHOBTB2 GTPase cycle /
オートファゴソーム / RHOBTB2 GTPase cycle /  クラスリン / ruffle /
クラスリン / ruffle /  filopodium / actin filament organization /
filopodium / actin filament organization /  マイクロフィラメント /
マイクロフィラメント /  ADP binding / sensory perception of sound /
ADP binding / sensory perception of sound /  intracellular protein transport / ruffle membrane /
intracellular protein transport / ruffle membrane /  エンドサイトーシス /
エンドサイトーシス /  actin filament binding /
actin filament binding /  マイクロフィラメント / apical part of cell /
マイクロフィラメント / apical part of cell /  actin binding /
actin binding /  細胞皮質 / cytoplasmic vesicle /
細胞皮質 / cytoplasmic vesicle /  核膜 /
核膜 /  calmodulin binding /
calmodulin binding /  エンドソーム / response to xenobiotic stimulus / lysosomal membrane / perinuclear region of cytoplasm /
エンドソーム / response to xenobiotic stimulus / lysosomal membrane / perinuclear region of cytoplasm /  ゴルジ体 / extracellular exosome /
ゴルジ体 / extracellular exosome /  核質 /
核質 /  ATP binding /
ATP binding /  生体膜 / identical protein binding /
生体膜 / identical protein binding /  細胞核 /
細胞核 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR /
溶液NMR /  simulated annealing
simulated annealing  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Cell Rep / 年: 2016
ジャーナル: Cell Rep / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2n0z.cif.gz
2n0z.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2n0z.ent.gz
pdb2n0z.ent.gz PDB形式
PDB形式 2n0z.json.gz
2n0z.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/n0/2n0z
https://data.pdbj.org/pub/pdb/validation_reports/n0/2n0z ftp://data.pdbj.org/pub/pdb/validation_reports/n0/2n0z
ftp://data.pdbj.org/pub/pdb/validation_reports/n0/2n0z リンク
リンク 集合体
集合体
 要素
要素

 Homo sapiens (ヒト) / 遺伝子: MYO6 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: MYO6 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: Q5JVM0, UniProt: Q9UM54*PLUS
Escherichia coli (大腸菌) / 参照: UniProt: Q5JVM0, UniProt: Q9UM54*PLUS 溶液NMR
溶液NMR 試料調製
試料調製 : AVANCE / 磁場強度: 700 MHz
: AVANCE / 磁場強度: 700 MHz 解析
解析 simulated annealing / ソフトェア番号: 2
simulated annealing / ソフトェア番号: 2  ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj








