+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2fum | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Catalytic domain of protein kinase PknB from Mycobacterium tuberculosis in complex with mitoxantrone | ||||||
 要素 要素 | Probable serine/threonine-protein kinase pknB | ||||||
 キーワード キーワード | TRANSFERASE / PROTEIN KINASE-INHIBITOR COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of growth rate / acetyltransferase activator activity / negative regulation of catalytic activity / negative regulation of fatty acid biosynthetic process / response to host immune response / positive regulation of catalytic activity / positive regulation of DNA binding / peptidoglycan biosynthetic process / protein serine/threonine/tyrosine kinase activity / peptidoglycan-based cell wall ...negative regulation of growth rate / acetyltransferase activator activity / negative regulation of catalytic activity / negative regulation of fatty acid biosynthetic process / response to host immune response / positive regulation of catalytic activity / positive regulation of DNA binding / peptidoglycan biosynthetic process / protein serine/threonine/tyrosine kinase activity / peptidoglycan-based cell wall / negative regulation of protein binding / manganese ion binding / regulation of cell shape / protein autophosphorylation / membrane => GO:0016020 / non-specific serine/threonine protein kinase / protein kinase activity / protein phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / ATP binding / identical protein binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.89 Å 分子置換 / 解像度: 2.89 Å | ||||||
 データ登録者 データ登録者 | Wehenkel, A. / Alzari, P.M. | ||||||
 引用 引用 |  ジャーナル: Febs Lett. / 年: 2006 ジャーナル: Febs Lett. / 年: 2006タイトル: The structure of PknB in complex with mitoxantrone, an ATP-competitive inhibitor, suggests a mode of protein kinase regulation in mycobacteria 著者: Wehenkel, A. / Fernandez, P. / Bellinzoni, M. / Catherinot, V. / Barilone, N. / Labesse, G. / Jackson, M. / Alzari, P.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2fum.cif.gz 2fum.cif.gz | 204.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2fum.ent.gz pdb2fum.ent.gz | 164 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2fum.json.gz 2fum.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2fum_validation.pdf.gz 2fum_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2fum_full_validation.pdf.gz 2fum_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  2fum_validation.xml.gz 2fum_validation.xml.gz | 42.2 KB | 表示 | |
| CIF形式データ |  2fum_validation.cif.gz 2fum_validation.cif.gz | 55.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fu/2fum https://data.pdbj.org/pub/pdb/validation_reports/fu/2fum ftp://data.pdbj.org/pub/pdb/validation_reports/fu/2fum ftp://data.pdbj.org/pub/pdb/validation_reports/fu/2fum | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1o6yS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Refine code: 2
NCSアンサンブル:
|
- 要素
要素
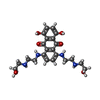
| #1: タンパク質 | 分子量: 32458.506 Da / 分子数: 4 / 断片: CATALYTIC DOMAIN / 由来タイプ: 組換発現 由来: (組換発現)  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌)生物種: Mycobacterium tuberculosis / 株: H37RV / 遺伝子: pknB / プラスミド: PET28 / 生物種 (発現宿主): Escherichia coli / 発現宿主:  #2: 化合物 | ChemComp-MIX / |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.42 Å3/Da / 溶媒含有率: 64.05 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.6 詳細: 1.2M NaAcetate 50mM NaCacodylate, pH 5.6, VAPOR DIFFUSION, HANGING DROP, temperature 291K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID14-3 / 波長: 0.931 Å / ビームライン: ID14-3 / 波長: 0.931 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 2005年3月10日 |
| 放射 | モノクロメーター: Diamond (111), Ge(220) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.931 Å / 相対比: 1 |
| 反射 | 解像度: 2.89→69.84 Å / Num. all: 28195 / Num. obs: 28195 / % possible obs: 63.47 % / Observed criterion σ(F): 1 / Observed criterion σ(I): 1 |
| 反射 シェル | 解像度: 2.89→3.21 Å / % possible all: 16 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1O6Y 解像度: 2.89→69.84 Å / Cor.coef. Fo:Fc: 0.938 / Cor.coef. Fo:Fc free: 0.905 / SU B: 53.161 / SU ML: 0.412 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.509 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: THE DATA IS HIGHLY ANISOTROPIC (2.89 A RESOLUTION LIMIT ALONG THE c* AXIS, BUT ONLY 3.5 A ALONG THE a* AND b* AXES), ACCOUNTING FOR THE POOR DATA COMPLETENESS AT THE HIGHER RESOLUTION SHELLS ...詳細: THE DATA IS HIGHLY ANISOTROPIC (2.89 A RESOLUTION LIMIT ALONG THE c* AXIS, BUT ONLY 3.5 A ALONG THE a* AND b* AXES), ACCOUNTING FOR THE POOR DATA COMPLETENESS AT THE HIGHER RESOLUTION SHELLS (DATA IS 99% COMPLETE AT 3.49 A, 45% IN THE 3.49-3.21 A SHELL, AND ONLY 16% IN THE 3.21-2.89 A SHELL.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 79.784 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.89→69.84 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Dom-ID: 1 / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.891→2.966 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj