+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ibi | ||||||
|---|---|---|---|---|---|---|---|
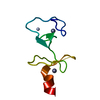
| タイトル | QUAIL CYSTEINE AND GLYCINE-RICH PROTEIN, NMR, 15 MINIMIZED MODEL STRUCTURES | ||||||
 要素 要素 | CYSTEINE-RICH PROTEIN 2 | ||||||
 キーワード キーワード | METAL BINDING PROTEIN / LIM DOMAIN CONTAINING PROTEINS /  METAL-BINDING PROTEIN METAL-BINDING PROTEIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |   Coturnix japonica (ウズラ) Coturnix japonica (ウズラ) | ||||||
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing | ||||||
 データ登録者 データ登録者 | Schuler, W. / Kloiber, K. / Matt, T. / Bister, K. / Konrat, R. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2001 ジャーナル: Biochemistry / 年: 2001タイトル: Application of cross-correlated NMR spin relaxation to the zinc-finger protein CRP2(LIM2): evidence for collective motions in LIM domains. 著者: Schuler, W. / Kloiber, K. / Matt, T. / Bister, K. / Konrat, R. #1:  ジャーナル: J.Biol.Chem. / 年: 1998 ジャーナル: J.Biol.Chem. / 年: 1998タイトル: STRUCTURE OF CYSTEINE- AND GLYCINE-RICH PROTEIN CRP2 著者: Konrat, R. / Kraeutler, B. / Weiskirchen, R. / Bister, K. #2:  ジャーナル: J.Biol.Chem. / 年: 1997 ジャーナル: J.Biol.Chem. / 年: 1997タイトル: Solution Structure of the Carboxyl-terminal LIM Domain from Quail Cystein-rich Protein CRP2 著者: Konrat, R. / Weiskirchen, R. / Kraeutler, B. / Bister, K. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ibi.cif.gz 1ibi.cif.gz | 268.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ibi.ent.gz pdb1ibi.ent.gz | 225.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ibi.json.gz 1ibi.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ib/1ibi https://data.pdbj.org/pub/pdb/validation_reports/ib/1ibi ftp://data.pdbj.org/pub/pdb/validation_reports/ib/1ibi ftp://data.pdbj.org/pub/pdb/validation_reports/ib/1ibi | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 12125.771 Da / 分子数: 1 / 断片: C-TERMINAL LIM DOMAIN, RESIDUES 82-194 / 由来タイプ: 組換発現 / 由来: (組換発現)   Coturnix japonica (ウズラ) / 遺伝子: CSRP2 / プラスミド: PET3D-QCRP2(LIM2) / 発現宿主: Coturnix japonica (ウズラ) / 遺伝子: CSRP2 / プラスミド: PET3D-QCRP2(LIM2) / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)PLYSS / 参照: UniProt: Q05158 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)PLYSS / 参照: UniProt: Q05158 |
|---|---|
| #2: 化合物 |
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: Projection data related to the backbone dihedrals phi, psi from CaHa(i)-DD/C'(i)-CSA and CAHA(i)-DD/C'(i-1)-CSA relaxation and 15N(i)-1HN(i)-DD/13CO(i)-CSA and 1H(i)-15N(i)-13C'(i) DD-CSA cross ...Text: Projection data related to the backbone dihedrals phi, psi from CaHa(i)-DD/C'(i)-CSA and CAHA(i)-DD/C'(i-1)-CSA relaxation and 15N(i)-1HN(i)-DD/13CO(i)-CSA and 1H(i)-15N(i)-13C'(i) DD-CSA cross correlation experiments. Side-chain dihedral angles chi1 from 2D quantitative J-spectroscopy experiments giving access to 3J(CO,Cgamma) and 3J(N,Cgamma). Differential relaxation of multiple-quantum coherences Gamma(i,j)=R(DQ)-R(ZQ), i,j denote 15N,1HN or 13C',15N, respectively. |
- 試料調製
試料調製
| 詳細 |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 | イオン強度: 20mM potassium phosphate; 50mM KCl / pH: 7.2 / 圧: ambient / 温度: 299 K | |||||||||
結晶化 | *PLUS 手法: その他 / 詳細: NMR |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Varian UNITYPLUS 500 / 製造業者: Varian / モデル : UNITYPLUS 500 / 磁場強度: 500 MHz : UNITYPLUS 500 / 磁場強度: 500 MHz |
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法:  simulated annealing / ソフトェア番号: 1 simulated annealing / ソフトェア番号: 1 詳細: This entry represents the C-terminal LIM domain of the quail cysteine rich protein CRP2(LIM2) consisting of 15 minimized final structures. Residues MET 82 - ALA 117 and ASN 175 - GLN 194 were ...詳細: This entry represents the C-terminal LIM domain of the quail cysteine rich protein CRP2(LIM2) consisting of 15 minimized final structures. Residues MET 82 - ALA 117 and ASN 175 - GLN 194 were not observed and there is no information regarding the conformation that these residues may adopt in solution. The only stretch of residues for which there is structural information is GLU 118 - LYS 174 so the simulations were restricted to the central part ALA 117 - ASN 175, containing the two zinc knuckles. In an initial stage zinc was covalently attached to a linearized template structure, then tetrahedral coordination was enforced by distance constraints. Finally the zinc ions were covalently attached to the coordinating residues using standard force field parameters. 653 inter- and intra-residue NOE restraints were defined as strong, medium, weak and very weak and applied during the final refinement as well as 28 torsion angle restraints and 15 hydrogen bond restraints. Omega dihedral angles were restrained to trans (E). Average rmsd from exp. distance restraints(angstroms) 0.01, average rmsd from idealized covalent geometry: bonds(angstroms): 0.01; angles(deg) 1.98; | ||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations,structures with the lowest energy 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 15 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

