+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9005 | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM reconstruction of IST1-CHMP1B copolymer filament bound to ssDNA at 2.9 Angstrom resolution | |||||||||||||||||||||||||||
 マップデータ マップデータ | Post processed map with imposed helical symmetry | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード |  ESCRT-III (ESCRT) / ESCRT-III (ESCRT) /  CHMP1B / IST1 / CHMP1B / IST1 /  ssDNA (デオキシリボ核酸) / ssDNA (デオキシリボ核酸) /  DNA BINDING PROTEIN (DNA結合タンパク質) / PROTEIN FIBRIL DNA BINDING PROTEIN (DNA結合タンパク質) / PROTEIN FIBRIL | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral capsid secondary envelopment / MIT domain binding /  abscission / amphisome membrane / multivesicular body-lysosome fusion / vesicle fusion with vacuole / ESCRT III complex disassembly / late endosome to lysosome transport / abscission / amphisome membrane / multivesicular body-lysosome fusion / vesicle fusion with vacuole / ESCRT III complex disassembly / late endosome to lysosome transport /  ESCRT III complex / kinetochore microtubule ...viral capsid secondary envelopment / MIT domain binding / ESCRT III complex / kinetochore microtubule ...viral capsid secondary envelopment / MIT domain binding /  abscission / amphisome membrane / multivesicular body-lysosome fusion / vesicle fusion with vacuole / ESCRT III complex disassembly / late endosome to lysosome transport / abscission / amphisome membrane / multivesicular body-lysosome fusion / vesicle fusion with vacuole / ESCRT III complex disassembly / late endosome to lysosome transport /  ESCRT III complex / kinetochore microtubule / cytoskeleton-dependent cytokinesis / endosome transport via multivesicular body sorting pathway / collateral sprouting / regulation of centrosome duplication / Sealing of the nuclear envelope (NE) by ESCRT-III / nuclear membrane reassembly / positive regulation of collateral sprouting / midbody abscission / multivesicular body sorting pathway / membrane coat / membrane fission / plasma membrane repair / late endosome to vacuole transport / multivesicular body membrane / ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway / ESCRT III complex / kinetochore microtubule / cytoskeleton-dependent cytokinesis / endosome transport via multivesicular body sorting pathway / collateral sprouting / regulation of centrosome duplication / Sealing of the nuclear envelope (NE) by ESCRT-III / nuclear membrane reassembly / positive regulation of collateral sprouting / midbody abscission / multivesicular body sorting pathway / membrane coat / membrane fission / plasma membrane repair / late endosome to vacuole transport / multivesicular body membrane / ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway /  multivesicular body assembly / multivesicular body assembly /  regulation of mitotic spindle assembly / Flemming body / regulation of mitotic spindle assembly / Flemming body /  nucleus organization / mitotic metaphase chromosome alignment / viral budding via host ESCRT complex / autophagosome maturation / autophagosome membrane / positive regulation of proteolysis / viral release from host cell / nucleus organization / mitotic metaphase chromosome alignment / viral budding via host ESCRT complex / autophagosome maturation / autophagosome membrane / positive regulation of proteolysis / viral release from host cell /  endoplasmic reticulum-Golgi intermediate compartment / endoplasmic reticulum-Golgi intermediate compartment /  核膜孔 / 核膜孔 /  エンドソーム / エンドソーム /  viral budding from plasma membrane / establishment of protein localization / viral budding from plasma membrane / establishment of protein localization /  protein localization / protein localization /  動原体 / 動原体 /  オートファジー / azurophil granule lumen / オートファジー / azurophil granule lumen /  protein transport / protein transport /  核膜 / midbody / endosome membrane / 核膜 / midbody / endosome membrane /  cadherin binding / cadherin binding /  細胞分裂 / lysosomal membrane / protein domain specific binding / intracellular membrane-bounded organelle / 細胞分裂 / lysosomal membrane / protein domain specific binding / intracellular membrane-bounded organelle /  中心体 / 中心体 /  クロマチン / Neutrophil degranulation / protein-containing complex binding / extracellular exosome / extracellular region / クロマチン / Neutrophil degranulation / protein-containing complex binding / extracellular exosome / extracellular region /  核質 / identical protein binding / 核質 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Talledge N / Frost A | |||||||||||||||||||||||||||
| 資金援助 |  米国, 8件 米国, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Biorxiv / 年: 2018 ジャーナル: Biorxiv / 年: 2018タイトル: The ESCRT-III proteins IST1 and CHMP1B assemble around nucleic acids 著者: Talledge N / McCullough J / Wenzel DM / Nguyen HC / Lalonde M / Bajorek M / Skalicky JJ / Frost A / Sundquist WI | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9005.map.gz emd_9005.map.gz | 54 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9005-v30.xml emd-9005-v30.xml emd-9005.xml emd-9005.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9005_fsc.xml emd_9005_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9005.png emd_9005.png | 207.5 KB | ||
| Filedesc metadata |  emd-9005.cif.gz emd-9005.cif.gz | 6.9 KB | ||
| その他 |  emd_9005_half_map_1.map.gz emd_9005_half_map_1.map.gz emd_9005_half_map_2.map.gz emd_9005_half_map_2.map.gz | 193.6 MB 193.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9005 http://ftp.pdbj.org/pub/emdb/structures/EMD-9005 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9005 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9005 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9005.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9005.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post processed map with imposed helical symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.838 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 2
| ファイル | emd_9005_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
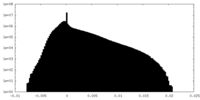
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_9005_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Helical assembly of IST1 and CHMP1B protein bound to ssDNA
| 全体 | 名称: Helical assembly of IST1 and CHMP1B protein bound to ssDNA |
|---|---|
| 要素 |
|
-超分子 #1: Helical assembly of IST1 and CHMP1B protein bound to ssDNA
| 超分子 | 名称: Helical assembly of IST1 and CHMP1B protein bound to ssDNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 細胞中の位置: cytoplasm Homo sapiens (ヒト) / 細胞中の位置: cytoplasm |
-分子 #1: IST1 homolog
| 分子 | 名称: IST1 homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 36 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 40.024711 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MLGSGFKAER LRVNLRLVIN RLKLLEKKKT ELAQKARKEI ADYLAAGKDE RARIRVEHII REDYLVEAME ILELYCDLLL ARFGLIQSM KELDSGLAES VSTLIWAAPR LQSEVAELKI VADQLCAKYS KEYGKLCRTN QIGTVNDRLM HKLSVEAPPK I LVERYLIE ...文字列: MLGSGFKAER LRVNLRLVIN RLKLLEKKKT ELAQKARKEI ADYLAAGKDE RARIRVEHII REDYLVEAME ILELYCDLLL ARFGLIQSM KELDSGLAES VSTLIWAAPR LQSEVAELKI VADQLCAKYS KEYGKLCRTN QIGTVNDRLM HKLSVEAPPK I LVERYLIE IAKNYNVPYE PDSVVMAEAP PGVETDLIDV GFTDDVKKGG PGRGGSGGFT APVGGPDGTV PMPMPMPMPM PS ANTPFSY PLPKGPSDFN GLPMGTYQAF PNIHPPQIPA TPPSYESVDD INADKNISSA QIVGPGPKPE ASAKLPSRPA DNY DNFVLP ELPSVPDTLP TASAGASTSA SEDIDFDDLS RRFEELKKKT UniProtKB: IST1 homolog |
-分子 #2: Charged multivesicular body protein 1b
| 分子 | 名称: Charged multivesicular body protein 1b / タイプ: protein_or_peptide / ID: 2 / コピー数: 36 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.140354 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MSNMEKHLFN LKFAAKELSR SAKKCDKEEK AEKAKIEKAI QKGNMEVARI HAENAIRQKN QAVNFLRMSA RVDAVAARVQ TAVTMGKVT KSMAGVVKSM DATLKTMNLE KISALMDKFE HQFETLDVQT QQMEDTMSST TTLTTPQNQV DMLLQEMADE A GLDLNMEL ...文字列: MSNMEKHLFN LKFAAKELSR SAKKCDKEEK AEKAKIEKAI QKGNMEVARI HAENAIRQKN QAVNFLRMSA RVDAVAARVQ TAVTMGKVT KSMAGVVKSM DATLKTMNLE KISALMDKFE HQFETLDVQT QQMEDTMSST TTLTTPQNQV DMLLQEMADE A GLDLNMEL PQGQTGSVGT SVASAEQDEL SQRLARLRDQ V UniProtKB: Charged multivesicular body protein 1b |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 292.15 K / 装置: FEI VITROBOT MARK III 詳細: 0 mm offset with 10 sec wait time and 2-4 sec blot. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.6 mm / 倍率(公称値): 29000 Bright-field microscopy / Cs: 2.6 mm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 3-32 / 撮影したグリッド数: 1 / 実像数: 2980 / 平均露光時間: 6.2 sec. / 平均電子線量: 62.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 52.5 |
|---|---|
| 得られたモデル |  PDB-6e8g: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X