+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8610 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of E. coli MCE protein PqiB, periplasmic domain | ||||||||||||
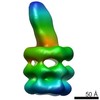
 マップデータ マップデータ | E. coli MCE protein PqiB, periplasmic domain | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 | Mce/MlaD / MlaD protein / intermembrane lipid transfer / outer membrane-bounded periplasmic space / identical protein binding /  細胞膜 / Intermembrane transport protein PqiB 細胞膜 / Intermembrane transport protein PqiB 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 10.0 Å クライオ電子顕微鏡法 / 解像度: 10.0 Å | ||||||||||||
 データ登録者 データ登録者 | Bhabha G / Ekiert DC | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Architectures of Lipid Transport Systems for the Bacterial Outer Membrane. 著者: Damian C Ekiert / Gira Bhabha / Georgia L Isom / Garrett Greenan / Sergey Ovchinnikov / Ian R Henderson / Jeffery S Cox / Ronald D Vale /   要旨: How phospholipids are trafficked between the bacterial inner and outer membranes through the hydrophilic space of the periplasm is not known. We report that members of the mammalian cell entry (MCE) ...How phospholipids are trafficked between the bacterial inner and outer membranes through the hydrophilic space of the periplasm is not known. We report that members of the mammalian cell entry (MCE) protein family form hexameric assemblies with a central channel capable of mediating lipid transport. The E. coli MCE protein, MlaD, forms a ring associated with an ABC transporter complex in the inner membrane. A soluble lipid-binding protein, MlaC, ferries lipids between MlaD and an outer membrane protein complex. In contrast, EM structures of two other E. coli MCE proteins show that YebT forms an elongated tube consisting of seven stacked MCE rings, and PqiB adopts a syringe-like architecture. Both YebT and PqiB create channels of sufficient length to span the periplasmic space. This work reveals diverse architectures of highly conserved protein-based channels implicated in the transport of lipids between the membranes of bacteria and some eukaryotic organelles. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8610.map.gz emd_8610.map.gz | 265.4 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8610-v30.xml emd-8610-v30.xml emd-8610.xml emd-8610.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8610.png emd_8610.png | 57.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8610 http://ftp.pdbj.org/pub/emdb/structures/EMD-8610 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8610 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8610 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8610.map.gz / 形式: CCP4 / 大きさ: 1.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8610.map.gz / 形式: CCP4 / 大きさ: 1.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli MCE protein PqiB, periplasmic domain | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : MlaFEDB
| 全体 | 名称: MlaFEDB |
|---|---|
| 要素 |
|
-超分子 #1: MlaFEDB
| 超分子 | 名称: MlaFEDB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: MlaF
| 分子 | 名称: MlaF / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MEQSVANLVD MRDVSFTRGN RCIFDNISL TVPRGKITAI M GPSGIGKT TLLRLIGGQI AP DHGEILF DGENIPAMSR SRL YTVRKR MSMLFQSGAL FTDM NVFDN VAYPLREHTQ LPAPL LHST VMMKLEAVGL RGAAKL MPS ELSGGMARRA ALARAIA LE ...文字列: MEQSVANLVD MRDVSFTRGN RCIFDNISL TVPRGKITAI M GPSGIGKT TLLRLIGGQI AP DHGEILF DGENIPAMSR SRL YTVRKR MSMLFQSGAL FTDM NVFDN VAYPLREHTQ LPAPL LHST VMMKLEAVGL RGAAKL MPS ELSGGMARRA ALARAIA LE PDLIMFDEPF VGQDPITM G VLVKLISELN SALGVTCVV VSHDVPEVLS IADHAWILAD KKIVAHGSA QALQANPDPR V RQFLDGIA DGPVPFRYPA GD YHADLLP GS |
-分子 #2: MlaE
| 分子 | 名称: MlaE / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MLLNALASLG HKGIKTLRTF GRAGLMLFN ALVGKPEFRK H APLLVRQL YNVGVLSMLI IV VSGVFIG MVLGLQGYLV LTT YSAETS LGMLVALSLL RELG PVVAA LLFAGRAGSA LTAEI GLMR ATEQLSSMEM MAVDPL RRV ISPRFWAGVI SLPLLTV IF ...文字列: MLLNALASLG HKGIKTLRTF GRAGLMLFN ALVGKPEFRK H APLLVRQL YNVGVLSMLI IV VSGVFIG MVLGLQGYLV LTT YSAETS LGMLVALSLL RELG PVVAA LLFAGRAGSA LTAEI GLMR ATEQLSSMEM MAVDPL RRV ISPRFWAGVI SLPLLTV IF VAVGIWGGSL VGVSWKGI D SGFFWSAMQN AVDWRMDLV NCLIKSVVFA ITVTWISLFN GYDAIPTSA GISRATTRTV V HSSLAVLG LDFVLTALMF GN |
-分子 #3: MlaD
| 分子 | 名称: MlaD / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MHHHHHHQHQ HENLYFQGMQ TKKNEIWVG IFLLAALLAA L FVCLKAAN VTSIRTEPTY TL YATFDNI GGLKARSPVS IGG VVVGRV ADITLDPKTY LPRV TLEIE QRYNHIPDTS SLSIR TSGL LGEQYLALNV GFEDPE LGT AILKDGDTIQ DTKSAMV LE ...文字列: MHHHHHHQHQ HENLYFQGMQ TKKNEIWVG IFLLAALLAA L FVCLKAAN VTSIRTEPTY TL YATFDNI GGLKARSPVS IGG VVVGRV ADITLDPKTY LPRV TLEIE QRYNHIPDTS SLSIR TSGL LGEQYLALNV GFEDPE LGT AILKDGDTIQ DTKSAMV LE DLIGQFLYGS KGDDNKNS G DAPAAAPGNN ETTEPVGTT K |
-分子 #4: MlaB
| 分子 | 名称: MlaB / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSESLSWMQT GDTLALSGEL DQDVLLPLW EMREEAVKGI T CIDLSRVS RVDTGGLALL LH LIDLAKK QGNNVTLQGV NDK VYTLAK LYNLPADVLP R |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 詳細: 20 mM Tris pH 8.0 and 150 mM NaCl, and exchanged from DDM into amphipol A8-35 as described in the manuscript |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 80.0 e/Å2 / 詳細: 80 e/A2 is total dose for 50 frames |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND4 |
|---|---|
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 角度割当 | タイプ: OTHER / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 10.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) / 使用した粒子像数: 28012 ) / 解像度のタイプ: BY AUTHOR / 解像度: 10.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) / 使用した粒子像数: 28012 |
-原子モデル構築 1
| 詳細 | crystal structure of MlaD and homology models for MlaF, MlaE and MlaB were fit into the density as described in the manuscript |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー