+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8547 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Kinesin-1 Binding Microtubules with ADP-AlFx in a One-head-bound State (low-pass filtered to 8.5 angstrom) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Bos taurus (ウシ) / Bos taurus (ウシ) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.7 Å クライオ電子顕微鏡法 / 解像度: 6.7 Å | |||||||||
 データ登録者 データ登録者 | Liu D / Liu X / Shang Z / Sindelar CV | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2017 ジャーナル: Elife / 年: 2017タイトル: Structural basis of cooperativity in kinesin revealed by 3D reconstruction of a two-head-bound state on microtubules. 著者: Daifei Liu / Xueqi Liu / Zhiguo Shang / Charles V Sindelar /  要旨: The detailed basis of walking by dimeric molecules of kinesin along microtubules has remained unclear, partly because available structural methods have been unable to capture microtubule-bound ...The detailed basis of walking by dimeric molecules of kinesin along microtubules has remained unclear, partly because available structural methods have been unable to capture microtubule-bound intermediates of this process. Utilizing novel electron cryomicroscopy methods, we solved structures of microtubule-attached, dimeric kinesin bound to an ATP analog. We find that under these conditions, the kinesin dimer can attach to the microtubule with either one or two motor domains, and we present sub-nanometer resolution reconstructions of both states. The former structure reveals a novel kinesin conformation that revises the current understanding of how ATP binding is coupled to forward stepping of the motor. The latter structure indicates how tension between the two motor domains keeps their cycles out of phase in order to stimulate directional motility. The methods presented here pave the way for future structural studies of a variety of challenging macromolecules that bind to microtubules and other filaments. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8547.map.gz emd_8547.map.gz | 147.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8547-v30.xml emd-8547-v30.xml emd-8547.xml emd-8547.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8547.png emd_8547.png | 74.5 KB | ||
| その他 |  emd_8547_half_map_1.map.gz emd_8547_half_map_1.map.gz emd_8547_half_map_2.map.gz emd_8547_half_map_2.map.gz | 118.5 MB 118.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8547 http://ftp.pdbj.org/pub/emdb/structures/EMD-8547 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8547 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8547 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8547.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8547.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 2.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_8547_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
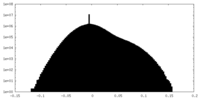
| 投影像・断面図 |
| ||||||||||||
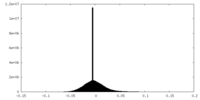
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_8547_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
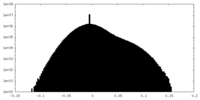
| 投影像・断面図 |
| ||||||||||||
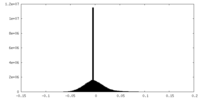
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Kinesin-1 (1-420) on microtubules with ADP-AlFx in a one-head-bou...
| 全体 | 名称: Kinesin-1 (1-420) on microtubules with ADP-AlFx in a one-head-bound state |
|---|---|
| 要素 |
|
-超分子 #1: Kinesin-1 (1-420) on microtubules with ADP-AlFx in a one-head-bou...
| 超分子 | 名称: Kinesin-1 (1-420) on microtubules with ADP-AlFx in a one-head-bound state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
-分子 #1: Kinesin-1
| 分子 | 名称: Kinesin-1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MADLAESNIK VMSRFRPLNE SEVNRGDKYI AKFQGEDTVV IASKPYAFDR VFQSSTSQEQ VYNDSAKKIV KDVLEGYNGT IFAYGQTSSG KTHTMEGKLH DPEGMGIIPR IVQDIFNYIY SMDENLEFHI KVSYFEIYLD KIRDLLDVSK TNLSVHEDKN RVPYVKGSTE ...文字列: MADLAESNIK VMSRFRPLNE SEVNRGDKYI AKFQGEDTVV IASKPYAFDR VFQSSTSQEQ VYNDSAKKIV KDVLEGYNGT IFAYGQTSSG KTHTMEGKLH DPEGMGIIPR IVQDIFNYIY SMDENLEFHI KVSYFEIYLD KIRDLLDVSK TNLSVHEDKN RVPYVKGSTE RFVSSPDEVM DTIDEGKSNR HVAVTNMNEH SSRSHSIFLI NVKQENTQTE QKLSGKLYLV DLAGSEKVSK TGAEGAVLDE AKNINKSLSA LGNVISALAE GSTYVPYRDS KMTRILQDSL GGNSRTTIVI SSSPSSYNES ETKSTLLFGQ RAKTIKNTVS VNVELTAEQW KKKYEKEKEK NKILRNTIQW LENELNRWRN GETVPIDEQF DKEKANLEAF TVDKDITLTN DKPATAIGVI GNFTDAERRK |
-分子 #2: Alpha-tubulin
| 分子 | 名称: Alpha-tubulin / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
| 配列 | 文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHPE QLITGKEDAA NNYARGHYTI GKEIIDLVLD RIRKLADQCT GLQGFLVFHS FGGGTGSGFT SLLMERLSVD YGKKSKLEFS ...文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHPE QLITGKEDAA NNYARGHYTI GKEIIDLVLD RIRKLADQCT GLQGFLVFHS FGGGTGSGFT SLLMERLSVD YGKKSKLEFS IYPAPQVSTA VVEPYNSILT THTTLEHSDC AFMVDNEAIY DICRRNLDIE RPTYTNLNRL ISQIVSSITA SLRFDGALNV DLTEFQTNLV PYPRIHFPLA TYAPVISAEK AYHEQLSVAE ITNACFEPAN QMVKCDPRHG KYMACCLLYR GDVVPKDVNA AIATIKTKRS IQFVDWCPTG FKVGINYQPP TVVPGGDLAK VQRAVCMLSN TTAIAEAWAR LDHKFDLMYA KRAFVHWYVG EGMEEGEFSE AREDMAALEK DYEEVGVDSV EGEGEEEGEE Y |
-分子 #3: Beta-tubulin
| 分子 | 名称: Beta-tubulin / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
| 配列 | 文字列: MREIVHLQAG QCGNQIGAKF WEVISDEHGI DPTGTYHGDS DLQLERINVY YNEATGGNYV PRAVLVDLEP GTMDSVRSGP FGQIFRPDNF VFGQSGAGNN WAKGHYTEGA ELVDAVLDVV RKEAESCDCL QGFQLTHSLG GGTGSGMGTL LISKIREEFP DRIMNTFSVV ...文字列: MREIVHLQAG QCGNQIGAKF WEVISDEHGI DPTGTYHGDS DLQLERINVY YNEATGGNYV PRAVLVDLEP GTMDSVRSGP FGQIFRPDNF VFGQSGAGNN WAKGHYTEGA ELVDAVLDVV RKEAESCDCL QGFQLTHSLG GGTGSGMGTL LISKIREEFP DRIMNTFSVV PSPKVSDTVV EPYNATLSVH QLVENTDETY CIDNEALYDI CFRTLKLTTP TYGDLNHLVS ATMSGVTTCL RFPGQLNADL RKLAVNMVPF PRLHFFMPGF APLTSRGSQQ YRALTVPELT QQMFDAKNMM AACDPRHGRY LTVAAVFRGR MSMKEVDEQM LSVQSKNSSY FVEWIPNNVK TAVCDIPPRG LKMAATFIGN STAIQELFKR ISEQFTAMFR RKAFLHWYTG EGMDEMEFTE AESNMNDLVS EYQQYQDATA EEGEFEEEAE EEVA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 平均電子線量: 1.65 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
|---|---|
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 6.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF 詳細: a modified version of frealign was used for reconstruction, kinesin dimer was masked before FSC calculation using half maps. 使用した粒子像数: 44298 |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X