+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6165 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Essential Structural and Functional Roles of the Cmr4 Subunit in RNA Cleavage by the Cmr CRISPR-Cas Complex | |||||||||
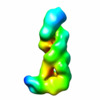
 マップデータ マップデータ | Reconstruction of Cmr1-6 with crRNA and target RNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / Cmr complex / RNA interference / Cmr proteins | |||||||||
| 生物種 |   Pyrococcus furiosus (古細菌) / unidentified (未定義) Pyrococcus furiosus (古細菌) / unidentified (未定義) | |||||||||
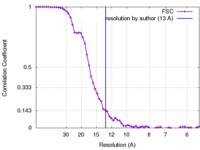
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 13.0 Å | |||||||||
 データ登録者 データ登録者 | Ramia NF / Spilman M / Tang L / Shao Y / Elmore J / Hale C / Cocozaki A / Bhattacharya N / Terns RM / Terns MP ...Ramia NF / Spilman M / Tang L / Shao Y / Elmore J / Hale C / Cocozaki A / Bhattacharya N / Terns RM / Terns MP / Li H / Stagg SM | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2014 ジャーナル: Cell Rep / 年: 2014タイトル: Essential structural and functional roles of the Cmr4 subunit in RNA cleavage by the Cmr CRISPR-Cas complex. 著者: Nancy F Ramia / Michael Spilman / Li Tang / Yaming Shao / Joshua Elmore / Caryn Hale / Alexis Cocozaki / Nilakshee Bhattacharya / Rebecca M Terns / Michael P Terns / Hong Li / Scott M Stagg /  要旨: The Cmr complex is the multisubunit effector complex of the type III-B clustered regularly interspaced short palindromic repeats (CRISPR)-Cas immune system. The Cmr complex recognizes a target RNA ...The Cmr complex is the multisubunit effector complex of the type III-B clustered regularly interspaced short palindromic repeats (CRISPR)-Cas immune system. The Cmr complex recognizes a target RNA through base pairing with the integral CRISPR RNA (crRNA) and cleaves the target at multiple regularly spaced locations within the complementary region. To understand the molecular basis of the function of this complex, we have assembled information from electron microscopic and X-ray crystallographic structural studies and mutagenesis of a complete Pyrococcus furiosus Cmr complex. Our findings reveal that four helically packed Cmr4 subunits, which make up the backbone of the Cmr complex, act as a platform to support crRNA binding and target RNA cleavage. Interestingly, we found a hook-like structural feature associated with Cmr4 that is likely the site of target RNA binding and cleavage. Our results also elucidate analogies in the mechanisms of crRNA and target molecule binding by the distinct Cmr type III-A and Cascade type I-E complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6165.map.gz emd_6165.map.gz | 13.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6165-v30.xml emd-6165-v30.xml emd-6165.xml emd-6165.xml | 13.5 KB 13.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6165_fsc.xml emd_6165_fsc.xml | 6.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  400_6165.gif 400_6165.gif 80_6165.gif 80_6165.gif | 27.9 KB 2.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6165 http://ftp.pdbj.org/pub/emdb/structures/EMD-6165 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6165 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6165 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6165_validation.pdf.gz emd_6165_validation.pdf.gz | 78.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6165_full_validation.pdf.gz emd_6165_full_validation.pdf.gz | 77.9 KB | 表示 | |
| XML形式データ |  emd_6165_validation.xml.gz emd_6165_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6165 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6165 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6165 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6165 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6165.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6165.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Cmr1-6 with crRNA and target RNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.78 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cmr complex with crRNA and target RNA
| 全体 | 名称: Cmr complex with crRNA and target RNA |
|---|---|
| 要素 |
|
-超分子 #1000: Cmr complex with crRNA and target RNA
| 超分子 | 名称: Cmr complex with crRNA and target RNA / タイプ: sample / ID: 1000 集合状態: 1 Cmr1 : 1 Cmr2 : 1 Cmr3 : 4 Cmr4 : 3 Cmr5 : 1 Cmr6 : 1 crRNA : 1 target RNA Number unique components: 8 |
|---|
-分子 #1: Cmr1
| 分子 | 名称: Cmr1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 組換発現 | 生物種:  |
-分子 #2: Cmr2
| 分子 | 名称: Cmr2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 組換発現 | 生物種:  |
-分子 #3: Cmr3
| 分子 | 名称: Cmr3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 組換発現 | 生物種:  |
-分子 #4: Cmr4
| 分子 | 名称: Cmr4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 4 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 組換発現 | 生物種:  |
-分子 #5: Cmr5
| 分子 | 名称: Cmr5 / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 組換発現 | 生物種:  |
-分子 #6: Cmr6
| 分子 | 名称: Cmr6 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 組換発現 | 生物種:  |
-分子 #7: crRNA
| 分子 | 名称: crRNA / タイプ: rna / ID: 7 / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
-分子 #8: target RNA
| 分子 | 名称: target RNA / タイプ: rna / ID: 8 / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: 20 mM Tris-HCl, pH 7.4, 500 mM NaCl, 5% v/v glycerol, 5 mM beta-mercaptoethanol |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: 2% w/v uranyl formate |
| グリッド | 詳細: 400 mesh copper grid with thin carbon support |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 150,000 times magnification. Legacy - Electron beam tilt params: 0 |
| 日付 | 2013年11月13日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 4478 / 平均電子線量: 15 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)