+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4907 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Molybdenum storage protein under turnover conditions | |||||||||
 マップデータ マップデータ | molybdenum storage protein under turnover conditions | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Molybdenum storage protein subunit alpha/beta / nutrient reservoir activity / molybdenum ion binding / Aspartate/glutamate/uridylate kinase /  Amino acid kinase family / Acetylglutamate kinase-like superfamily / Amino acid kinase family / Acetylglutamate kinase-like superfamily /  細胞質 / Molybdenum storage protein subunit beta / Molybdenum storage protein subunit alpha 細胞質 / Molybdenum storage protein subunit beta / Molybdenum storage protein subunit alpha 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Azotobacter vinelandii DJ (窒素固定) / Azotobacter vinelandii DJ (窒素固定) /   Azotobacter vinelandii (strain DJ / ATCC BAA-1303) (窒素固定) Azotobacter vinelandii (strain DJ / ATCC BAA-1303) (窒素固定) | |||||||||
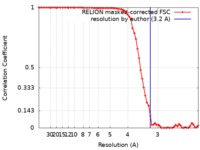
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Bruenle S / Mills DJ / Vonck J / Ermler U | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2019 ジャーナル: Proc Natl Acad Sci U S A / 年: 2019タイトル: Molybdate pumping into the molybdenum storage protein via an ATP-powered piercing mechanism. 著者: Steffen Brünle / Martin L Eisinger / Juliane Poppe / Deryck J Mills / Julian D Langer / Janet Vonck / Ulrich Ermler /  要旨: The molybdenum storage protein (MoSto) deposits large amounts of molybdenum as polyoxomolybdate clusters in a heterohexameric (αβ) cage-like protein complex under ATP consumption. Here, we suggest ...The molybdenum storage protein (MoSto) deposits large amounts of molybdenum as polyoxomolybdate clusters in a heterohexameric (αβ) cage-like protein complex under ATP consumption. Here, we suggest a unique mechanism for the ATP-powered molybdate pumping process based on X-ray crystallography, cryoelectron microscopy, hydrogen-deuterium exchange mass spectrometry, and mutational studies of MoSto from . First, we show that molybdate, ATP, and Mg consecutively bind into the open ATP-binding groove of the β-subunit, which thereafter becomes tightly locked by fixing the previously disordered N-terminal arm of the α-subunit over the β-ATP. Next, we propose a nucleophilic attack of molybdate onto the γ-phosphate of β-ATP, analogous to the similar reaction of the structurally related UMP kinase. The formed instable phosphoric-molybdic anhydride becomes immediately hydrolyzed and, according to the current data, the released and accelerated molybdate is pressed through the cage wall, presumably by turning aside the Metβ149 side chain. A structural comparison between MoSto and UMP kinase provides valuable insight into how an enzyme is converted into a molecular machine during evolution. The postulated direct conversion of chemical energy into kinetic energy via an activating molybdate kinase and an exothermic pyrophosphatase reaction to overcome a proteinous barrier represents a novelty in ATP-fueled biochemistry, because normally, ATP hydrolysis initiates large-scale conformational changes to drive a distant process. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4907.map.gz emd_4907.map.gz | 28.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4907-v30.xml emd-4907-v30.xml emd-4907.xml emd-4907.xml | 21.6 KB 21.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4907_fsc.xml emd_4907_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4907.png emd_4907.png | 215.5 KB | ||
| マスクデータ |  emd_4907_msk_1.map emd_4907_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_4907_half_map_1.map.gz emd_4907_half_map_1.map.gz emd_4907_half_map_2.map.gz emd_4907_half_map_2.map.gz | 23.3 MB 23.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4907 http://ftp.pdbj.org/pub/emdb/structures/EMD-4907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4907 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4907.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4907.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | molybdenum storage protein under turnover conditions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4907_msk_1.map emd_4907_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: MoSto half map 1
| ファイル | emd_4907_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MoSto half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: MoSto half map 2
| ファイル | emd_4907_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MoSto half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dimer of A3B3 heterohexamer of molybdenum storage protein
| 全体 | 名称: Dimer of A3B3 heterohexamer of molybdenum storage protein |
|---|---|
| 要素 |
|
-超分子 #1: Dimer of A3B3 heterohexamer of molybdenum storage protein
| 超分子 | 名称: Dimer of A3B3 heterohexamer of molybdenum storage protein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 分子量 | 理論値: 380 KDa |
-分子 #1: Molybdenum storage protein subunit alpha
| 分子 | 名称: Molybdenum storage protein subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Azotobacter vinelandii (strain DJ / ATCC BAA-1303) (窒素固定) Azotobacter vinelandii (strain DJ / ATCC BAA-1303) (窒素固定) |
| 分子量 | 理論値: 29.376773 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MTDTTNSIKH VISPLARQTL QDRDLTRPVA GKRPIRLLPW LQVVKIGGRV MDRGADAILP LVEELRKLLP EHRLLILTGA GVRARHVFS VGLDLGLPVG SLAPLAASEA GQNGHILAAM LASEGVSYVE HPTVADQLAI HLSATRAVVG SAFPPYHHHE F PGSRIPPH ...文字列: MTDTTNSIKH VISPLARQTL QDRDLTRPVA GKRPIRLLPW LQVVKIGGRV MDRGADAILP LVEELRKLLP EHRLLILTGA GVRARHVFS VGLDLGLPVG SLAPLAASEA GQNGHILAAM LASEGVSYVE HPTVADQLAI HLSATRAVVG SAFPPYHHHE F PGSRIPPH RADTGAFLLA DAFGAAGLTI VENVDGIYTA DPNGPDRGQA RFLPETSATD LAKSEGPLPV DRALLDVMAT AR HIERVQV VNGLVPGRLT AALRGEHVGT LIRTGVRPA |
-分子 #2: Molybdenum storage protein subunit beta
| 分子 | 名称: Molybdenum storage protein subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Azotobacter vinelandii (strain DJ / ATCC BAA-1303) (窒素固定) Azotobacter vinelandii (strain DJ / ATCC BAA-1303) (窒素固定) |
| 分子量 | 理論値: 28.378775 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MANSTAELEE LLMQRSLTDP QLQAAAAAAA DFRILPDATV IKIGGQSVID RGRAAVYPLV DEIVAARKNH KLLIGTGAGT RARHLYSIA AGLGLPAGVL AQLGSSVADQ NAAMLGQLLA KHGIPVVGGA GLSAVPLSLA EVNAVVFSGM PPYKLWMRPA A EGVIPPYR ...文字列: MANSTAELEE LLMQRSLTDP QLQAAAAAAA DFRILPDATV IKIGGQSVID RGRAAVYPLV DEIVAARKNH KLLIGTGAGT RARHLYSIA AGLGLPAGVL AQLGSSVADQ NAAMLGQLLA KHGIPVVGGA GLSAVPLSLA EVNAVVFSGM PPYKLWMRPA A EGVIPPYR TDAGCFLLAE QFGCKQMIFV KDEDGLYTAN PKTSKDATFI PRISVDEMKA KGLHDSILEF PVLDLLQSAQ HV REVQVVN GLVPGNLTRA LAGEHVGTII TAS |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 12 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: bis(mu4-oxo)-tetrakis(mu3-oxo)-hexakis(mu2-oxo)-hexadecaoxo-octam...
| 分子 | 名称: bis(mu4-oxo)-tetrakis(mu3-oxo)-hexakis(mu2-oxo)-hexadecaoxo-octamolybdenum (VI) タイプ: ligand / ID: 5 / コピー数: 12 / 式: 8M0 |
|---|---|
| 分子量 | 理論値: 1.215503 KDa |
| Chemical component information |  ChemComp-8M0: |
-分子 #6: oxidanyl-[[2,2,4,4,4-pentakis($l^{1}-oxidanyl)-1-(oxidanylmolybde...
| 分子 | 名称: oxidanyl-[[2,2,4,4,4-pentakis($l^{1}-oxidanyl)-1-(oxidanylmolybdenio)-1$l^{3},3-dioxa-2$l^{5},4$l^{5}-dimolybdacyclobut-2-yl]oxy]molybdenum タイプ: ligand / ID: 6 / コピー数: 12 / 式: J8E |
|---|---|
| 分子量 | 理論値: 545.77 Da |
| Chemical component information |  ChemComp-J8E: |
-分子 #7: MOLYBDATE ION
| 分子 | 名称: MOLYBDATE ION / タイプ: ligand / ID: 7 / コピー数: 42 / 式: MOO |
|---|---|
| 分子量 | 理論値: 159.938 Da |
| Chemical component information |  ChemComp-MOO: |
-分子 #8: MO(VI)(=O)(OH)2 CLUSTER
| 分子 | 名称: MO(VI)(=O)(OH)2 CLUSTER / タイプ: ligand / ID: 8 / コピー数: 6 / 式: OMO |
|---|---|
| 分子量 | 理論値: 145.954 Da |
| Chemical component information |  ChemComp-OMO: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 6.5 / 構成要素 - 濃度: 50.0 mM / 構成要素 - 名称: MOPS/NaOH 詳細: 1 mM molybdate and 1 mM mg-ATP were added before vitrification. |
| グリッド | モデル: C-flat / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 0.9 µm / 倍率(補正後): 45045 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 30000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 30000 |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: JEOL / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 1238 / 平均露光時間: 8.0 sec. / 平均電子線量: 54.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X