+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4404 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | eIF2B:eIF2 complex, phosphorylated on eIF2 alpha serine 52. | |||||||||||||||
 マップデータ マップデータ | eIF2B:eIF2 complex with eIF2 alpha phosphorylated on serine 52 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Translational control eIF2 phosphorylation Integrated stress response eIF2B TRANSLATION / TRANSLATION | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of cellular response to amino acid starvation / positive regulation of cellular response to heat / Recycling of eIF2:GDP / Cellular response to mitochondrial stress / eukaryotic translation initiation factor 2B complex / ABC-family proteins mediated transport / methionyl-initiator methionine tRNA binding / eukaryotic translation initiation factor 2 complex / cytoplasmic translational initiation / multi-eIF complex ...negative regulation of cellular response to amino acid starvation / positive regulation of cellular response to heat / Recycling of eIF2:GDP / Cellular response to mitochondrial stress / eukaryotic translation initiation factor 2B complex / ABC-family proteins mediated transport / methionyl-initiator methionine tRNA binding / eukaryotic translation initiation factor 2 complex / cytoplasmic translational initiation / multi-eIF complex / eukaryotic 43S preinitiation complex / guanyl-nucleotide exchange factor complex / positive regulation of cellular response to amino acid starvation / formation of translation preinitiation complex / eukaryotic 48S preinitiation complex / positive regulation of translational fidelity / protein-synthesizing GTPase / regulation of translational initiation / Formation of the ternary complex, and subsequently, the 43S complex / Translation initiation complex formation / Ribosomal scanning and start codon recognition / L13a-mediated translational silencing of Ceruloplasmin expression / positive regulation of translational initiation / enzyme regulator activity / translation initiation factor binding / translation initiation factor activity / guanyl-nucleotide exchange factor activity / translational initiation / cytoplasmic stress granule / ribosome binding / ribosome / GTPase activity / mRNA binding / GTP binding / mitochondrion / RNA binding / zinc ion binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   | |||||||||||||||
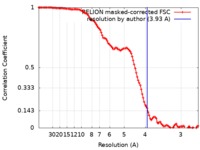
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.93 Å | |||||||||||||||
 データ登録者 データ登録者 | Adomavicius T / Roseman AM | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 | ジャーナル: Elife / 年: 2017 タイトル: Fail-safe control of translation initiation by dissociation of eIF2α phosphorylated ternary complexes. 著者: Martin D Jennings / Christopher J Kershaw / Tomas Adomavicius / Graham D Pavitt /  要旨: Phosphorylation of eIF2α controls translation initiation by restricting the levels of active eIF2-GTP/Met-tRNAi ternary complexes (TC). This modulates the expression of all eukaryotic mRNAs and ...Phosphorylation of eIF2α controls translation initiation by restricting the levels of active eIF2-GTP/Met-tRNAi ternary complexes (TC). This modulates the expression of all eukaryotic mRNAs and contributes to the cellular integrated stress response. Key to controlling the activity of eIF2 are translation factors eIF2B and eIF5, thought to primarily function with eIF2-GDP and TC respectively. Using a steady-state kinetics approach with purified proteins we demonstrate that eIF2B binds to eIF2 with equal affinity irrespective of the presence or absence of competing guanine nucleotides. We show that eIF2B can compete with Met-tRNAi for eIF2-GTP and can destabilize TC. When TC is formed with unphosphorylated eIF2, eIF5 can out-compete eIF2B to stabilize TC/eIF5 complexes. However when TC/eIF5 is formed with phosphorylated eIF2, eIF2B outcompetes eIF5 and destabilizes TC. These data uncover competition between eIF2B and eIF5 for TC and identify that phosphorylated eIF2-GTP translation initiation intermediate complexes can be inhibited by eIF2B. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4404.map.gz emd_4404.map.gz | 117.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4404-v30.xml emd-4404-v30.xml emd-4404.xml emd-4404.xml | 37.5 KB 37.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4404_fsc.xml emd_4404_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4404.png emd_4404.png | 218.6 KB | ||
| マスクデータ |  emd_4404_msk_1.map emd_4404_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-4404.cif.gz emd-4404.cif.gz | 10.2 KB | ||
| その他 |  emd_4404_additional.map.gz emd_4404_additional.map.gz emd_4404_half_map_1.map.gz emd_4404_half_map_1.map.gz emd_4404_half_map_2.map.gz emd_4404_half_map_2.map.gz | 58.4 MB 97.1 MB 97.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4404 http://ftp.pdbj.org/pub/emdb/structures/EMD-4404 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4404 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4404 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4404.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4404.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | eIF2B:eIF2 complex with eIF2 alpha phosphorylated on serine 52 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
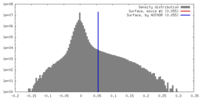
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4404_msk_1.map emd_4404_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
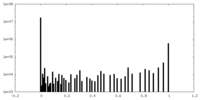
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: LocScale version of main map
| ファイル | emd_4404_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocScale version of main map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: relion half-map 1
| ファイル | emd_4404_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | relion half-map 1 | ||||||||||||
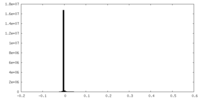
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: relion half-map 2
| ファイル | emd_4404_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | relion half-map 2 | ||||||||||||
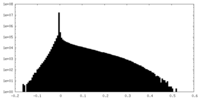
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of translation initiation factors eIF2 and eIF2B
| 全体 | 名称: Complex of translation initiation factors eIF2 and eIF2B |
|---|---|
| 要素 |
|
-超分子 #1: Complex of translation initiation factors eIF2 and eIF2B
| 超分子 | 名称: Complex of translation initiation factors eIF2 and eIF2B タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 838 KDa |
-分子 #1: Translation initiation factor eIF-2B subunit alpha
| 分子 | 名称: Translation initiation factor eIF-2B subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.062027 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSEFNITETY LRFLEEDTEM TMPIAAIEAL VTLLRIKTPE TAAEMINTIK SSTEELIKSI PNSVSLRAGC DIFMRFVLRN LHLYGDWEN CKQHLIENGQ LFVSRAKKSR NKIAEIGVDF IADDDIILVH GYSRAVFSLL NHAANKFIRF RCVVTESRPS K QGNQLYTL ...文字列: MSEFNITETY LRFLEEDTEM TMPIAAIEAL VTLLRIKTPE TAAEMINTIK SSTEELIKSI PNSVSLRAGC DIFMRFVLRN LHLYGDWEN CKQHLIENGQ LFVSRAKKSR NKIAEIGVDF IADDDIILVH GYSRAVFSLL NHAANKFIRF RCVVTESRPS K QGNQLYTL LEQKGIPVTL IVDSAVGAVI DKVDKVFVGA EGVAESGGII NLVGTYSVGV LAHNARKPFY VVTESHKFVR MF PLSSDDL PMAGPPLDFT RRTDDLEDAL RGPTIDYTAQ EYITALITDL GVLTPSAVSE ELIKMWYD UniProtKB: Translation initiation factor eIF2B subunit alpha |
-分子 #2: Translation initiation factor eIF-2B subunit delta
| 分子 | 名称: Translation initiation factor eIF-2B subunit delta / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 70.945195 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSESEAKSRS ATPPSKAKQA TPTTTAAANG EKKLTNKELK ELKKQEKAAK RAAMKQANGI SIEQQQQQAQ MKKEKKQLQR EQQQKREQK QKNANKKKQN ERNVKKSTLF GHLETTEERR ATILALTSAV SSPKTSRITA AGLMVPVVAS ALSGSNVLTA S SLMPVGPN ...文字列: MSESEAKSRS ATPPSKAKQA TPTTTAAANG EKKLTNKELK ELKKQEKAAK RAAMKQANGI SIEQQQQQAQ MKKEKKQLQR EQQQKREQK QKNANKKKQN ERNVKKSTLF GHLETTEERR ATILALTSAV SSPKTSRITA AGLMVPVVAS ALSGSNVLTA S SLMPVGPN ASSTVSASAP ASTTTTLPAS SAALSAGTSS ASTNTPTAIQ QEIASSNASD VAKTLASISL EAGEFNVIPG IS SVIPTVL EQSFDNSSLI SSVKELLLNK DLIHPSILLL TSHLAHYKIV GSIPRCIAML EVFQIVIKDY QTPKGTTLSR NLT SYLSHQ IDLLKKARPL SVTMGNAIRW LKQEISLIDP STPDKAAKKD LCEKIGQFAK EKIELADQLI IDNASTQIEE STTI VTYGS SKVLTELLLH NAISLKKNIK VIVVDSRPLF EGRKMAETLR NAGVNVMYAL ITSLDTIFNM DVDYVFLGAH SILSN GFLY SRAGTAMLAM SAKRRNIPVL VCCESLKFSQ RVQLDSVTFN ELADPNDLVN IDYENPVERR GNKGALLNQF IKERKF EKK KLAMENKPKG NKIGGKKGSE GESKDASNEE DSNSKNILDG WQELPSLNIV NILYDLTPPE YIKKVITEFG ALPPSSV PV ILREYKGSA UniProtKB: Translation initiation factor eIF2B subunit delta |
-分子 #3: Translation initiation factor eIF-2B subunit beta
| 分子 | 名称: Translation initiation factor eIF-2B subunit beta / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.621441 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSQAFTSVH PNAATSDVNV TIDTFVAKLK RRQVQGSYAI ALETLQLLMR FISAARWNHV NDLIEQIRDL GNSLEKAHPT AFSCGNVIR RILAVLRDEV EEDTMSTTVT STSVAEPLIS SMFNLLQKPE QPHQNRKNSS GSSSMKTKTD YRQVAIQGIK D LIDEIKNI ...文字列: MSSQAFTSVH PNAATSDVNV TIDTFVAKLK RRQVQGSYAI ALETLQLLMR FISAARWNHV NDLIEQIRDL GNSLEKAHPT AFSCGNVIR RILAVLRDEV EEDTMSTTVT STSVAEPLIS SMFNLLQKPE QPHQNRKNSS GSSSMKTKTD YRQVAIQGIK D LIDEIKNI DEGIQQIAID LIHDHEILLT PTPDSKTVLK FLITARERSN RTFTVLVTEG FPNNTKNAHE FAKKLAQHNI ET LVVPDSA VFALMSRVGK VIIGTKAVFV NGGTISSNSG VSSVCECARE FRTPVFAVAG LYKLSPLYPF DVEKFVEFGG SQR ILPRMD PRKRLDTVNQ ITDYVPPENI DIYITNVGGF NPSFIYRIAW DNYKQIDVHL DKNKA UniProtKB: Translation initiation factor eIF2B subunit beta |
-分子 #4: Translation initiation factor eIF-2B subunit epsilon
| 分子 | 名称: Translation initiation factor eIF-2B subunit epsilon タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 81.249062 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGKKGQKKS GLGNHGKNSD MDVEDRLQAV VLTDSYETRF MPLTAVKPRC LLPLANVPLI EYTLEFLAKA GVHEVFLICS SHANQINDY IENSKWNLPW SPFKITTIMS PEARCTGDVM RDLDNRGIIT GDFILVSGDV LTNIDFSKML EFHKKMHLQD K DHISTMCL ...文字列: MAGKKGQKKS GLGNHGKNSD MDVEDRLQAV VLTDSYETRF MPLTAVKPRC LLPLANVPLI EYTLEFLAKA GVHEVFLICS SHANQINDY IENSKWNLPW SPFKITTIMS PEARCTGDVM RDLDNRGIIT GDFILVSGDV LTNIDFSKML EFHKKMHLQD K DHISTMCL SKASTYPKTR TIEPAAFVLD KSTSRCIYYQ DLPLPSSREK TSIQIDPELL DNVDEFVIRN DLIDCRIDIC TS HVPLIFQ ENFDYQSLRT DFVKGVISSD ILGKHIYAYL TDEYAVRVES WQTYDTISQD FLGRWCYPLV LDSNIQDDQT YSY ESRHIY KEKDVVLAQS CKIGKCTAIG SGTKIGEGTK IENSVIGRNC QIGENIRIKN SFIWDDCIIG NNSIIDHSLI ASNA TLGSN VRLNDGCIIG FNVKIDDNMD LDRNTKISAS PLKNAGSRMY DNESNEQFDQ DLDDQTLAVS IVGDKGVGYI YESEV SDDE DSSTEACKEI NTLSNQLDEL YLSDDSISSA TKKTKKRRTM SVNSIYTDRE EIDSEFEDED FEKEGIATVE RAMENN HDL DTALLELNTL RMSMNVTYHE VRIATITALL RRVYHFIATQ TLGPKDAVVK VFNQWGLLFK RQAFDEEEYI DLMNIIM EK IVEQSFDKPD LILFSALVSL YDNDIIEEDV IYKWWDNVST DPRYDEVKKL TVKWVEWLQN ADEESSSEEE UniProtKB: Translation initiation factor eIF2B subunit epsilon |
-分子 #5: Translation initiation factor eIF-2B subunit gamma
| 分子 | 名称: Translation initiation factor eIF-2B subunit gamma / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 65.76832 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSIQAFVFCG KGSNLAPFTQ PDFPFQTQNK DSTAATSGDK LNELVNSALD STVINEFMQH STRLPKALLP IGNRPMIEYV LDWCDQADF KEISVVAPVD EIELIESGLT SFLSLRKQQF ELIYKALSNS NHSHHLQDPK KINFIPSKAN STGESLQKEL L PRINGDFV ...文字列: MSIQAFVFCG KGSNLAPFTQ PDFPFQTQNK DSTAATSGDK LNELVNSALD STVINEFMQH STRLPKALLP IGNRPMIEYV LDWCDQADF KEISVVAPVD EIELIESGLT SFLSLRKQQF ELIYKALSNS NHSHHLQDPK KINFIPSKAN STGESLQKEL L PRINGDFV ILPCDFVTDI PPQVLVDQFR NRDDNNLAMT IYYKNSLDSS IDKKQQQKQK QQQFFTVYSE NEDSERQPIL LD VYSQRDV TKTKYLQIRS HLLWNYPNLT VSTKLLNSFI YFCSFELCQL LKLGPQSMSR QASFKDPFTG NQQQQNPPTT DDD EDRNHD DDDDYKPSAT SIQPTYFKKK NDLILDPINC NKSLSKVFRD LSRRSWQHSK PREPIGIFIL PNETLFIRAN NLNA YMDAN RFVLKIKSQT MFTKNIQIQS AAIGADAIVD PKCQISAHSN VKMSVLGTQA NIGSRCRVAG SLLFPGVHLG DEVIL ENCI IGPMAKIGSK CKLSNCYIEG HYVVEPKNNF KGETLANVYL DEDEEDELIY DDSVIAGESE IAEETDSDDR SDEDSD DSE YTDEYEYEDD GLFER UniProtKB: Translation initiation factor eIF2B subunit gamma |
-分子 #6: Eukaryotic translation initiation factor 2 subunit alpha
| 分子 | 名称: Eukaryotic translation initiation factor 2 subunit alpha タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.843633 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSTSHCRFYE NKYPEIDDIV MVNVQQIAEM GAYVKLLEYD NIEGMILLSE L(SEP)RRRIRSIQ KLIRVGKNDV AVVLRV DKE KGYIDLSKRR VSSEDIIKCE EKYQKSKTVH SILRYCAEKF QIPLEELYKT IAWPLSRKFG HAYEAFKLSI IDETVWE GI ...文字列: MSTSHCRFYE NKYPEIDDIV MVNVQQIAEM GAYVKLLEYD NIEGMILLSE L(SEP)RRRIRSIQ KLIRVGKNDV AVVLRV DKE KGYIDLSKRR VSSEDIIKCE EKYQKSKTVH SILRYCAEKF QIPLEELYKT IAWPLSRKFG HAYEAFKLSI IDETVWE GI EPPSKDVLDE LKNYISKRLT PQAVKIRADV EVSCFSYEGI DAIKDALKSA EDMSTEQMQV KVKLVAAPLY VLTTQALD K QKGIEQLESA IEKITEVITK YGGVCNITMP PKAVTATEDA ELQALLESKE LDNRSDSEDD EDESDDE UniProtKB: Eukaryotic translation initiation factor 2 subunit alpha |
-分子 #7: Eukaryotic translation initiation factor 2 subunit beta
| 分子 | 名称: Eukaryotic translation initiation factor 2 subunit beta タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 31.631309 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSDLAAELG FDPALKKKKK TKKVIPDDFD AAVNGKENGS GDDLFAGLKK KKKKSKSVSA DAEAEKEPTD DIAEALGELS LKKKKKKTK DSSVDAFEKE LAKAGLDNVD AESKEGTPSA NSSIQQEVGL PYSELLSRFF NILRTNNPEL AGDRSGPKFR I PPPVCLRD ...文字列: MSSDLAAELG FDPALKKKKK TKKVIPDDFD AAVNGKENGS GDDLFAGLKK KKKKSKSVSA DAEAEKEPTD DIAEALGELS LKKKKKKTK DSSVDAFEKE LAKAGLDNVD AESKEGTPSA NSSIQQEVGL PYSELLSRFF NILRTNNPEL AGDRSGPKFR I PPPVCLRD GKKTIFSNIQ DIAEKLHRSP EHLIQYLFAE LGTSGSVDGQ KRLVIKGKFQ SKQMENVLRR YILEYVTCKT CK SINTELK REQSNRLFFM VCKSCGSTRS VSSIKTGFQA TVGKRRRM UniProtKB: Eukaryotic translation initiation factor 2 subunit beta |
-分子 #8: Eukaryotic translation initiation factor 2 subunit gamma
| 分子 | 名称: Eukaryotic translation initiation factor 2 subunit gamma タイプ: protein_or_peptide / ID: 8 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.942699 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSDLQDQEPS IIINGNLEPV GEPDIVEETE VVAQETQETQ DADKPKKKVA FTGLEEDGET EEEKRKREFE EGGGLPEQPL NPDFSKLNP LSAEIINRQA TINIGTIGHV AHGKSTVVRA ISGVQTVRFK DELERNITIK LGYANAKIYK CQEPTCPEPD C YRSFKSDK ...文字列: MSDLQDQEPS IIINGNLEPV GEPDIVEETE VVAQETQETQ DADKPKKKVA FTGLEEDGET EEEKRKREFE EGGGLPEQPL NPDFSKLNP LSAEIINRQA TINIGTIGHV AHGKSTVVRA ISGVQTVRFK DELERNITIK LGYANAKIYK CQEPTCPEPD C YRSFKSDK EISPKCQRPG CPGRYKLVRH VSFVDCPGHD ILMSTMLSGA AVMDAALLLI AGNESCPQPQ TSEHLAAIEI MK LKHVIIL QNKVDLMREE SALEHQKSIL KFIRGTIADG APIVPISAQL KYNIDAVNEF IVKTIPVPPR DFMISPRLIV IRS FDVNKP GAEIEDLKGG VAGGSILNGV FKLGDEIEIR PGIVTKDDKG KIQCKPIFSN IVSLFAEQND LKFAVPGGLI GVGT KVDPT LCRADRLVGQ VVGAKGHLPN IYTDIEINYF LLRRLLGVKT DGQKQAKVRK LEPNEVLMVN IGSTATGARV VAVKA DMAR LQLTSPACTE INEKIALSRR IEKHWRLIGW ATIKKGTTLE PIA UniProtKB: Eukaryotic translation initiation factor 2 subunit gamma |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Solutions for sample preparation were made fresh, filter sterilized, and degassed. | ||||||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: LACEY / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: CARBON / 支持フィルム - #1 - トポロジー: CONTINUOUS / 支持フィルム - #1 - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa 詳細: Ultra-thin carbon support film, 3nm - on lacey carbon grids from Agar Scientific were used for 35 degree tilted data collection. For zero tilt data collection, 200 mesh Au Quantifoil, R2/2 grids were used. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 294 K / 装置: FEI VITROBOT MARK III / 詳細: Blot for 2 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | Tilt at zero and 35 degrees. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 2-48 / 撮影したグリッド数: 2 / 実像数: 4533 / 平均露光時間: 12.0 sec. / 平均電子線量: 40.0 e/Å2 詳細: 2 separate data collections for zero degree (2255 images) and tilted (2278 images) specimen. Both on Titan Krios |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 5.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 37313 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 詳細 | Modeller was used to build homology model of S. cerevisiae eIF2B structure based on S. pombe crystal structure. Subunits of the homology model, along with eIF2 alpha domains 1 and 2, were then rigid body fitted into our map using UCSF Chimera. The model then was refined using phenix and manually adjusted in Coot. | ||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: correlation coeficient | ||||||
| 得られたモデル |  PDB-6i3m: |
-原子モデル構築 2
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | eIF2 alpha domain 3, eIF2 gamma, and eIF2 beta (3JAP) were rigid body fitted into our map using Chimera. | ||||||||
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient | ||||||||
| 得られたモデル |  PDB-6i3m: |
 ムービー
ムービー コントローラー
コントローラー































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)