+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of OSCA1.2-liposome-inside-out closed state | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | OSCA/TMEM63 channel / mechanosensitive channel /  PLANT PROTEIN (植物) PLANT PROTEIN (植物) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mechanosensitive monoatomic ion channel activity / calcium-activated cation channel activity / monoatomic cation transport / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.56 Å クライオ電子顕微鏡法 / 解像度: 3.56 Å | ||||||||||||
 データ登録者 データ登録者 | Zhang Y / Han Y | ||||||||||||
| 資金援助 |  中国, 中国,  オーストラリア, 3件 オーストラリア, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Mechanical activation opens a lipid-lined pore in OSCA ion channels. 著者: Yaoyao Han / Zijing Zhou / Ruitao Jin / Fei Dai / Yifan Ge / Xisan Ju / Xiaonuo Ma / Sitong He / Ling Yuan / Yingying Wang / Wei Yang / Xiaomin Yue / Zhongwen Chen / Yadong Sun / Ben Corry / ...著者: Yaoyao Han / Zijing Zhou / Ruitao Jin / Fei Dai / Yifan Ge / Xisan Ju / Xiaonuo Ma / Sitong He / Ling Yuan / Yingying Wang / Wei Yang / Xiaomin Yue / Zhongwen Chen / Yadong Sun / Ben Corry / Charles D Cox / Yixiao Zhang /   要旨: OSCA/TMEM63 channels are the largest known family of mechanosensitive channels, playing critical roles in plant and mammalian mechanotransduction. Here we determined 44 cryogenic electron microscopy ...OSCA/TMEM63 channels are the largest known family of mechanosensitive channels, playing critical roles in plant and mammalian mechanotransduction. Here we determined 44 cryogenic electron microscopy structures of OSCA/TMEM63 channels in different environments to investigate the molecular basis of OSCA/TMEM63 channel mechanosensitivity. In nanodiscs, we mimicked increased membrane tension and observed a dilated pore with membrane access in one of the OSCA1.2 subunits. In liposomes, we captured the fully open structure of OSCA1.2 in the inside-in orientation, in which the pore shows a large lateral opening to the membrane. Unusually for ion channels, structural, functional and computational evidence supports the existence of a 'proteo-lipidic pore' in which lipids act as a wall of the ion permeation pathway. In the less tension-sensitive homologue OSCA3.1, we identified an 'interlocking' lipid tightly bound in the central cleft, keeping the channel closed. Mutation of the lipid-coordinating residues induced OSCA3.1 activation, revealing a conserved open conformation of OSCA channels. Our structures provide a global picture of the OSCA channel gating cycle, uncover the importance of bound lipids and show that each subunit can open independently. This expands both our understanding of channel-mediated mechanotransduction and channel pore formation, with important mechanistic implications for the TMEM16 and TMC protein families. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38503.map.gz emd_38503.map.gz | 35.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38503-v30.xml emd-38503-v30.xml emd-38503.xml emd-38503.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_38503.png emd_38503.png | 103.3 KB | ||
| Filedesc metadata |  emd-38503.cif.gz emd-38503.cif.gz | 5.7 KB | ||
| その他 |  emd_38503_half_map_1.map.gz emd_38503_half_map_1.map.gz emd_38503_half_map_2.map.gz emd_38503_half_map_2.map.gz | 42.2 MB 42.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38503 http://ftp.pdbj.org/pub/emdb/structures/EMD-38503 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38503 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38503 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8xngMC  8xajC  8xryC  8xs0C  8xs4C  8xs5C  8xvxC  8xvyC  8xvzC  8xw0C  8xw1C  8xw2C  8xw3C  8xw4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38503.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38503.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.055 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_38503_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
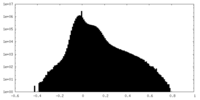
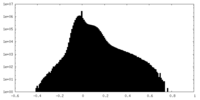
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_38503_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
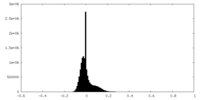
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of OSCA1.2-liposome-inside-out closed state
| 全体 | 名称: Cryo-EM structure of OSCA1.2-liposome-inside-out closed state |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of OSCA1.2-liposome-inside-out closed state
| 超分子 | 名称: Cryo-EM structure of OSCA1.2-liposome-inside-out closed state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
-分子 #1: Calcium permeable stress-gated cation channel 1
| 分子 | 名称: Calcium permeable stress-gated cation channel 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 89.003664 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MATLQDIGVS AGINILSAFV FFIIFAVLRL QPFNDRVYFS KWYLKGLRSS PARGGAFAQR FVNLDFRSYM KFLNWMPEAL KMPEPELID HAGLDSVVYL RIYWLGLKIF TPIAVLAWAV LVPVNWTNNT LEMAKQLRNV TSSDIDKLSV SNIPEYSMRF W THIVMAYA ...文字列: MATLQDIGVS AGINILSAFV FFIIFAVLRL QPFNDRVYFS KWYLKGLRSS PARGGAFAQR FVNLDFRSYM KFLNWMPEAL KMPEPELID HAGLDSVVYL RIYWLGLKIF TPIAVLAWAV LVPVNWTNNT LEMAKQLRNV TSSDIDKLSV SNIPEYSMRF W THIVMAYA FTIWTCYVLM KEYETIANMR LQFVASEARR PDQFTVLVRN VPPDADESVS ELVEHFFLVN HPDHYLTHQV VC NANKLAD LVKKKKKLQN WLDYYQLKYA RNNSQRIMVK LGFLGLWGQK VDAIEHYIAE IDKISKEISK EREEVVNDPK AIM PAAFVS FKTRWAAAVC AQTQQTRNPT QWLTEWAPEP RDVFWSNLAI PYVSLTVRRL IMHVAFFFLT FFFIVPIAFV QSLA TIEGI VKAAPFLKFI VDDKFMKSVI QGFLPGIALK LFLAFLPSIL MIMSKFEGFT SISSLERRAA FRYYIFNLVN VFLAS VIAG AAFEQLNSFL NQSANQIPKT IGVAIPMKAT FFITYIMVDG WAGVAGEILM LKPLIMFHLK NAFLVKTDKD REEAMD PGS IGFNTGEPRI QLYFLLGLVY APVTPMLLPF ILVFFALAYI VYRHQIINVY NQEYESAAAF WPDVHGRVIA ALVISQL LL MGLLGTKHAA LAAPFLIALP VLTIGFHHFC KGRYEPAFIR YPLQEAMMKD TLETAREPNL NLKGYLQNAY VHPVFKGD E DDYDIDDKLG KFEDEAIIVP TKRQSRRNTP APSIISGDDS PSLPFSGKLV SNSLEVLFQ UniProtKB: Calcium permeable stress-gated cation channel 1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.4 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.4 µm最小 デフォーカス(公称値): 1.4000000000000001 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 49.41 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.56 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 100731 |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X