+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30545 | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
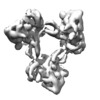
| タイトル | Cryo-EM structure of Schizosaccharomyces pombe Atg9 of trimer | ||||||||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 オートファジー / オートファジー /  : / : /  phospholipid scramblase activity / autophagy of mitochondrion / protein localization to phagophore assembly site / phagophore assembly site membrane / fungal-type vacuole membrane / phagophore assembly site / phospholipid scramblase activity / autophagy of mitochondrion / protein localization to phagophore assembly site / phagophore assembly site membrane / fungal-type vacuole membrane / phagophore assembly site /  オートファゴソーム / オートファゴソーム /  オートファジー ... オートファジー ... オートファジー / オートファジー /  : / : /  phospholipid scramblase activity / autophagy of mitochondrion / protein localization to phagophore assembly site / phagophore assembly site membrane / fungal-type vacuole membrane / phagophore assembly site / phospholipid scramblase activity / autophagy of mitochondrion / protein localization to phagophore assembly site / phagophore assembly site membrane / fungal-type vacuole membrane / phagophore assembly site /  オートファゴソーム / オートファゴソーム /  オートファジー / cytoplasmic vesicle membrane / オートファジー / cytoplasmic vesicle membrane /  オートファジー / オートファジー /  protein transport / membrane => GO:0016020 / protein transport / membrane => GO:0016020 /  ゴルジ体 / endoplasmic reticulum membrane ゴルジ体 / endoplasmic reticulum membrane類似検索 - 分子機能 | ||||||||||||||||||||||||||||||
| 生物種 |   Schizosaccharomyces pombe (分裂酵母) Schizosaccharomyces pombe (分裂酵母) | ||||||||||||||||||||||||||||||
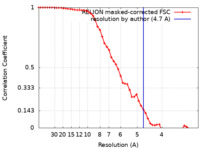
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.7 Å クライオ電子顕微鏡法 / 解像度: 4.7 Å | ||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Matoba K / Tsutsumi A | ||||||||||||||||||||||||||||||
| 資金援助 |  日本, 9件 日本, 9件
| ||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Atg9 is a lipid scramblase that mediates autophagosomal membrane expansion. 著者: Kazuaki Matoba / Tetsuya Kotani / Akihisa Tsutsumi / Takuma Tsuji / Takaharu Mori / Daisuke Noshiro / Yuji Sugita / Norimichi Nomura / So Iwata / Yoshinori Ohsumi / Toyoshi Fujimoto / Hitoshi ...著者: Kazuaki Matoba / Tetsuya Kotani / Akihisa Tsutsumi / Takuma Tsuji / Takaharu Mori / Daisuke Noshiro / Yuji Sugita / Norimichi Nomura / So Iwata / Yoshinori Ohsumi / Toyoshi Fujimoto / Hitoshi Nakatogawa / Masahide Kikkawa / Nobuo N Noda /  要旨: The molecular function of Atg9, the sole transmembrane protein in the autophagosome-forming machinery, remains unknown. Atg9 colocalizes with Atg2 at the expanding edge of the isolation membrane (IM) ...The molecular function of Atg9, the sole transmembrane protein in the autophagosome-forming machinery, remains unknown. Atg9 colocalizes with Atg2 at the expanding edge of the isolation membrane (IM), where Atg2 receives phospholipids from the endoplasmic reticulum (ER). Here we report that yeast and human Atg9 are lipid scramblases that translocate phospholipids between outer and inner leaflets of liposomes in vitro. Cryo-EM of fission yeast Atg9 reveals a homotrimer, with two connected pores forming a path between the two membrane leaflets: one pore, located at a protomer, opens laterally to the cytoplasmic leaflet; the other, at the trimer center, traverses the membrane vertically. Mutation of residues lining the pores impaired IM expansion and autophagy activity in yeast and abolished Atg9's ability to transport phospholipids between liposome leaflets. These results suggest that phospholipids delivered by Atg2 are translocated from the cytoplasmic to the luminal leaflet by Atg9, thereby driving autophagosomal membrane expansion. | ||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30545.map.gz emd_30545.map.gz | 7.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30545-v30.xml emd-30545-v30.xml emd-30545.xml emd-30545.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30545_fsc.xml emd_30545_fsc.xml | 5.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30545.png emd_30545.png | 54.5 KB | ||
| マスクデータ |  emd_30545_msk_1.map emd_30545_msk_1.map | 10.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_30545_half_map_1.map.gz emd_30545_half_map_1.map.gz emd_30545_half_map_2.map.gz emd_30545_half_map_2.map.gz | 7.9 MB 7.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30545 http://ftp.pdbj.org/pub/emdb/structures/EMD-30545 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30545 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30545 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30545.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30545.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.545 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_30545_msk_1.map emd_30545_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
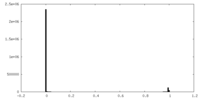
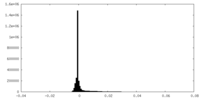
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_30545_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
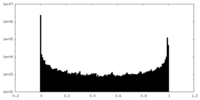
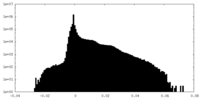
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_30545_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
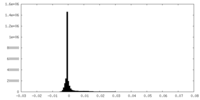
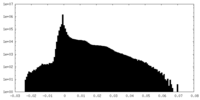
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Protein detergent complex (Atg9-LMNG ) treated with GraFix
| 全体 | 名称: Protein detergent complex (Atg9-LMNG ) treated with GraFix |
|---|---|
| 要素 |
|
-超分子 #1: Protein detergent complex (Atg9-LMNG ) treated with GraFix
| 超分子 | 名称: Protein detergent complex (Atg9-LMNG ) treated with GraFix タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Schizosaccharomyces pombe (分裂酵母) Schizosaccharomyces pombe (分裂酵母) |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) / 組換株: BJ926 / 組換プラスミド: pRS426 Saccharomyces cerevisiae (パン酵母) / 組換株: BJ926 / 組換プラスミド: pRS426 |
-分子 #1: Atg9
| 分子 | 名称: Atg9 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Schizosaccharomyces pombe (分裂酵母) Schizosaccharomyces pombe (分裂酵母) |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: MFYQPAQNKK QYDDLADIEA QNNVPNTQEV LEAWQESLDS DEDESSPLEE SNGFTISEHD DFVKSVPRKN NPTDLLYSGK LLDSDEPPS VHGNSSKVPS KHPSPSFPET TSLRNLQNGS KQKPALPNFN DPHFYNEDVT RSGHPNRSIY TQLPRNEFSN A RVLWNRLS ...文字列: MFYQPAQNKK QYDDLADIEA QNNVPNTQEV LEAWQESLDS DEDESSPLEE SNGFTISEHD DFVKSVPRKN NPTDLLYSGK LLDSDEPPS VHGNSSKVPS KHPSPSFPET TSLRNLQNGS KQKPALPNFN DPHFYNEDVT RSGHPNRSIY TQLPRNEFSN A RVLWNRLS ARDRVLWRWA NVENLDSFLQ QVYTYYTGKG LSCIIVHRLF QILTVSFVIG FTTFITSCID WPAVTPHGSL AG VTKSQCI AQMSPITYLV LWLFLSFLLA LWIYYLTDIP RLWQMREFYI HALKIATADM PTVSWQRVLY RLLKLKNVNA LTA EDGRVV SLHNMKRLDA YAIANRIMRK DNYFIALINN GIINIELPLL HRRILTHTTE WNINWCIFNF VFDEQGQLRS AFRN PNSRK RLSEELRRRF IVAGFLNCLF APIVAIYLVI HNFFRYFNEY HKNPGALSTR RYTPLALWTF REYNELQHFF DERIN DSYA AASHYVSQFP DFNMIRLFKY ISFILGSFTA ILVIITVFDP ELMVTFEITK DRSVLFYLGL FGSLIAVSRS IIPDET LVF APEKALRRVI TFTHYMPGWW SDNMHSKAVQ QEFCSLYSYR IVNLLWEILG ILLTPVLLFF TFPSCSQDIV DFFREHT IN VEGVGYVCSY AVFQDNPPYE SVASLVQSRK ISPLIQNKPE LSRIYFYEQF NTEAPRRDLR GSLEVLFQ |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.3 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER/RHODIUM / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 8.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X