+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22975 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Lethocerus Myosin II complete coiled-coil domain resolved in its native environment | |||||||||
 マップデータ マップデータ | One myosin molecule segmented out of the helically extended reconstruction | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Lethocerus indicus (タイワンタガメ) Lethocerus indicus (タイワンタガメ) | |||||||||
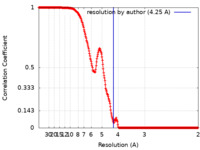
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.25 Å クライオ電子顕微鏡法 / 解像度: 4.25 Å | |||||||||
 データ登録者 データ登録者 | Rahmani H / Hu Z / Daneshparvar N / Taylor D / Taylor KA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: The myosin II coiled-coil domain atomic structure in its native environment. 著者: Hamidreza Rahmani / Wen Ma / Zhongjun Hu / Nadia Daneshparvar / Dianne W Taylor / J Andrew McCammon / Thomas C Irving / Robert J Edwards / Kenneth A Taylor /  要旨: The atomic structure of the complete myosin tail within thick filaments isolated from flight muscle is described and compared to crystal structures of recombinant, human cardiac myosin tail segments. ...The atomic structure of the complete myosin tail within thick filaments isolated from flight muscle is described and compared to crystal structures of recombinant, human cardiac myosin tail segments. Overall, the agreement is good with three exceptions: the proximal S2, in which the filament has heads attached but the crystal structure doesn't, and skip regions 2 and 4. At the head-tail junction, the tail α-helices are asymmetrically structured encompassing well-defined unfolding of 12 residues for one myosin tail, ∼4 residues of the other, and different degrees of α-helix unwinding for both tail α-helices, thereby providing an atomic resolution description of coiled-coil "uncoiling" at the head-tail junction. Asymmetry is observed in the nonhelical C termini; one C-terminal segment is intercalated between ribbons of myosin tails, the other apparently terminating at Skip 4 of another myosin tail. Between skip residues, crystal and filament structures agree well. Skips 1 and 3 also agree well and show the expected α-helix unwinding and coiled-coil untwisting in response to skip residue insertion. Skips 2 and 4 are different. Skip 2 is accommodated in an unusual manner through an increase in α-helix radius and corresponding reduction in rise/residue. Skip 4 remains helical in one chain, with the other chain unfolded, apparently influenced by the acidic myosin C terminus. The atomic model may shed some light on thick filament mechanosensing and is a step in understanding the complex roles that thick filaments of all species undergo during muscle contraction. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22975.map.gz emd_22975.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22975-v30.xml emd-22975-v30.xml emd-22975.xml emd-22975.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22975_fsc.xml emd_22975_fsc.xml | 26.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22975.png emd_22975.png | 249 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22975 http://ftp.pdbj.org/pub/emdb/structures/EMD-22975 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22975 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22975 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7kogMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10675 (タイトル: The Myosin II Coiled Coil Domain Atomic Structure in its Native Environment EMPIAR-10675 (タイトル: The Myosin II Coiled Coil Domain Atomic Structure in its Native EnvironmentData size: 18.8 TB / Data #1: Frame Stacks 1/3 [micrographs - multiframe] / Data #2: Frame Stacks 2/3 [micrographs - multiframe] / Data #3: Frame Stacks 3/3 [micrographs - multiframe] Data #4: Micrographs before frames alignment [micrographs - single frame] Data #5: Micrographs: frame-aligned and dose-weighted [micrographs - single frame] Data #6: Particle Stack [picked particles - single frame - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22975.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22975.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | One myosin molecule segmented out of the helically extended reconstruction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.98 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Lethocerus flight muscle myosin filament
| 全体 | 名称: Lethocerus flight muscle myosin filament |
|---|---|
| 要素 |
|
-超分子 #1: Lethocerus flight muscle myosin filament
| 超分子 | 名称: Lethocerus flight muscle myosin filament / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The sample is a bipolar helical structure, with helical repeat 145 Angstrom and helical turn 33.98 degree. The sample has C4 symmetry. The map contains 6 unique features: myosin molecule with ...詳細: The sample is a bipolar helical structure, with helical repeat 145 Angstrom and helical turn 33.98 degree. The sample has C4 symmetry. The map contains 6 unique features: myosin molecule with completely resolved rods, 4 resolved non-myosin densities among the myosin rods and an annular region inside of annulus occupied by myosin rods that most likely contains paramyosin. The 4 non-myosin densities may contain parts of the proteins myofilin and flightin. |
|---|---|
| 由来(天然) | 生物種:   Lethocerus indicus (タイワンタガメ) / 器官: myocyte / 組織: dorsal longitudinal indirect flight muscle / Organelle: Sarcomere / 細胞中の位置: myofibril Lethocerus indicus (タイワンタガメ) / 器官: myocyte / 組織: dorsal longitudinal indirect flight muscle / Organelle: Sarcomere / 細胞中の位置: myofibril |
-分子 #1: Myosin heavy chain isoform Mhc_X1
| 分子 | 名称: Myosin heavy chain isoform Mhc_X1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lethocerus indicus (タイワンタガメ) Lethocerus indicus (タイワンタガメ) |
| 分子量 | 理論値: 225.227562 KDa |
| 配列 | 文字列: MPGSKPTKTE EEDDPTPYLF VSLEQKRIDQ TKPYDAKKAC WVPDEHEGFV QGEIRGTKGD IVSVHLPNGE TKDFKKDQVG QVNPPKFEK CEDMSNLTYL NDASVLYNLK QRYYNKLIYT YSGLFCVAIN PYKRFPVYTM RCAKLYRGKR RNEVPPHIFA I SDGAYVNM ...文字列: MPGSKPTKTE EEDDPTPYLF VSLEQKRIDQ TKPYDAKKAC WVPDEHEGFV QGEIRGTKGD IVSVHLPNGE TKDFKKDQVG QVNPPKFEK CEDMSNLTYL NDASVLYNLK QRYYNKLIYT YSGLFCVAIN PYKRFPVYTM RCAKLYRGKR RNEVPPHIFA I SDGAYVNM LTNKENQSML ITGESGAGKT ENTKKVIAYF ATVGASTKKE EAASAASQKK GTLEDQVVQT NPVLEAFGNA KT VRNDNSS RFGKFIRIHF GPSGKLAGAD IETYLLEKAR VISQQSLERS YHIFYQVMSG AVPGVKELCL LSNDIYEYNY VSQ GKVTIP SVDDGEEFQA TDQAFDVLGF TQEEKDDIYK ITASVMHMGC MKFKQRGREE QAEADGTAEG ERVAKLLGLE AADL YKNLL KPRIKVGNEF VTQGRNLNQV IYSVGALSKG VFDRLFKFLV KKCNETLDTK QKRQHFIGVL DIAGFEIFDF NGFEQ LCIN FTNEKLQQFF NHHMFVLEQE EYTREGITWA FIDFGMDLVA CIDLIEKPMG ILSILEEESM FPKATDKTFE EKLMNN HLG KSPNFQKPKP PKPGCQAAHF AISHYAGVVS YNLTGWLEKN KDPLNDTVVD QFKKGSNKLL VEIFADHPGQ SGAPEAG GG GKGGRGKKGG GFATVSSSYK EQLNNLMTTL KSTQPHFVRC IIPNELKQPG LIDSHLVMHQ LTCNGVLEGI RICRKGFP N RMVYPDFKLR YMILAPATMA AEPDPKKAAD KCLKEVGLES ETYRIGHTKV FFRAGVLGQL EEMRDERLSK IIGWMQSHI RGYLARKQFK KYQDQRLSLQ VVQRNLRKYM ALRTWPWWKM WTKVKPLLNV ANVEEEMRKL EELVATTQAA LEKEEKARKE VEALNAKLI QEKTDLLRNL EGEKGSISSI QEKAAKLQAQ KSDLESQLMD TQERLQQEED NRNQMFQQKK KLEQEVGGLK K DIEDLELS LQKSDQDKAS KDHQIRNLND EIAHQDELIN KLNKEKKMQG EHTQKTAEEL QASEDKVNHL TKVKAKLEQT LD ELEDSLE REKKLRGDVE KAKRKVEGDL KLTQEAVADL ERNKKELEQT IQRKDKEIAS LTAKLEDEQS IVSKTQKQIK ELQ SRIEEL EEEVEAERQA RGKAEKQRAD LARELEELGE RLEEAGGATS AQIELNKKRE AEMSKLRRDL EESNIQHEST LANL RKKHN DAVSEMGEQI DQLNKLKTKA EHDRTHVQND LNNTRHALDQ MCREKAATEK IAKQLQHQVN EIQGKLDEAN RTLND FDSA KKKLSIENSD LLRQLEEAES QVSQLSKIKV SLTTQLEDTK RLADEEARER ATLLGKFRNL EHDLDNIREQ LEEEAE GKA DIQRQLSKAN AEAQLWRTKY ESEGVARAEE LEEAKRKLQA RLAEAEETIE SLNQKVIALE KTKQRLATEV EDLQLEV DR ATAIANAAEK KAKAIDKIIG EWKLKVDDLA AELDASQKEC RNYSTELFRL KGAYEEAQEQ LEAVRRENKN LADEVKDL L DQIGEGGRNI HEIEKQKKRL EVEKDELQAA LEEAEAALEQ EENKVLRSQL ELSQVRQEID RRIQEKEEEF ENTRKNHQR ALDSMQASLE AEAKGKAEAL RMKKKLEADI NELEIALDHA NKANSEAQKT IKKYQQQLKD VQTALEEEQR ARDDAREQLG ISERRANAL QNELEESRTL LEQADRGRRQ AEQELGDAHE QINELAAQAT SASAAKRKLE GELQTLHADL DELLNEAKNS E EKAKKAMV DAARLADELR AEQDHAQTQE KLRKALETQI KELQIRLDEA ETNALKGGKK AIAKLEQRVR ELENELDGEQ RR HADAQKN LRKSERRIKE LSFQADEDRK NHERMQDLVD KLQQKIKTYK RQIEEAEEIA ALNLAKFRKA QQELEEAEER ADL AEQAIA KFRTKGGRAG SAARAMSPVA HRPPVKHPLD GSTFPPRFDL HEDMM |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-64 (8k x 8k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 8192 pixel / デジタル化 - サイズ - 縦: 8192 pixel / デジタル化 - 画像ごとのフレーム数: 1-34 / 撮影したグリッド数: 1 / 実像数: 3507 / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー