+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22491 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 spike in complex with the S2H13 neutralizing antibody Fab fragment (local refinement of the receptor-binding motif and Fab variable domains) | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / entry receptor-mediated virion attachment to host cell / receptor-mediated endocytosis of virus by host cell / Attachment and Entry /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell /  receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) / Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Park YJ / Tortorici MA / Walls AC / Czudnochowski N / Seattle Structural Genomics Center for Infectious Disease (SSGCID) / Snell G / Veesler D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: Mapping Neutralizing and Immunodominant Sites on the SARS-CoV-2 Spike Receptor-Binding Domain by Structure-Guided High-Resolution Serology. 著者: Luca Piccoli / Young-Jun Park / M Alejandra Tortorici / Nadine Czudnochowski / Alexandra C Walls / Martina Beltramello / Chiara Silacci-Fregni / Dora Pinto / Laura E Rosen / John E Bowen / ...著者: Luca Piccoli / Young-Jun Park / M Alejandra Tortorici / Nadine Czudnochowski / Alexandra C Walls / Martina Beltramello / Chiara Silacci-Fregni / Dora Pinto / Laura E Rosen / John E Bowen / Oliver J Acton / Stefano Jaconi / Barbara Guarino / Andrea Minola / Fabrizia Zatta / Nicole Sprugasci / Jessica Bassi / Alessia Peter / Anna De Marco / Jay C Nix / Federico Mele / Sandra Jovic / Blanca Fernandez Rodriguez / Sneha V Gupta / Feng Jin / Giovanni Piumatti / Giorgia Lo Presti / Alessandra Franzetti Pellanda / Maira Biggiogero / Maciej Tarkowski / Matteo S Pizzuto / Elisabetta Cameroni / Colin Havenar-Daughton / Megan Smithey / David Hong / Valentino Lepori / Emiliano Albanese / Alessandro Ceschi / Enos Bernasconi / Luigia Elzi / Paolo Ferrari / Christian Garzoni / Agostino Riva / Gyorgy Snell / Federica Sallusto / Katja Fink / Herbert W Virgin / Antonio Lanzavecchia / Davide Corti / David Veesler /      要旨: Analysis of the specificity and kinetics of neutralizing antibodies (nAbs) elicited by SARS-CoV-2 infection is crucial for understanding immune protection and identifying targets for vaccine design. ...Analysis of the specificity and kinetics of neutralizing antibodies (nAbs) elicited by SARS-CoV-2 infection is crucial for understanding immune protection and identifying targets for vaccine design. In a cohort of 647 SARS-CoV-2-infected subjects, we found that both the magnitude of Ab responses to SARS-CoV-2 spike (S) and nucleoprotein and nAb titers correlate with clinical scores. The receptor-binding domain (RBD) is immunodominant and the target of 90% of the neutralizing activity present in SARS-CoV-2 immune sera. Whereas overall RBD-specific serum IgG titers waned with a half-life of 49 days, nAb titers and avidity increased over time for some individuals, consistent with affinity maturation. We structurally defined an RBD antigenic map and serologically quantified serum Abs specific for distinct RBD epitopes leading to the identification of two major receptor-binding motif antigenic sites. Our results explain the immunodominance of the receptor-binding motif and will guide the design of COVID-19 vaccines and therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22491.map.gz emd_22491.map.gz | 502.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22491-v30.xml emd-22491-v30.xml emd-22491.xml emd-22491.xml | 24.7 KB 24.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22491.png emd_22491.png | 42.3 KB | ||
| その他 |  emd_22491_additional_1.map.gz emd_22491_additional_1.map.gz emd_22491_half_map_1.map.gz emd_22491_half_map_1.map.gz emd_22491_half_map_2.map.gz emd_22491_half_map_2.map.gz | 121.9 MB 226.6 MB 226.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22491 http://ftp.pdbj.org/pub/emdb/structures/EMD-22491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22491 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7jv2MC  7jv4C  7jv6C  7jvaC  7jvcC  7jw0C  7jx3C  7jxcC  7jxdC  7jxeC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22491.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22491.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_22491_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
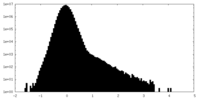
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_22491_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_22491_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike in complex with the S2H13 neutralizing antibody ...
| 全体 | 名称: SARS-CoV-2 spike in complex with the S2H13 neutralizing antibody Fab fragment |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike in complex with the S2H13 neutralizing antibody ...
| 超分子 | 名称: SARS-CoV-2 spike in complex with the S2H13 neutralizing antibody Fab fragment タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: spike glycoprotein
| 超分子 | 名称: spike glycoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: S2H13 neutralizing antibody Fab fragment
| 超分子 | 名称: S2H13 neutralizing antibody Fab fragment / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
-分子 #1: S2H13 Fab heavy chain
| 分子 | 名称: S2H13 Fab heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.086424 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: EVQLVESGGD SVQPGGSLRL SCAAAGFTFS SYWMNWVRQA PGKGLEWVAN IKQDGSEKYY VDSVKGRFTI SRDNAKNSLY LQMNSLRAE DTAVYYCALS SGYSGYAGNY WGQGTLVTVS S |
-分子 #2: S2H13 Fab light chain
| 分子 | 名称: S2H13 Fab light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.526824 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: QAVVTQEPSL TVSPGGTVTL TCGSSTGAVT SGHYPYWFQQ KPGQAPRTLI YDTSNKHSWT PARFSGSLLG GKAALTLSGA RPEDEAEYY CLLSYSGARG VFGGGTKLTV L |
-分子 #3: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
| 分子量 | 理論値: 141.532797 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GTQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF L GVYYHKNN ...文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GTQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF L GVYYHKNN KSWMESEFRV YSSANNCTFE YVSQPFLMDL EGKQGNFKNL REFVFKNIDG YFKIYSKHTP INLVRDLPQG FS ALEPLVD LPIGINITRF QTLLALHRSY LTPGDSSSGW TAGAAAYYVG YLQPRTFLLK YNENGTITDA VDCALDPLSE TKC TLKSFT VEKGIYQTSN FRVQPTESIV RFPNITNLCP FGEVFNATRF ASVYAWNRKR ISNCVADYSV LYNSASFSTF KCYG VSPTK LNDLCFTNVY ADSFVIRGDE VRQIAPGQTG KIADYNYKLP DDFTGCVIAW NSNNLDSKVG GNYNYLYRLF RKSNL KPFE RDISTEIYQA GSTPCNGVEG FNCYFPLQSY GFQPTNGVGY QPYRVVVLSF ELLHAPATVC GPKKSTNLVK NKCVNF NFN GLTGTGVLTE SNKKFLPFQQ FGRDIADTTD AVRDPQTLEI LDITPCSFGG VSVITPGTNT SNQVAVLYQD VNCTEVP VA IHADQLTPTW RVYSTGSNVF QTRAGCLIGA EHVNNSYECD IPIGAGICAS YQTQTNSPSG AGSVASQSII AYTMSLGA E NSVAYSNNSI AIPTNFTISV TTEILPVSMT KTSVDCTMYI CGDSTECSNL LLQYGSFCTQ LNRALTGIAV EQDKNTQEV FAQVKQIYKT PPIKDFGGFN FSQILPDPSK PSKRSFIEDL LFNKVTLADA GFIKQYGDCL GDIAARDLIC AQKFNGLTVL PPLLTDEMI AQYTSALLAG TITSGWTFGA GAALQIPFAM QMAYRFNGIG VTQNVLYENQ KLIANQFNSA IGKIQDSLSS T ASALGKLQ DVVNQNAQAL NTLVKQLSSN FGAISSVLND ILSRLDPPEA EVQIDRLITG RLQSLQTYVT QQLIRAAEIR AS ANLAATK MSECVLGQSK RVDFCGKGYH LMSFPQSAPH GVVFLHVTYV PAQEKNFTTA PAICHDGKAH FPREGVFVSN GTH WFVTQR NFYEPQIITT DNTFVSGNCD VVIGIVNNTV YDPLQPELDS FKEELDKYFK NHTSPDVDLG DISGINASVV NIQK EIDRL NEVAKNLNES LIDLQELGKY EQYIKGSGRE NLYFQGGGGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGHH HHHHH H |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 70.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 311071 |
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X