+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20327 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Spastin Hexamer in complex with substrate peptide | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAA+ ATPase / Microtubule Severing / MOTOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytokinetic process / microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / endoplasmic reticulum tubular network / positive regulation of microtubule depolymerization / cytoskeleton-dependent cytokinesis / mitotic nuclear membrane reassembly / nuclear membrane reassembly / Sealing of the nuclear envelope (NE) by ESCRT-III ...cytokinetic process / microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / endoplasmic reticulum tubular network / positive regulation of microtubule depolymerization / cytoskeleton-dependent cytokinesis / mitotic nuclear membrane reassembly / nuclear membrane reassembly / Sealing of the nuclear envelope (NE) by ESCRT-III / membrane fission / anterograde axonal transport / microtubule bundle formation / mitotic spindle disassembly / protein hexamerization / exit from mitosis / axonal transport of mitochondrion / beta-tubulin binding / positive regulation of cytokinesis / mitotic cytokinesis / alpha-tubulin binding / endoplasmic reticulum to Golgi vesicle-mediated transport / lipid droplet / axon cytoplasm / axonogenesis / isomerase activity / protein homooligomerization / spindle pole / midbody / microtubule binding / cytoplasmic vesicle / nuclear membrane / microtubule / endosome / axon / centrosome / endoplasmic reticulum membrane / protein-containing complex binding / perinuclear region of cytoplasm / ATP hydrolysis activity / extracellular exosome / nucleoplasm / ATP binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Han H / Schubert HL | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2020 ジャーナル: J Biol Chem / 年: 2020タイトル: Structure of spastin bound to a glutamate-rich peptide implies a hand-over-hand mechanism of substrate translocation. 著者: Han Han / Heidi L Schubert / John McCullough / Nicole Monroe / Michael D Purdy / Mark Yeager / Wesley I Sundquist / Christopher P Hill /  要旨: Many members of the AAA+ ATPase family function as hexamers that unfold their protein substrates. These AAA unfoldases include spastin, which plays a critical role in the architecture of eukaryotic ...Many members of the AAA+ ATPase family function as hexamers that unfold their protein substrates. These AAA unfoldases include spastin, which plays a critical role in the architecture of eukaryotic cells by driving the remodeling and severing of microtubules, which are cytoskeletal polymers of tubulin subunits. Here, we demonstrate that a human spastin binds weakly to unmodified peptides from the C-terminal segment of human tubulin α1A/B. A peptide comprising alternating glutamate and tyrosine residues binds more tightly, which is consistent with the known importance of glutamylation for spastin microtubule severing activity. A cryo-EM structure of the spastin-peptide complex at 4.2 Å resolution revealed an asymmetric hexamer in which five spastin subunits adopt a helical, spiral staircase configuration that binds the peptide within the central pore, whereas the sixth subunit of the hexamer is displaced from the peptide/substrate, as if transitioning from one end of the helix to the other. This configuration differs from a recently published structure of spastin from , which forms a six-subunit spiral without a transitioning subunit. Our structure resembles other recently reported AAA unfoldases, including the meiotic clade relative Vps4, and supports a model in which spastin utilizes a hand-over-hand mechanism of tubulin translocation and microtubule remodeling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20327.map.gz emd_20327.map.gz | 5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20327-v30.xml emd-20327-v30.xml emd-20327.xml emd-20327.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20327.png emd_20327.png | 72 KB | ||
| Filedesc metadata |  emd-20327.cif.gz emd-20327.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20327 http://ftp.pdbj.org/pub/emdb/structures/EMD-20327 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20327 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20327 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20327_validation.pdf.gz emd_20327_validation.pdf.gz | 361.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20327_full_validation.pdf.gz emd_20327_full_validation.pdf.gz | 361.2 KB | 表示 | |
| XML形式データ |  emd_20327_validation.xml.gz emd_20327_validation.xml.gz | 6 KB | 表示 | |
| CIF形式データ |  emd_20327_validation.cif.gz emd_20327_validation.cif.gz | 7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20327 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20327 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20327 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20327 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6pekMC  6penMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10382 (タイトル: Structure of spastin bound to a glutamate-rich peptide implies a hand-over-hand mechanism of substrate translocation. EMPIAR-10382 (タイトル: Structure of spastin bound to a glutamate-rich peptide implies a hand-over-hand mechanism of substrate translocation.Data size: 1.8 TB Data #1: Unaligned multi-frame movies of ADPBeFx-bound Spastin [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20327.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20327.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.056 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
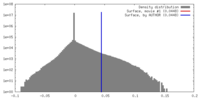
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Spastin Hexamer in complex with substrate peptide
| 全体 | 名称: Spastin Hexamer in complex with substrate peptide |
|---|---|
| 要素 |
|
-超分子 #1: Spastin Hexamer in complex with substrate peptide
| 超分子 | 名称: Spastin Hexamer in complex with substrate peptide / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Spastin
| 分子 | 名称: Spastin / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO / EC番号: microtubule-severing ATPase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 54.498328 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAKRSSGAA PAPASASAPA PVPGGEAERV RVFHKQAFEY ISIALRIDED EKAGQKEQAV EWYKKGIEEL EKGIAVIVTG QGEQCERAR RLQAKMMTNL VMAKDRLQLL ESGAVPKRKD PLTHTSNSLP RSKTVMKTGS AGLSGHHRAP SYSGLSMVSG V KQGSGPAP ...文字列: MAAKRSSGAA PAPASASAPA PVPGGEAERV RVFHKQAFEY ISIALRIDED EKAGQKEQAV EWYKKGIEEL EKGIAVIVTG QGEQCERAR RLQAKMMTNL VMAKDRLQLL ESGAVPKRKD PLTHTSNSLP RSKTVMKTGS AGLSGHHRAP SYSGLSMVSG V KQGSGPAP TTHKGTPKTN RTNKPSTPTT ATRKKKDLKN FRNVDSNLAN LIMNEIVDNG TAVKFDDIAG QDLAKQALQE IV ILPSLRP ELFTGLRAPA RGLLLFGPPG NGKTMLAKAV AAESNATFFN ISAASLTSKY VGEGEKLVRA LFAVARELQP SII FIDEVD SLLCERREGE HDASRRLKTE FLIEFDGVQS AGDDRVLVMG ATNRPQELDE AVLRRFIKRV YVSLPNEETR LLLL KNLLC KQGSPLTQKE LAQLARMTDG YSGSDLTALA KDAALGPIRE LKPEQVKNMS ASEMRNIRLS DFTESLKKIK RSVSP QTLE AYIRWNKDFG DTTV UniProtKB: Spastin |
-分子 #2: substrate peptide, TYR-GLU-TYR-GLU-TYR-GLU-TYR-GLU
| 分子 | 名称: substrate peptide, TYR-GLU-TYR-GLU-TYR-GLU-TYR-GLU / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 1.479453 KDa |
| 配列 | 文字列: EYEYEYEYEY |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 5 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: BERYLLIUM TRIFLUORIDE ION
| 分子 | 名称: BERYLLIUM TRIFLUORIDE ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: BEF |
|---|---|
| 分子量 | 理論値: 66.007 Da |
| Chemical component information |  ChemComp-BEF: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 57.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)