+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10045 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
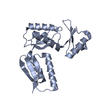
| タイトル | Inner membrane ring of the Shigella type 3 secretion system | |||||||||
 マップデータ マップデータ | Masked C24 map of inner membrane ring of Shigella type 3 secretion system, post-processed, sharpened (b=-128) and filtered at 3.55 A. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.55 Å クライオ電子顕微鏡法 / 解像度: 3.55 Å | |||||||||
 データ登録者 データ登録者 | Lunelli M / Kamprad A | |||||||||
| 資金援助 | 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2020 ジャーナル: PLoS Pathog / 年: 2020タイトル: Cryo-EM structure of the Shigella type III needle complex. 著者: Michele Lunelli / Antje Kamprad / Jörg Bürger / Thorsten Mielke / Christian M T Spahn / Michael Kolbe /  要旨: The Type III Secretion Systems (T3SS) needle complex is a conserved syringe-shaped protein translocation nanomachine with a mass of about 3.5 MDa essential for the survival and virulence of many Gram- ...The Type III Secretion Systems (T3SS) needle complex is a conserved syringe-shaped protein translocation nanomachine with a mass of about 3.5 MDa essential for the survival and virulence of many Gram-negative bacterial pathogens. This system is composed of a membrane-embedded basal body and an extracellular needle that deliver effector proteins into host cells. High-resolution structures of the T3SS from different organisms and infection stages are needed to understand the underlying molecular mechanisms of effector translocation. Here, we present the cryo-electron microscopy structure of the isolated Shigella T3SS needle complex. The inner membrane (IM) region of the basal body adopts 24-fold rotational symmetry and forms a channel system that connects the bacterial periplasm with the export apparatus cage. The secretin oligomer adopts a heterogeneous architecture with 16- and 15-fold cyclic symmetry in the periplasmic N-terminal connector and C-terminal outer membrane ring, respectively. Two out of three IM subunits bind the secretin connector via a β-sheet augmentation. The cryo-EM map also reveals the helical architecture of the export apparatus core, the inner rod, the needle and their intervening interfaces. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10045.map.gz emd_10045.map.gz | 31.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10045-v30.xml emd-10045-v30.xml emd-10045.xml emd-10045.xml | 22.5 KB 22.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10045_fsc.xml emd_10045_fsc.xml | 16.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10045.png emd_10045.png | 87.9 KB | ||
| マスクデータ |  emd_10045_msk_1.map emd_10045_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10045_additional.map.gz emd_10045_additional.map.gz emd_10045_half_map_1.map.gz emd_10045_half_map_1.map.gz emd_10045_half_map_2.map.gz emd_10045_half_map_2.map.gz | 32.5 MB 331.3 MB 330 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10045 http://ftp.pdbj.org/pub/emdb/structures/EMD-10045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10045 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10045.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10045.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked C24 map of inner membrane ring of Shigella type 3 secretion system, post-processed, sharpened (b=-128) and filtered at 3.55 A. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.38369 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10045_msk_1.map emd_10045_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Masked C24 map of inner membrane ring of...
| ファイル | emd_10045_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked C24 map of inner membrane ring of Shigella type 3 secretion system, post-processed, sharpened (b=-128) and filtered at 3.2 A. Used for model building and refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map
| ファイル | emd_10045_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map
| ファイル | emd_10045_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The periplasmic inner membrane ring of the Shigella type 3 secret...
| 全体 | 名称: The periplasmic inner membrane ring of the Shigella type 3 secretion system |
|---|---|
| 要素 |
|
-超分子 #1: The periplasmic inner membrane ring of the Shigella type 3 secret...
| 超分子 | 名称: The periplasmic inner membrane ring of the Shigella type 3 secretion system タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Focused reconstruction from isolated needle complexes of the Shigella type 3 secretion system |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) / 株: M90T / 細胞中の位置: Membrane Shigella flexneri (フレクスナー赤痢菌) / 株: M90T / 細胞中の位置: Membrane |
-分子 #1: Protein MxiG
| 分子 | 名称: Protein MxiG / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
| 分子量 | 理論値: 43.053844 KDa |
| 配列 | 文字列: MSEAKNSNLA PFRLLVKLTN GVGDEFPLYY GNNLIVLGRT IETLEFGNDN FPENIIPVTD SKSDGIIYLT ISKDNICQFS DEKGEQIDI NSQFNSFEYD GISFHLKNMR EDKSRGHILN GMYKNHSVFF FFAVIVVLII IFSLSLKKDE VKEIAEIIDD K RYGIVNTG ...文字列: MSEAKNSNLA PFRLLVKLTN GVGDEFPLYY GNNLIVLGRT IETLEFGNDN FPENIIPVTD SKSDGIIYLT ISKDNICQFS DEKGEQIDI NSQFNSFEYD GISFHLKNMR EDKSRGHILN GMYKNHSVFF FFAVIVVLII IFSLSLKKDE VKEIAEIIDD K RYGIVNTG QCNYILAETQ NDAVWASVAL NKTGFTKCRY ILVSNKEINR IQQYINQRFP FINLYVLNLV SDKAELLVFL SK ERNSSKD TELDKLKNAL IVEFPYIKNI KFNYLSDHNA RGDAKGIFTK VNVQYKEICE NNKVTYSVRE ELTDEKLELI NRL ISEHKN IYGDQYIEFS VLLIDDDFKG KSYLNSKDSY VMLNDKHWFF LDKNK |
-分子 #2: Lipoprotein MxiJ
| 分子 | 名称: Lipoprotein MxiJ / タイプ: protein_or_peptide / ID: 2 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
| 分子量 | 理論値: 27.542055 KDa |
| 配列 | 文字列: MIRYKGFILF LLLMLIGCEQ REELISNLSQ RQANEIISVL ERHNITARKV DGGKQGISVQ VEKGTFASAV DLMRMYDLPN PERVDISQM FPTDSLVSSP RAEKARLYSA IEQRLEQSLV SIGGVISAKI HVSYDLEEKN ISSKPMHISV IAIYDSPKES E LLVSNIKR ...文字列: MIRYKGFILF LLLMLIGCEQ REELISNLSQ RQANEIISVL ERHNITARKV DGGKQGISVQ VEKGTFASAV DLMRMYDLPN PERVDISQM FPTDSLVSSP RAEKARLYSA IEQRLEQSLV SIGGVISAKI HVSYDLEEKN ISSKPMHISV IAIYDSPKES E LLVSNIKR FLKNTFSDVK YENISVILTP KEEYVYTNVQ PVKEVKSEFL TNEVIYLFLG MAVLVVILLV WAFKTGWFKR NK I |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 5.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: Sample applied on grid 5 ul, incubation time 5 min on ice, then moved into Vitrobot and 5 ul sample applied again. Blot time: 2 sec Blot force: -2 Drain time: 0 sec. |
| 詳細 | Isolated needle complex in detergent solution |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.004 µm / 最小 デフォーカス(公称値): 0.0015 µm / 倍率(公称値): 101179 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.004 µm / 最小 デフォーカス(公称値): 0.0015 µm / 倍率(公称値): 101179 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 5238 / 平均露光時間: 1.5 sec. / 平均電子線量: 25.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 詳細 | Model was built and refined in the map low-pass filtered at 3.2 A | ||||||
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 140 当てはまり具合の基準: Correlation coefficient and geometry | ||||||
| 得られたモデル |  PDB-6rwx: |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X