+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0336 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the yeast Sec complex | |||||||||
 マップデータ マップデータ | Sharpened full map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Sec61 (Sec61) / Sec61 (Sec61) /  Sec63 / Sec71 / Sec72 / Sec66 / Sec63 / Sec71 / Sec72 / Sec66 /  protein translocation / protein translocation /  translocon (トランスロコン) / translocon (トランスロコン) /  endoplasmic reticulum (小胞体) / endoplasmic reticulum (小胞体) /  secretion (分泌) / secretion (分泌) /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein transmembrane import into intracellular organelle /  misfolded protein transport / Sec62/Sec63 complex / misfolded protein transport / Sec62/Sec63 complex /  translocon complex / cytosol to endoplasmic reticulum transport / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / rough endoplasmic reticulum membrane / Ssh1 translocon complex / Sec61 translocon complex / protein-transporting ATPase activity ...protein transmembrane import into intracellular organelle / translocon complex / cytosol to endoplasmic reticulum transport / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / rough endoplasmic reticulum membrane / Ssh1 translocon complex / Sec61 translocon complex / protein-transporting ATPase activity ...protein transmembrane import into intracellular organelle /  misfolded protein transport / Sec62/Sec63 complex / misfolded protein transport / Sec62/Sec63 complex /  translocon complex / cytosol to endoplasmic reticulum transport / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / rough endoplasmic reticulum membrane / Ssh1 translocon complex / Sec61 translocon complex / protein-transporting ATPase activity / translocon complex / cytosol to endoplasmic reticulum transport / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / rough endoplasmic reticulum membrane / Ssh1 translocon complex / Sec61 translocon complex / protein-transporting ATPase activity /  post-translational protein targeting to endoplasmic reticulum membrane / filamentous growth / SRP-dependent cotranslational protein targeting to membrane, translocation / peptide transmembrane transporter activity / post-translational protein targeting to endoplasmic reticulum membrane / filamentous growth / SRP-dependent cotranslational protein targeting to membrane, translocation / peptide transmembrane transporter activity /  signal sequence binding / SRP-dependent cotranslational protein targeting to membrane / signal sequence binding / SRP-dependent cotranslational protein targeting to membrane /  post-translational protein targeting to membrane, translocation / nuclear inner membrane / retrograde protein transport, ER to cytosol / protein transmembrane transporter activity / : / guanyl-nucleotide exchange factor activity / cell periphery / post-translational protein targeting to membrane, translocation / nuclear inner membrane / retrograde protein transport, ER to cytosol / protein transmembrane transporter activity / : / guanyl-nucleotide exchange factor activity / cell periphery /  ribosome binding / endoplasmic reticulum membrane / structural molecule activity / ribosome binding / endoplasmic reticulum membrane / structural molecule activity /  小胞体 / 小胞体 /  ミトコンドリア / ミトコンドリア /  生体膜 / 生体膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae S288c (パン酵母) / Saccharomyces cerevisiae S288c (パン酵母) /   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) | |||||||||
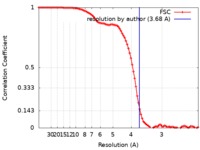
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.68 Å クライオ電子顕微鏡法 / 解像度: 3.68 Å | |||||||||
 データ登録者 データ登録者 | Park E / Itskanov S | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2019 ジャーナル: Science / 年: 2019タイトル: Structure of the posttranslational Sec protein-translocation channel complex from yeast. 著者: Samuel Itskanov / Eunyong Park /  要旨: The Sec61 protein-conducting channel mediates transport of many proteins, such as secretory proteins, across the endoplasmic reticulum (ER) membrane during or after translation. Posttranslational ...The Sec61 protein-conducting channel mediates transport of many proteins, such as secretory proteins, across the endoplasmic reticulum (ER) membrane during or after translation. Posttranslational transport is enabled by two additional membrane proteins associated with the channel, Sec63 and Sec62, but its mechanism is poorly understood. We determined a structure of the Sec complex (Sec61-Sec63-Sec71-Sec72) from by cryo-electron microscopy (cryo-EM). The structure shows that Sec63 tightly associates with Sec61 through interactions in cytosolic, transmembrane, and ER-luminal domains, prying open Sec61's lateral gate and translocation pore and thus activating the channel for substrate engagement. Furthermore, Sec63 optimally positions binding sites for cytosolic and luminal chaperones in the complex to enable efficient polypeptide translocation. Our study provides mechanistic insights into eukaryotic posttranslational protein translocation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0336.map.gz emd_0336.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0336-v30.xml emd-0336-v30.xml emd-0336.xml emd-0336.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0336_fsc.xml emd_0336_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0336.png emd_0336.png | 58 KB | ||
| Filedesc metadata |  emd-0336.cif.gz emd-0336.cif.gz | 6.7 KB | ||
| その他 |  emd_0336_additional.map.gz emd_0336_additional.map.gz | 59.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0336 http://ftp.pdbj.org/pub/emdb/structures/EMD-0336 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0336 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0336 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0336.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0336.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened full map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unfiltered full map
| ファイル | emd_0336_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered full map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
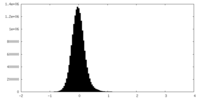
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Posttranslational Sec protein-translocation channel complex
| 全体 | 名称: Posttranslational Sec protein-translocation channel complex |
|---|---|
| 要素 |
|
-超分子 #1: Posttranslational Sec protein-translocation channel complex
| 超分子 | 名称: Posttranslational Sec protein-translocation channel complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288c (パン酵母) Saccharomyces cerevisiae S288c (パン酵母) |
-分子 #1: Protein transport protein SEC61
| 分子 | 名称: Protein transport protein SEC61 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)株: ATCC 204508 / S288c |
| 分子量 | 理論値: 52.978148 KDa |
| 配列 | 文字列: MSSNRVLDLF KPFESFLPEV IAPERKVPYN QKLIWTGVSL LIFLILGQIP LYGIVSSETS DPLYWLRAML ASNRGTLLEL GVSPIITSS MIFQFLQGTQ LLQIRPESKQ DRELFQIAQK VCAIILILGQ ALVVVMTGNY GAPSDLGLPI CLLLIFQLMF A SLIVMLLD ...文字列: MSSNRVLDLF KPFESFLPEV IAPERKVPYN QKLIWTGVSL LIFLILGQIP LYGIVSSETS DPLYWLRAML ASNRGTLLEL GVSPIITSS MIFQFLQGTQ LLQIRPESKQ DRELFQIAQK VCAIILILGQ ALVVVMTGNY GAPSDLGLPI CLLLIFQLMF A SLIVMLLD ELLSKGYGLG SGISLFTATN IAEQIFWRAF APTTVNSGRG KEFEGAVIAF FHLLAVRKDK KRALVEAFYR TN LPNMFQV LMTVAIFLFV LYLQGFRYEL PIRSTKVRGQ IGIYPIKLFY TSNTPIMLQS ALTSNIFLIS QILFQKYPTN PLI RLIGVW GIRPGTQGPQ MALSGLAYYI QPLMSLSEAL LDPIKTIVYI TFVLGSCAVF SKTWIEISGT SPRDIAKQFK DQGM VINGK RETSIYRELK KIIPTAAAFG GATIGALSVG SDLLGTLGSG ASILMATTTI YGYYEAAAKE GGFTKNLVPG FSDLM UniProtKB:  Protein transport protein SEC61 Protein transport protein SEC61 |
-分子 #2: Protein transport protein SSS1
| 分子 | 名称: Protein transport protein SSS1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)株: ATCC 204508 / S288c |
| 分子量 | 理論値: 8.958641 KDa |
| 配列 | 文字列: MARASEKGEE KKQSNNQVEK LVEAPVEFVR EGTQFLAKCK KPDLKEYTKI VKAVGIGFIA VGIIGYAIKL IHIPIRYVIV UniProtKB:  Protein transport protein SSS1 Protein transport protein SSS1 |
-分子 #3: Protein transport protein SBH1
| 分子 | 名称: Protein transport protein SBH1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)株: ATCC 204508 / S288c |
| 分子量 | 理論値: 8.723155 KDa |
| 配列 | 文字列: MSSPTPPGGQ RTLQKRKQGS SQKVAASAPK KNTNSNNSIL KIYSDEATGL RVDPLVVLFL AVGFIFSVVA LHVISKVAGK LF UniProtKB:  Protein transport protein SBH1 Protein transport protein SBH1 |
-分子 #4: Protein translocation protein SEC63
| 分子 | 名称: Protein translocation protein SEC63 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)株: ATCC 204508 / S288c |
| 分子量 | 理論値: 75.432258 KDa |
| 配列 | 文字列: MPTNYEYDEA SETWPSFILT GLLMVVGPMT LLQIYQIFFG ANAEDGNSGK SKEFNEEVFK NLNEEYTSDE IKQFRRKFDK NSNKKSKIW SRRNIIIIVG WILVAILLQR INSNDAIKDA ATKLFDPYEI LGISTSASDR DIKSAYRKLS VKFHPDKLAK G LTPDEKSV ...文字列: MPTNYEYDEA SETWPSFILT GLLMVVGPMT LLQIYQIFFG ANAEDGNSGK SKEFNEEVFK NLNEEYTSDE IKQFRRKFDK NSNKKSKIW SRRNIIIIVG WILVAILLQR INSNDAIKDA ATKLFDPYEI LGISTSASDR DIKSAYRKLS VKFHPDKLAK G LTPDEKSV MEETYVQITK AYESLTDELV RQNYLKYGHP DGPQSTSHGI ALPRFLVDGS ASPLLVVCYV ALLGLILPYF VS RWWARTQ SYTKKGIHNV TASNFVSNLV NYKPSEIVTT DLILHWLSFA HEFKQFFPDL QPTDFEKLLQ DHINRRDSGK LNN AKFRIV AKCHSLLHGL LDIACGFRNL DIALGAINTF KCIVQAVPLT PNCQILQLPN VDKEHFITKT GDIHTLGKLF TLED AKIGE VLGIKDQAKL NETLRVASHI PNLKIIKADF LVPGENQVTP SSTPYISLKV LVRSAKQPLI PTSLIPEENL TEPQD FESQ RDPFAMMSKQ PLVPYSFAPF FPTKRRGSWC CLVSSQKDGK ILQTPIIIEK LSYKNLNDDK DFFDKRIKMD LTKHEK FDI NDWEIGTIKI PLGQPAPETV GDFFFRVIVK STDYFTTDLD ITMNMKVRDS PAVEQVEVYS EEDDEYSTDD DETESDD ES DASDYTDIDT DTEAEDDESP E UniProtKB:  Protein translocation protein SEC63 Protein translocation protein SEC63 |
-分子 #5: Translocation protein SEC66
| 分子 | 名称: Translocation protein SEC66 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)株: ATCC 204508 / S288c |
| 分子量 | 理論値: 24.263939 KDa |
| 配列 | 文字列: MSEFNETKFS NNGTFFETEE PIVETKSISV YTPLIYVFIL VVSLVMFASS YRKKQAKKIS EQPSIFDEND AHDLYFQIKE MSENEKIHE KVLKAALLNR GAESVRRSLK LKELAPQINL LYKNGSIGED YWKRFETEVK LIELEFKDTL QEAERLQPGW V QLFVMVCK ...文字列: MSEFNETKFS NNGTFFETEE PIVETKSISV YTPLIYVFIL VVSLVMFASS YRKKQAKKIS EQPSIFDEND AHDLYFQIKE MSENEKIHE KVLKAALLNR GAESVRRSLK LKELAPQINL LYKNGSIGED YWKRFETEVK LIELEFKDTL QEAERLQPGW V QLFVMVCK EICFNQALSR RYQSILKRKE VCIKEWELKI NNDGRLVN UniProtKB: Translocation protein SEC66 |
-分子 #6: Translocation protein SEC72
| 分子 | 名称: Translocation protein SEC72 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)株: ATCC 204508 / S288c |
| 分子量 | 理論値: 21.63109 KDa |
| 配列 | 文字列: MVTLEYNANS KLITASDAVV ALSTETNIDQ INVLTTSLIG ETNPNFTPQP NEALSKMIKG LFESGMKNLQ QKKLNEALKN VSLAIEMAQ RKRAPWEAFA IQLPELHFML RSKIDLCLIL GKHLEALQDL DFLLGTGLIQ PDVFVRKADC LLKLRQWEEA R ATCERGLA ...文字列: MVTLEYNANS KLITASDAVV ALSTETNIDQ INVLTTSLIG ETNPNFTPQP NEALSKMIKG LFESGMKNLQ QKKLNEALKN VSLAIEMAQ RKRAPWEAFA IQLPELHFML RSKIDLCLIL GKHLEALQDL DFLLGTGLIQ PDVFVRKADC LLKLRQWEEA R ATCERGLA LAPEDMKLRA LLIETARNLA EYNGE UniProtKB: Translocation protein SEC72 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 43103 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 43103 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 8.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z
Z Y
Y X
X