+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5fmg | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure and function based design of Plasmodium-selective proteasome inhibitors | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  HYDROLASE (加水分解酵素) / HYDROLASE (加水分解酵素) /  PROTEASOME (プロテアソーム) / 20S / PROTEASOME (プロテアソーム) / 20S /  PLASMODIUM (マラリア原虫) / PLASMODIUM (マラリア原虫) /  MALARIA (マラリア) / MALARIA (マラリア) /  INHIBITOR (酵素阻害剤) / INHIBITOR (酵素阻害剤) /  DRUG DESIGN (医薬品設計) / DRUG DESIGN (医薬品設計) /  CRYO-EM (低温電子顕微鏡法) CRYO-EM (低温電子顕微鏡法) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報AUF1 (hnRNP D0) binds and destabilizes mRNA / Cross-presentation of soluble exogenous antigens (endosomes) / ABC-family proteins mediated transport / MAPK6/MAPK4 signaling /  : / Orc1 removal from chromatin / CDK-mediated phosphorylation and removal of Cdc6 / KEAP1-NFE2L2 pathway / UCH proteinases / Ub-specific processing proteases ...AUF1 (hnRNP D0) binds and destabilizes mRNA / Cross-presentation of soluble exogenous antigens (endosomes) / ABC-family proteins mediated transport / MAPK6/MAPK4 signaling / : / Orc1 removal from chromatin / CDK-mediated phosphorylation and removal of Cdc6 / KEAP1-NFE2L2 pathway / UCH proteinases / Ub-specific processing proteases ...AUF1 (hnRNP D0) binds and destabilizes mRNA / Cross-presentation of soluble exogenous antigens (endosomes) / ABC-family proteins mediated transport / MAPK6/MAPK4 signaling /  : / Orc1 removal from chromatin / CDK-mediated phosphorylation and removal of Cdc6 / KEAP1-NFE2L2 pathway / UCH proteinases / Ub-specific processing proteases / : / Orc1 removal from chromatin / CDK-mediated phosphorylation and removal of Cdc6 / KEAP1-NFE2L2 pathway / UCH proteinases / Ub-specific processing proteases /  Neddylation / Antigen processing: Ubiquitination & Proteasome degradation / Neutrophil degranulation / proteasome core complex / proteasomal ubiquitin-independent protein catabolic process / Neddylation / Antigen processing: Ubiquitination & Proteasome degradation / Neutrophil degranulation / proteasome core complex / proteasomal ubiquitin-independent protein catabolic process /  proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome core complex, alpha-subunit complex / threonine-type endopeptidase activity / proteasomal protein catabolic process / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process / proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome core complex, alpha-subunit complex / threonine-type endopeptidase activity / proteasomal protein catabolic process / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process /  endopeptidase activity / endopeptidase activity /  hydrolase activity / hydrolase activity /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||
 データ登録者 データ登録者 | Li, H. / O'Donoghue, A.J. / van der Linden, W.A. / Xie, S.C. / Yoo, E. / Foe, I.T. / Tilley, L. / Craik, C.S. / da Fonseca, P.C.A. / Bogyo, M. | ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Structure- and function-based design of Plasmodium-selective proteasome inhibitors. 著者: Hao Li / Anthony J O'Donoghue / Wouter A van der Linden / Stanley C Xie / Euna Yoo / Ian T Foe / Leann Tilley / Charles S Craik / Paula C A da Fonseca / Matthew Bogyo /    要旨: The proteasome is a multi-component protease complex responsible for regulating key processes such as the cell cycle and antigen presentation. Compounds that target the proteasome are potentially ...The proteasome is a multi-component protease complex responsible for regulating key processes such as the cell cycle and antigen presentation. Compounds that target the proteasome are potentially valuable tools for the treatment of pathogens that depend on proteasome function for survival and replication. In particular, proteasome inhibitors have been shown to be toxic for the malaria parasite Plasmodium falciparum at all stages of its life cycle. Most compounds that have been tested against the parasite also inhibit the mammalian proteasome, resulting in toxicity that precludes their use as therapeutic agents. Therefore, better definition of the substrate specificity and structural properties of the Plasmodium proteasome could enable the development of compounds with sufficient selectivity to allow their use as anti-malarial agents. To accomplish this goal, here we use a substrate profiling method to uncover differences in the specificities of the human and P. falciparum proteasome. We design inhibitors based on amino-acid preferences specific to the parasite proteasome, and find that they preferentially inhibit the β2-subunit. We determine the structure of the P. falciparum 20S proteasome bound to the inhibitor using cryo-electron microscopy and single-particle analysis, to a resolution of 3.6 Å. These data reveal the unusually open P. falciparum β2 active site and provide valuable information about active-site architecture that can be used to further refine inhibitor design. Furthermore, consistent with the recent finding that the proteasome is important for stress pathways associated with resistance of artemisinin family anti-malarials, we observe growth inhibition synergism with low doses of this β2-selective inhibitor in artemisinin-sensitive and -resistant parasites. Finally, we demonstrate that a parasite-selective inhibitor could be used to attenuate parasite growth in vivo without appreciable toxicity to the host. Thus, the Plasmodium proteasome is a chemically tractable target that could be exploited by next-generation anti-malarial agents. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5fmg.cif.gz 5fmg.cif.gz | 904.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5fmg.ent.gz pdb5fmg.ent.gz | 698.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5fmg.json.gz 5fmg.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fm/5fmg https://data.pdbj.org/pub/pdb/validation_reports/fm/5fmg ftp://data.pdbj.org/pub/pdb/validation_reports/fm/5fmg ftp://data.pdbj.org/pub/pdb/validation_reports/fm/5fmg | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 5種, 10分子 AOFTGUHVJX
| #1: タンパク質 |  プロテアソーム プロテアソーム分子量: 29531.656 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8IAR3,  proteasome endopeptidase complex proteasome endopeptidase complex#6: タンパク質 |  プロテアソーム プロテアソーム分子量: 28871.697 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8IK90 #7: タンパク質 |  プロテアソーム プロテアソーム分子量: 29324.295 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: O77396 #8: タンパク質 |  プロテアソーム プロテアソーム分子量: 29143.936 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8I0U7,  proteasome endopeptidase complex proteasome endopeptidase complex#10: タンパク質 | 分子量: 24533.131 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8I261,  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|
-PROTEASOME SUBUNIT ALPHA ... , 4種, 8分子 BPCQDRES
| #2: タンパク質 |  プロテアソーム プロテアソーム分子量: 26556.391 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: C6KST3,  proteasome endopeptidase complex proteasome endopeptidase complex#3: タンパク質 |  プロテアソーム プロテアソーム分子量: 27977.664 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8IDG3,  proteasome endopeptidase complex proteasome endopeptidase complex#4: タンパク質 |  プロテアソーム プロテアソーム分子量: 27263.285 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8IDG2,  proteasome endopeptidase complex proteasome endopeptidase complex#5: タンパク質 |  プロテアソーム プロテアソーム分子量: 28417.367 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8IBI3,  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|
-PROTEASOME SUBUNIT BETA ... , 5種, 10分子 IWKYLZMaNb
| #9: タンパク質 |  プロテアソーム プロテアソーム分子量: 25104.885 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8I6T3,  proteasome endopeptidase complex proteasome endopeptidase complex#11: タンパク質 |  プロテアソーム プロテアソーム分子量: 22889.105 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8IKC9,  proteasome endopeptidase complex proteasome endopeptidase complex#12: タンパク質 |  プロテアソーム プロテアソーム分子量: 23620.646 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q8IJT1,  proteasome endopeptidase complex proteasome endopeptidase complex#13: タンパク質 |  プロテアソーム プロテアソーム分子量: 27301.203 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: C0H4E8,  proteasome endopeptidase complex proteasome endopeptidase complex#14: タンパク質 |  プロテアソーム プロテアソーム分子量: 30909.893 Da / 分子数: 2 / 由来タイプ: 天然 詳細: THE SAMPLE PROTEIN COMPONENTS ARE DEPOSITED IN THE PLASMODB (THE PLASMODIUM GENOME RESOURCE) 由来: (天然)   PLASMODIUM FALCIPARUM (マラリア病原虫) PLASMODIUM FALCIPARUM (マラリア病原虫)参照: UniProt: Q7K6A9,  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|
-非ポリマー , 1種, 2分子 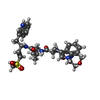
| #15: 化合物 |
|---|
-詳細
| 配列の詳細 | 5FMG A PLASMODB PF3D7_0807500 5FMG O PLASMODB PF3D7_0807500 5FMG B PLASMODB PF3D7_0608500 5FMG P ...5FMG A PLASMODB PF3D7_0807500 5FMG O PLASMODB PF3D7_0807500 5FMG B PLASMODB PF3D7_0608500 5FMG P PLASMODB PF3D7_0608500 5FMG C PLASMODB PF3D7_1353800 5FMG Q PLASMODB PF3D7_1353800 5FMG D PLASMODB PF3D7_1353900 5FMG R PLASMODB PF3D7_1353900 5FMG E PLASMODB PF3D7_0727400 5FMG S PLASMODB PF3D7_0727400 5FMG F PLASMODB PF3D7_1474800 5FMG T PLASMODB PF3D7_1474800 5FMG G PLASMODB PF3D7_0317000 5FMG U PLASMODB PF3D7_0317000 5FMG H PLASMODB PF3D7_0931800 5FMG V PLASMODB PF3D7_0931800 5FMG I PLASMODB PF3D7_1328100 5FMG W PLASMODB PF3D7_1328100 5FMG J PLASMODB PF3D7_0108000 5FMG X PLASMODB PF3D7_0108000 5FMG K PLASMODB PF3D7_1470900 5FMG Y PLASMODB PF3D7_1470900 5FMG L PLASMODB PF3D7_1011400 5FMG Z PLASMODB PF3D7_1011400 5FMG M PLASMODB PF3D7_0518300 5FMG A PLASMODB PF3D7_0518300 5FMG N PLASMODB PF3D7_0803800 5FMG B PLASMODB PF3D7_0803800 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: PLASMODIUM FALCIPARUM 20S PROTEASOME CORE BOUND TO A SPECIFIC INHIBITOR タイプ: COMPLEX |
|---|---|
| 緩衝液 | 名称: 50MM TRIS HCL, 5MM MGCL2, 1MM DITHIOTREITOL / pH: 7.5 / 詳細: 50MM TRIS HCL, 5MM MGCL2, 1MM DITHIOTREITOL |
| 試料 | 濃度: 0.1 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | 詳細: OTHER |
急速凍結 | 装置: FEI VITROBOT MARK III / 凍結剤: ETHANE 詳細: VITRIFICATION 1 -- CRYOGEN- ETHANE, HUMIDITY- 95, INSTRUMENT- FEI VITROBOT MARK III, METHOD- BLOT 2.5 SECONDS BEFORE PLUNGING, |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS / 日付: 2014年9月3日 詳細: EACH EXPOSURE WAS RECORDED AS 17 INDIVIDUAL FRAMES CAPTURED AT A RATE OF 0.056 SECOND PER FRAME, EACH WITH AN ELECTRON DOSE OF 2.8 ELECTRONS PER SQUARE ANGSTROM. DATA-SET RECORDED USING EPU SOFTWARE. |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 75000 X / 最大 デフォーカス(公称値): 3200 nm / 最小 デフォーカス(公称値): 1600 nm Bright-field microscopy / 倍率(公称値): 75000 X / 最大 デフォーカス(公称値): 3200 nm / 最小 デフォーカス(公称値): 1600 nm |
| 撮影 | 電子線照射量: 4.8 e/Å2 フィルム・検出器のモデル: FEI FALCON II (4k x 4k) |
| 画像スキャン | デジタル画像の数: 1816 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | 詳細: FULL RECORDED IMAGE | ||||||||||||
| 対称性 | 点対称性 : C2 (2回回転対称 : C2 (2回回転対称 ) ) | ||||||||||||
3次元再構成 | 手法: PROJECTION MATCHING / 解像度: 3.6 Å / 粒子像の数: 97720 / ピクセルサイズ(公称値): 1.04 Å / ピクセルサイズ(実測値): 1.04 Å 詳細: DISORDERED REGIONS WERE NOT MODELED SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-3231. (DEPOSITION ID: 14006). 対称性のタイプ: POINT | ||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL / 詳細: METHOD--FLEXIBLE | ||||||||||||
| 原子モデル構築 | PDB-ID: 1IRU | ||||||||||||
| 精密化 | 最高解像度: 3.6 Å | ||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 3.6 Å
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





