+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11891 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacillus subtilis ribosome-associated quality control complex state B, multibody refinement focussed on RqcH. Ribosomal 50S subunit with P-tRNA, RqcH, and RqcP/YabO | ||||||||||||||||||
 マップデータ マップデータ | Bacillus subtilis 50S ribosme-associated quality control complex state B. in complex P-tRNA, RqcH and RqcP/YabO | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RQC complex /  ribosomal large subunit binding / rescue of stalled ribosome / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / ribosomal large subunit binding / rescue of stalled ribosome / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit /  tRNA binding / structural constituent of ribosome / tRNA binding / structural constituent of ribosome /  翻訳 (生物学) / response to antibiotic / 翻訳 (生物学) / response to antibiotic /  RNA binding RNA binding類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Bacillus subtilis (strain 168) (枯草菌) / Bacillus subtilis (strain 168) (枯草菌) /   Bacillus subtilis subsp. subtilis str. 168 (枯草菌) Bacillus subtilis subsp. subtilis str. 168 (枯草菌) | ||||||||||||||||||
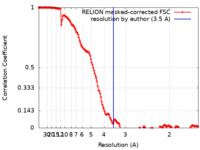
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Crowe-McAuliffe C / Wilson DN | ||||||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  スウェーデン, 5件 スウェーデン, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Structural Basis for Bacterial Ribosome-Associated Quality Control by RqcH and RqcP. 著者: Caillan Crowe-McAuliffe / Hiraku Takada / Victoriia Murina / Christine Polte / Sergo Kasvandik / Tanel Tenson / Zoya Ignatova / Gemma C Atkinson / Daniel N Wilson / Vasili Hauryliuk /    要旨: In all branches of life, stalled translation intermediates are recognized and processed by ribosome-associated quality control (RQC) pathways. RQC begins with the splitting of stalled ribosomes, ...In all branches of life, stalled translation intermediates are recognized and processed by ribosome-associated quality control (RQC) pathways. RQC begins with the splitting of stalled ribosomes, leaving an unfinished polypeptide still attached to the large subunit. Ancient and conserved NEMF family RQC proteins target these incomplete proteins for degradation by the addition of C-terminal "tails." How such tailing can occur without the regular suite of translational components is, however, unclear. Using single-particle cryo-electron microscopy (EM) of native complexes, we show that C-terminal tailing in Bacillus subtilis is mediated by NEMF protein RqcH in concert with RqcP, an Hsp15 family protein. Our structures reveal how these factors mediate tRNA movement across the ribosomal 50S subunit to synthesize polypeptides in the absence of mRNA or the small subunit. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11891.map.gz emd_11891.map.gz | 13.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11891-v30.xml emd-11891-v30.xml emd-11891.xml emd-11891.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11891_fsc.xml emd_11891_fsc.xml | 14.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11891.png emd_11891.png | 39.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11891 http://ftp.pdbj.org/pub/emdb/structures/EMD-11891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11891 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7asaMC  7as8C  7as9C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10540 (タイトル: Affinity-purified RqcH-ribosome-associated quality control complexes from Bacillus subtilis EMPIAR-10540 (タイトル: Affinity-purified RqcH-ribosome-associated quality control complexes from Bacillus subtilisData size: 514.6 Data #1: Unaligned multi-frame micrographs of affinity-purified RqcH ribosome-associated quality control complexes from Bacillus subtilis [micrographs - multiframe])  EMPIAR-10541 (タイトル: Affinity-purified RqcP/YabO-ribosome-associated quality control complexes from Bacillus subtilis EMPIAR-10541 (タイトル: Affinity-purified RqcP/YabO-ribosome-associated quality control complexes from Bacillus subtilisData size: 358.1 Data #1: Unaligned multiframe micrographs of an affinity-purified RqcP/YabO sample [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11891.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11891.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Bacillus subtilis 50S ribosme-associated quality control complex state B. in complex P-tRNA, RqcH and RqcP/YabO | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 50S ribosomal subunit in complex with RqcH, P-tRNA, and YabO/RqcP
| 全体 | 名称: 50S ribosomal subunit in complex with RqcH, P-tRNA, and YabO/RqcP リボソーム リボソーム |
|---|---|
| 要素 |
|
-超分子 #1: 50S ribosomal subunit in complex with RqcH, P-tRNA, and YabO/RqcP
| 超分子 | 名称: 50S ribosomal subunit in complex with RqcH, P-tRNA, and YabO/RqcP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 分子量 | 理論値: 1.6 MDa |
-超分子 #2: RqcH
| 超分子 | 名称: RqcH / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (strain 168) (枯草菌) Bacillus subtilis (strain 168) (枯草菌) |
| 組換発現 | 生物種:   Bacillus subtilis (strain 168) (枯草菌) Bacillus subtilis (strain 168) (枯草菌) |
-超分子 #3: P-tRNA, and YabO/RqcP
| 超分子 | 名称: P-tRNA, and YabO/RqcP / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#5 |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (strain 168) (枯草菌) Bacillus subtilis (strain 168) (枯草菌) |
-分子 #1: Rqc2 homolog RqcH
| 分子 | 名称: Rqc2 homolog RqcH / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (strain 168) (枯草菌) / 株: 168 Bacillus subtilis (strain 168) (枯草菌) / 株: 168 |
| 分子量 | 理論値: 68.341391 KDa |
| 組換発現 | 生物種:   Bacillus subtilis subsp. subtilis str. 168 (枯草菌) Bacillus subtilis subsp. subtilis str. 168 (枯草菌) |
| 配列 | 文字列: MSFDGMFTYG MTHELNEKIM GGRITKIHQP YKHDVIFHIR AKGKNQKLLL SAHPSYSRVH ITAQAYENPS EPPMFCMLLR KHIEGGFIE KIEQAGLDRI MIFHIKSRNE IGDETVRKLY VEIMGRHSNI ILTDAAENVI IDGLKHLSPS MNSYRTVLPG Q DYKLPPAQ ...文字列: MSFDGMFTYG MTHELNEKIM GGRITKIHQP YKHDVIFHIR AKGKNQKLLL SAHPSYSRVH ITAQAYENPS EPPMFCMLLR KHIEGGFIE KIEQAGLDRI MIFHIKSRNE IGDETVRKLY VEIMGRHSNI ILTDAAENVI IDGLKHLSPS MNSYRTVLPG Q DYKLPPAQ DKISPLEASE DDILRHLSFQ EGRLDKQIVD HFSGVSPLFA KEAVHRAGLA NKVTLPKALL ALFAEVKEHR FI PNITTVN GKEYFYLLEL THLKGEARRF DSLSELLDRF YFGKAERDRV KQQAQDLERF VVNERKKNAN KIKKLEKTLE YSE NAKEFQ LYGELLTANL YMLKKGDKQA EVINYYDEES PTITIPLNPN KTPSENAQAY FTKYQKAKNS VAVVEEQIRL AQEE IEYFD QLIQQLSSAS PRDISEIREE LVEGKYLRPK QQKGQKKQKP HNPVLETYES TSGLTILVGK NNRQNEYLTT RVAAR DDIW LHTKDIPGSH VVIRSSEPDE QTIMEAATIA AYFSKAKDSS SVPVDYTKIR HVKKPNGAKP GFVTYDSQHT VFVTPD ADT VIKLKKSGSG GDYKDHDGDY KDHDIDYKDD DDKG |
-分子 #2: Uncharacterized protein YabO
| 分子 | 名称: Uncharacterized protein YabO / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (strain 168) (枯草菌) / 株: 168 Bacillus subtilis (strain 168) (枯草菌) / 株: 168 |
| 分子量 | 理論値: 9.737266 KDa |
| 配列 | 文字列: MRLDKFLKVS RLIKRRTLAK EVADQGRISI NGNQAKASSD VKPGDELTVR FGQKLVTVQV NELKDTTKKE EAANMYTILK EEKLGE |
-分子 #5: 50S ribosomal protein L11
| 分子 | 名称: 50S ribosomal protein L11 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (strain 168) (枯草菌) / 株: 168 Bacillus subtilis (strain 168) (枯草菌) / 株: 168 |
| 分子量 | 理論値: 14.951442 KDa |
| 配列 | 文字列: MAKKVVKVVK LQIPAGKANP APPVGPALGQ AGVNIMGFCK EFNARTADQA GLIIPVEISV YEDRSFTFIT KTPPAAVLLK KAAGIESGS GEPNRNKVAT VKRDKVREIA ETKMPDLNAA DVEAAMRMVE GTARSMGIVI ED |
-分子 #3: tRNA-Ala-1-1
| 分子 | 名称: tRNA-Ala-1-1 / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis subsp. subtilis str. 168 (枯草菌) Bacillus subtilis subsp. subtilis str. 168 (枯草菌)株: 168 |
| 分子量 | 理論値: 24.491547 KDa |
| 配列 | 文字列: GGGGCCUUAG CUCAGCUGGG AGAGCGCCUG CUUUGCACGC AGGAGGUCAG CGGUUCGAUC CCGCUAGGCU CCACCA |
-分子 #4: 23S rRNA
| 分子 | 名称: 23S rRNA / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis subsp. subtilis str. 168 (枯草菌) Bacillus subtilis subsp. subtilis str. 168 (枯草菌)株: 168 |
| 分子量 | 理論値: 949.010938 KDa |
| 配列 | 文字列: GGUUAAGUUA GAAAGGGCGC ACGGUGGAUG CCUUGGCACU AGGAGCCGAU GAAGGACGGG ACGAACACCG AUAUGCUUCG GGGAGCUGU AAGCAAGCUU UGAUCCGGAG AUUUCCGAAU GGGGAAACCC ACCACUCGUA AUGGAGUGGU AUCCAUAUCU G AAUUCAUA ...文字列: GGUUAAGUUA GAAAGGGCGC ACGGUGGAUG CCUUGGCACU AGGAGCCGAU GAAGGACGGG ACGAACACCG AUAUGCUUCG GGGAGCUGU AAGCAAGCUU UGAUCCGGAG AUUUCCGAAU GGGGAAACCC ACCACUCGUA AUGGAGUGGU AUCCAUAUCU G AAUUCAUA GGAUAUGAGA AGGCAGACCC GGGGAACUGA AACAUCUAAG UACCCGGAGG AAGAGAAAGC AAAUGCGAUU CC CUGAGUA GCGGCGAGCG AAACGGGAUC AGCCCAAACC AAGAGGCUUG CCUCUUGGGG UUGUAGGACA CUCUGUACGG AGU UACAAA GGAACGAGGU AGAUGAAGAG GUCUGGAAAG GCCCGCCAUA GGAGGUAACA GCCCUGUAGU CAAAACUUCG UUCU CUCCU GAGUGGAUCC UGAGUACGGC GGAACACGUG AAAUUCCGUC GGAAUCCGGG AGGACCAUCU CCCAAGGCUA AAUAC UCCC UAGUGACCGA UAGUGAACCA GUACCGUGAG GGAAAGGUGA AAAGCACCCC GGAAGGGGAG UGAAAGAGAU CCUGAA ACC GUGUGCCUAC AAGUAGUCAG AGCCCGUUAA CGGGUGAUGG CGUGCCUUUU GUAGAAUGAA CCGGCGAGUU ACGAUCU CG UGCAAGGUUA AGCAGAAGAU GCGGAGCCGC AGCGAAAGCG AGUCUGAAUA GGGCGCAUGA GUACGUGGUC GUAGACCC G AAACCAGGUG AUCUACCCAU GUCCAGGGUG AAGUUCAGGU AACACUGAAU GGAGGCCCGA ACCCACGCAC GUUGAAAAG UGCGGGGAUG AGGUGUGGGU AGGGGUGAAA UGCCAAUCGA ACCUGGAGAU AGCUGGUUCU CUCCGAAAUA GCUUUAGGGC UAGCCUCAA GGUAAGAGUC UUGGAGGUAG AGCACUGAUU GGACUAGGGG CCCCUACCGG GUUACCGAAU UCAGUCAAAC U CCGAAUGC CAAUGACUUA UCCUUGGGAG UCAGACUGCG AGUGAUAAGA UCCGUAGUCG AAAGGGAAAC AGCCCAGACC GC CAGCUAA GGUCCCAAAG UAUACGUUAA GUGGAAAAGG AUGUGGAGUU GCUUAGACAA CCAGGAUGUU GGCUUAGAAG CAG CCACCA UUUAAAGAGU GCGUAAUAGC UCACUGGUCG AGUGACUCUG CGCCGAAAAU GUACCGGGGC UAAACGUAUC ACCG AAGCU GCGGACUGUU CUUCGAACAG UGGUAGGAGA GCGUUCUAAG GGCUGUGAAG CCAGACCGGA AGGACUGGUG GAGCG CUUA GAAGUGAGAA UGCCGGUAUG AGUAGCGAAA GAGGGGUGAG AAUCCCCUCC ACCGAAUGCC UAAGGUUUCC UGAGGA AGG CUCGUCCGCU CAGGGUUAGU CGGGACCUAA GCCGAGGCCG AAAGGCGUAG GCGAUGGACA ACAGGUUGAU AUUCCUG UA CCACCUCCUC ACCAUUUGAG CAAUGGGGGG ACGCAGGAGG AUAGGGUAAG CGCGGUAUUG GAUAUCCGCG UCCAAGCA G UUAGGCUGGG AAAUAGGCAA AUCCGUUUCC CAUAAGGCUG AGCUGUGAUG GCGAGCGAAA UAUAGUAGCG AAGUUCCUG AUUCCACACU GCCAAGAAAA GCCUCUAGCG AGGUGAGAGG UGCCCGUACC GCAAACCGAC ACAGGUAGGC GAGGAGAGAA UCCUAAGGU GAUCGAGAGA ACUCUCGUUA AGGAACUCGG CAAAAUGACC CCGUAACUUC GGGAGAAGGG GUGCUCUGUU A GGGUGCAA GCCCGAGAGA GCCGCAGUGA AUAGGCCCAG GCGACUGUUU AGCAAAAACA CAGGUCUCUG CGAAGCCGUA AG GCGAAGU AUAGGGGCUG ACGCCUGCCC GGUGCUGGAA GGUUAAGAGG AGCGCUUAGC GUAAGCGAAG GUGCGAAUUG AAG CCCCAG UAAACGGCGG CCGUAACUAU AACGGUCCUA AGGUAGCGAA AUUCCUUGUC GGGUAAGUUC CGACCCGCAC GAAA GGCGC AACGAUCUGG GCACUGUCUC AACGAGAGAC UCGGUGAAAU UAUAGUACCU GUGAAGAUGC AGGUUACCCG CGACA GGAC GGAAAGACCC CGUGGAGCUU UACUGCAGCC UGAUAUUGAA UGUUGGUACA GCUUGUACAG GAUAGGUAGG AGCCUU GGA AACCGGAGCG CCAGCUUCGG UGGAGGCAUC GGUGGGAUAC UACCCUGGCU GUAUUGACCU UCUAACCCGC CGCCCUU AU CGGGCGGGGA GACAGUGUCA GGUGGGCAGU UUGACUGGGG CGGUCGCCUC CUAAAAGGUA ACGGAGGCGC CCAAAGGU U CCCUCAGAAU GGUUGGAAAU CAUUCGCAGA GUGUAAAGGC ACAAGGGAGC UUGACUGCGA GACCUACAAG UCGAGCAGG GACGAAAGUC GGGCUUAGUG AUCCGGUGGU UCCGCAUGGA AGGGCCAUCG CUCAACGGAU AAAAGCUACC CCGGGGAUAA CAGGCUUAU CUCCCCCAAG AGUCCACAUC GACGGGGAGG UUUGGCACCU CGAUGUCGGC UCAUCGCAUC CUGGGGCUGU A GUCGGUCC CAAGGGUUGG GCUGUUCGCC CAUUAAAGCG GUACGCGAGC UGGGUUCAGA ACGUCGUGAG ACAGUUCGGU CC CUAUCCG UCGCGGGCGC AGGAAAUUUG AGAGGAGCUG UCCUUAGUAC GAGAGGACCG GGAUGGACGC ACCGCUGGUG UAC CAGUUG UUCUGCCAAG GGCAUCGCUG GGUAGCUAUG UGCGGACGGG AUAAGUGCUG AAAGCAUCUA AGCAUGAAGC CCCC CUCAA GAUGAGAUUU CCCAUUCCGC AAGGAAGUAA GAUCCCUGAA AGAUGAUCAG GUUGAUAGGU CUGAGGUGGA AGUGU GGCG ACACAUGGAG CUGACAGAUA CUAAUCGAUC GAGGACUUAA CC |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 平均電子線量: 29.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー