+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4kt0 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of a virus like photosystem I from the cyanobacterium Synechocystis PCC 6803 | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード |  ELECTRON TRANSPORT (電子伝達系) / ELECTRON TRANSPORT (電子伝達系) /  photosynthetic reaction center / photosynthetic reaction center /  membrane complex (生体膜) / membrane complex (生体膜) /  plastocyanin (プラストシアニン) / cytochrome C6 / plastocyanin (プラストシアニン) / cytochrome C6 /  Ferredoxin (フェレドキシン) Ferredoxin (フェレドキシン) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 plasma membrane-derived photosystem I / plasma membrane-derived photosystem I /  チラコイド / チラコイド /  photosystem I reaction center / photosystem I reaction center /  光化学系I / photosynthetic electron transport in photosystem I / 光化学系I / photosynthetic electron transport in photosystem I /  光化学系I / 光化学系I /  chlorophyll binding / plasma membrane-derived thylakoid membrane / chlorophyll binding / plasma membrane-derived thylakoid membrane /  光合成 / 4 iron, 4 sulfur cluster binding ... 光合成 / 4 iron, 4 sulfur cluster binding ... plasma membrane-derived photosystem I / plasma membrane-derived photosystem I /  チラコイド / チラコイド /  photosystem I reaction center / photosystem I reaction center /  光化学系I / photosynthetic electron transport in photosystem I / 光化学系I / photosynthetic electron transport in photosystem I /  光化学系I / 光化学系I /  chlorophyll binding / plasma membrane-derived thylakoid membrane / chlorophyll binding / plasma membrane-derived thylakoid membrane /  光合成 / 4 iron, 4 sulfur cluster binding / 光合成 / 4 iron, 4 sulfur cluster binding /  electron transfer activity / membrane => GO:0016020 / magnesium ion binding / electron transfer activity / membrane => GO:0016020 / magnesium ion binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Mazor, Y. / Nataf, D. / Toporik, H. / Nelson, N. | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2014 ジャーナル: Elife / 年: 2014タイトル: Crystal structures of virus-like photosystem I complexes from the mesophilic cyanobacterium Synechocystis PCC 6803. 著者: Mazor, Y. / Nataf, D. / Toporik, H. / Nelson, N. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4kt0.cif.gz 4kt0.cif.gz | 1.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4kt0.ent.gz pdb4kt0.ent.gz | 972.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4kt0.json.gz 4kt0.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kt/4kt0 https://data.pdbj.org/pub/pdb/validation_reports/kt/4kt0 ftp://data.pdbj.org/pub/pdb/validation_reports/kt/4kt0 ftp://data.pdbj.org/pub/pdb/validation_reports/kt/4kt0 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | AUTHOR STATED THAT THERE IS NO QUATERNARY STRUCTURE FOR THIS ENTRY. PISA FAILED TO GENERATE ASSEMBLY. THEREFORE THE ASU IS INDICATED AS ASSEMBLY TEMPORARILY. |
- 要素
要素
-Photosystem I P700 chlorophyll a apoprotein ... , 2種, 2分子 AB
| #1: タンパク質 |  光化学系I / PsaA 光化学系I / PsaA分子量: 83036.398 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: 10HIS tag was added to the C terminus of PsaL. The mutated chain is lost during purification. 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)株: Synechocystis sp. PCC 6803  / 遺伝子: BEST7613_2234, MYO_18690, psaA / 遺伝子: BEST7613_2234, MYO_18690, psaA参照: UniProt: L8AHT3, UniProt: P29254*PLUS,  光化学系I 光化学系I |
|---|---|
| #2: タンパク質 |  光化学系I / PsaB 光化学系I / PsaB分子量: 81369.531 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psaB, BEST7613_2235, MYO_18700 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8AIC0, UniProt: P29255*PLUS,  光化学系I 光化学系I |
-Photosystem I subunit ... , 2種, 2分子 DF
| #4: タンパク質 |  光化学系I 光化学系I分子量: 15663.749 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psaD, BEST7613_1459, MYO_11100 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8AFM8, UniProt: P19569*PLUS,  光化学系I 光化学系I |
|---|---|
| #6: タンパク質 |  光化学系I 光化学系I分子量: 18267.082 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psaF, BEST7613_2928 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8AII8, UniProt: P29256*PLUS,  光化学系I 光化学系I |
-Photosystem I reaction center subunit ... , 4種, 4分子 EJKM
| #5: タンパク質 |  光化学系I 光化学系I分子量: 8154.086 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psaE, BEST7613_5968, MYO_118260 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8ASH8, UniProt: P12975*PLUS,  光化学系I 光化学系I |
|---|---|
| #7: タンパク質・ペプチド |  光化学系I 光化学系I分子量: 4535.415 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psaJ, BEST7613_2927, MYO_115450 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8AGL9, UniProt: Q55329*PLUS,  光化学系I 光化学系I |
| #8: タンパク質 |  光化学系I / Photosystem I subunit X 光化学系I / Photosystem I subunit X分子量: 13732.274 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psaK, BEST7613_4536, MYO_129960 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8APJ0, UniProt: P74564*PLUS,  光化学系I 光化学系I |
| #9: タンパク質・ペプチド |  光化学系I / PSI-M 光化学系I / PSI-M分子量: 3382.063 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psbM, psaM, BEST7613_1787, MYO_14340 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8ADF9, UniProt: P72986*PLUS,  光化学系I 光化学系I |
-タンパク質 / 糖 , 2種, 3分子 C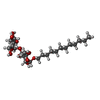

| #16: 糖 | | #3: タンパク質 | |  光化学系I / 9 kDa polypeptide / PSI-C / Photosystem I subunit VII / PsaC 光化学系I / 9 kDa polypeptide / PSI-C / Photosystem I subunit VII / PsaC分子量: 8837.261 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)遺伝子: psaC, BEST7613_5694, MYO_120930 / 発現宿主:   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: L8AST2, UniProt: P32422*PLUS,  光化学系I 光化学系I |
|---|
-非ポリマー , 9種, 149分子 
















| #10: 化合物 |  フィロキノン フィロキノン#11: 化合物 |  鉄・硫黄クラスター 鉄・硫黄クラスター#12: 化合物 | ChemComp-LHG /  Phosphatidylglycerol Phosphatidylglycerol#13: 化合物 |  クロロフィルa クロロフィルa#14: 化合物 | ChemComp-BCR /  Β-カロテン Β-カロテン#15: 化合物 | ChemComp-CLA /  クロロフィルa クロロフィルa#17: 化合物 | ChemComp-LMG / | #18: 化合物 | ChemComp-CL / |  塩化物 塩化物#19: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.94 Å3/Da / 溶媒含有率: 68.75 % |
|---|---|
結晶化 | 温度: 278 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: 4.5% PEG3350, 30mM Tricine-NaOH pH8, 55mM NaCl, 50mM Glycine, 0.005% Nonyl- beta -D-Maltoside., pH 8.5, VAPOR DIFFUSION, SITTING DROP, temperature 278K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID29 / 波長: 0.97625 Å / ビームライン: ID29 / 波長: 0.97625 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2013年2月16日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.97625 Å / 相対比: 1 : 0.97625 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→30 Å / Num. all: 92450 / Num. obs: 91895 / % possible obs: 99.4 % / Observed criterion σ(F): 1 / Observed criterion σ(I): 1 / 冗長度: 4.6 % / Biso Wilson estimate: 61.4 Å2 / Rmerge(I) obs: 0.139 / Net I/σ(I): 7.9 |
| 反射 シェル | 解像度: 2.8→2.95 Å / 冗長度: 4.1 % / Rmerge(I) obs: 2.382 / Mean I/σ(I) obs: 1.1 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB entry 1JB0 with truncated side chains and chlorophyll tails 解像度: 2.8→29.977 Å / SU ML: 0.4 / Isotropic thermal model: single parameter per residue. / σ(F): 1.35 / 位相誤差: 28.46 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 94.61 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→29.977 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: -9.005 Å / Origin y: -7.8261 Å / Origin z: 21.7491 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: chain A or chain B or chain C or chain D or chain E or chain F or chain J or chain K or chain M |
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj


















