+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 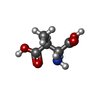 データベース: PDB化学物質要素 / ID: 2AS データベース: PDB化学物質要素 / ID: 2AS | ||||
|---|---|---|---|---|---|
| 名称 | 名称: (| ウィキペディア |  ウィキペディア - L-threo-3-Methylaspartate: l-threo-3-Methylaspartate is an unusual amino acid formed by glutamate mutase and can be metabolised by methylaspartate ammonia-lyase. It is found in the structures of the antibiotics friulimicin and vicenistatin and in carbon metabolism of haloarchaea (Methylaspartate cycle).
... ウィキペディア - L-threo-3-Methylaspartate: l-threo-3-Methylaspartate is an unusual amino acid formed by glutamate mutase and can be metabolised by methylaspartate ammonia-lyase. It is found in the structures of the antibiotics friulimicin and vicenistatin and in carbon metabolism of haloarchaea (Methylaspartate cycle).
...コメント | 抗生剤*YM | |
-Chemical information
| 組成 |  | ||||||
|---|---|---|---|---|---|---|---|
| その他 | タイプ: L-peptide linking / PDB分類: HETAIN / 1文字コード: X / 3文字コード: 2AS / 理論座標の詳細: Corina / モデル座標のPDB-ID: 1W3M | ||||||
| 履歴 |
| ||||||
 外部リンク 外部リンク |  UniChem / UniChem /  Brenda Brenda       / /  ChEBI / ChEBI /  ChEMBL / ChEMBL /  DrugBank / DrugBank /  KEGG_Ligand / KEGG_Ligand /  Metabolights / Metabolights /  NMRShiftDB / NMRShiftDB /  日化辞 / 日化辞 /  PubChem / PubChem /  PubChem_TPharma / PubChem_TPharma /  SureChEMBL / SureChEMBL /  ZINC / ZINC /  ChemSpider / ChemSpider /  Wikipedia search / Wikipedia search /  Google search Google search |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
-詳細
-SMILES
| ACDLabs 10.04 | | CACTVS 3.352 | OpenEye OEToolkits 1.7.0 | |
|---|
-SMILES CANONICAL
| CACTVS 3.352 | | OpenEye OEToolkits 1.7.0 | |
|---|
-InChI
| InChI 1.03 |
|---|
-InChIKey
| InChI 1.03 |
|---|
-SYSTEMATIC NAME
| ACDLabs 10.04 | (| OpenEye OEToolkits 1.6.1 | ( | |
|---|
-PDBエントリ
全4件を表示しています

PDB-1i9c: 
GLUTAMATE MUTASE FROM CLOSTRIDIUM COCHLEARIUM: COMPLEX WITH ADENOSYLCOBALAMIN AND SUBSTRATE

PDB-1kkr: 
CRYSTAL STRUCTURE OF CITROBACTER AMALONATICUS METHYLASPARTATE AMMONIA LYASE CONTAINING (2S,3S)-3-METHYLASPARTIC ACID

PDB-3wv5: 
Complex structure of VinN with 3-methylaspartate

PDB-5xnk: 
Crystal structure of Microcystis aeruginosa PCC 7806 aspartate racemase in complex with DL-methyl-aspartate
 ムービー
ムービー コントローラー
コントローラー