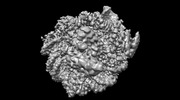
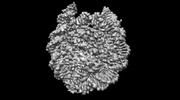
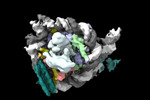
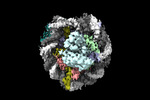
登録情報 データベース : EMDB / ID : EMD-26260タイトル Structure of LIN28b nucleosome bound 2 OCT4 複合体 : Complex of nucleosome bound to two OCT-4sタンパク質・ペプチド : Histone H3.1タンパク質・ペプチド : Histone H4タンパク質・ペプチド : Histone H2A type 2-Cタンパク質・ペプチド : Histone H2B type 2-EDNA : DNA (162-MER)DNA : DNA (162-MER)タンパク質・ペプチド : Maltodextrin-binding protein,POU domain, class 5, transcription factor 1タンパク質・ペプチド : Single-chain variable fragment / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / Mus musculus (ハツカネズミ)手法 / / 解像度 : 2.6 Å Tengfei L / Guan R / Bai Y 資金援助 Organization Grant number 国 National Institutes of Health/National Cancer Institute (NIH/NCI)
ジャーナル : Mol Cell / 年 : 2023タイトル : Structural mechanism of LIN28B nucleosome targeting by OCT4.著者 : Ruifang Guan / Tengfei Lian / Bing-Rui Zhou / David Wheeler / Yawen Bai / 要旨 : Pioneer transcription factors are essential for cell fate changes by targeting closed chromatin. OCT4 is a crucial pioneer factor that can induce cell reprogramming. However, the structural basis of ... Pioneer transcription factors are essential for cell fate changes by targeting closed chromatin. OCT4 is a crucial pioneer factor that can induce cell reprogramming. However, the structural basis of how pioneer factors recognize the in vivo nucleosomal DNA targets is unknown. Here, we determine the high-resolution structures of the nucleosome containing human LIN28B DNA and its complexes with the OCT4 DNA binding region. Three OCT4s bind the pre-positioned nucleosome by recognizing non-canonical DNA sequences. Two use their POUS domains while the other uses the POUS-loop-POUHD region; POUHD serves as a wedge to unwrap ∼25 base pair DNA. Our analysis of previous genomic data and determination of the ESRRB-nucleosome-OCT4 structure confirmed the generality of these structural features. Moreover, biochemical studies suggest that multiple OCT4s cooperatively open the H1-condensed nucleosome array containing the LIN28B nucleosome. Thus, our study suggests a mechanism of how OCT4 can target the nucleosome and open closed chromatin. 履歴 登録 2022年2月18日 - ヘッダ(付随情報) 公開 2023年6月28日 - マップ公開 2023年6月28日 - 更新 2023年6月28日 - 現状 2023年6月28日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード nucleosome (ヌクレオソーム) /
nucleosome (ヌクレオソーム) /  transcription factor (転写因子) /
transcription factor (転写因子) /  transcription (転写 (生物学)) / CHROMATIN BINDING PROTEIN-DNA complex / TRANSCRIPTION-DNA complex
transcription (転写 (生物学)) / CHROMATIN BINDING PROTEIN-DNA complex / TRANSCRIPTION-DNA complex 機能・相同性情報
機能・相同性情報 regulation of asymmetric cell division / endodermal cell fate specification / Specification of primordial germ cells / heart induction / POU5F1 (OCT4), SOX2, NANOG activate genes related to proliferation ...cell fate commitment involved in formation of primary germ layer / cardiac cell fate determination / POU5F1 (OCT4), SOX2, NANOG repress genes related to differentiation / Formation of the anterior neural plate / endodermal-mesodermal cell signaling /
regulation of asymmetric cell division / endodermal cell fate specification / Specification of primordial germ cells / heart induction / POU5F1 (OCT4), SOX2, NANOG activate genes related to proliferation ...cell fate commitment involved in formation of primary germ layer / cardiac cell fate determination / POU5F1 (OCT4), SOX2, NANOG repress genes related to differentiation / Formation of the anterior neural plate / endodermal-mesodermal cell signaling /  regulation of asymmetric cell division / endodermal cell fate specification / Specification of primordial germ cells / heart induction / POU5F1 (OCT4), SOX2, NANOG activate genes related to proliferation / Transcriptional regulation of pluripotent stem cells / regulation of DNA methylation-dependent heterochromatin formation / Germ layer formation at gastrulation / miRNA binding / somatic stem cell population maintenance / blastocyst development / carbohydrate transmembrane transporter activity / anatomical structure morphogenesis / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / BMP signaling pathway / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / epigenetic regulation of gene expression / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Inhibition of DNA recombination at telomere / Meiotic synapsis / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication / SUMOylation of chromatin organization proteins /
regulation of asymmetric cell division / endodermal cell fate specification / Specification of primordial germ cells / heart induction / POU5F1 (OCT4), SOX2, NANOG activate genes related to proliferation / Transcriptional regulation of pluripotent stem cells / regulation of DNA methylation-dependent heterochromatin formation / Germ layer formation at gastrulation / miRNA binding / somatic stem cell population maintenance / blastocyst development / carbohydrate transmembrane transporter activity / anatomical structure morphogenesis / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / BMP signaling pathway / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / epigenetic regulation of gene expression / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Inhibition of DNA recombination at telomere / Meiotic synapsis / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication / SUMOylation of chromatin organization proteins /  DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / negative regulation of miRNA transcription / SIRT1 negatively regulates rRNA expression / HCMV Late Events /
DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / negative regulation of miRNA transcription / SIRT1 negatively regulates rRNA expression / HCMV Late Events /  innate immune response in mucosa / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / G2/M DNA damage checkpoint / HDMs demethylate histones / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / PKMTs methylate histone lysines / RMTs methylate histone arginines /
innate immune response in mucosa / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / G2/M DNA damage checkpoint / HDMs demethylate histones / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / PKMTs methylate histone lysines / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / DNA-binding transcription repressor activity, RNA polymerase II-specific / response to wounding /
遺伝的組換え / Pre-NOTCH Transcription and Translation / DNA-binding transcription repressor activity, RNA polymerase II-specific / response to wounding /  nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / positive regulation of canonical Wnt signaling pathway / UCH proteinases /
nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / positive regulation of canonical Wnt signaling pathway / UCH proteinases /  ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / sequence-specific double-stranded DNA binding / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / RUNX1 regulates transcription of genes involved in differentiation of HSCs / outer membrane-bounded periplasmic space /
ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / sequence-specific double-stranded DNA binding / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / RUNX1 regulates transcription of genes involved in differentiation of HSCs / outer membrane-bounded periplasmic space /  遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response / Senescence-Associated Secretory Phenotype (SASP) /
遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response / Senescence-Associated Secretory Phenotype (SASP) /  遺伝子発現の調節 / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression /
遺伝子発現の調節 / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression /  transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding / sequence-specific DNA binding /
transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding / sequence-specific DNA binding /  chromosome, telomeric region / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / Ub-specific processing proteases / defense response to Gram-positive bacterium /
chromosome, telomeric region / transcription cis-regulatory region binding / DNA-binding transcription factor activity, RNA polymerase II-specific / Ub-specific processing proteases / defense response to Gram-positive bacterium /  cadherin binding / RNA polymerase II cis-regulatory region sequence-specific DNA binding
cadherin binding / RNA polymerase II cis-regulatory region sequence-specific DNA binding
 Homo sapiens (ヒト) /
Homo sapiens (ヒト) / 
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) 単粒子再構成法 /
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.6 Å
クライオ電子顕微鏡法 / 解像度: 2.6 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Mol Cell / 年: 2023
ジャーナル: Mol Cell / 年: 2023
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_26260.map.gz
emd_26260.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-26260-v30.xml
emd-26260-v30.xml emd-26260.xml
emd-26260.xml EMDBヘッダ
EMDBヘッダ emd_26260.png
emd_26260.png http://ftp.pdbj.org/pub/emdb/structures/EMD-26260
http://ftp.pdbj.org/pub/emdb/structures/EMD-26260 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26260
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26260 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_26260.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_26260.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト) クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm
Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー