+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDAR6 |
|---|---|
 試料 試料 | human CSF-1:CSF-1R extracellular signalling complex
|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  ジャーナル: Structure / 年: 2015 ジャーナル: Structure / 年: 2015タイトル: Structure and Assembly Mechanism of the Signaling Complex Mediated by Human CSF-1. 著者: Jan Felix / Steven De Munck / Kenneth Verstraete / Leander Meuris / Nico Callewaert / Jonathan Elegheert / Savvas N Savvides /  要旨: Human colony-stimulating factor 1 receptor (hCSF-1R) is unique among the hematopoietic receptors because it is activated by two distinct cytokines, CSF-1 and interleukin-34 (IL-34). Despite ever- ...Human colony-stimulating factor 1 receptor (hCSF-1R) is unique among the hematopoietic receptors because it is activated by two distinct cytokines, CSF-1 and interleukin-34 (IL-34). Despite ever-growing insights into the central role of hCSF-1R signaling in innate and adaptive immunity, inflammatory diseases, and cancer, the structural basis of the functional dichotomy of hCSF-1R has remained elusive. Here, we report crystal structures of ternary complexes between hCSF-1 and hCSF-1R, including their complete extracellular assembly, and propose a mechanism for the cooperative human CSF-1:CSF-1R complex that relies on the adoption by dimeric hCSF-1 of an active conformational state and homotypic receptor interactions. Furthermore, we trace the cytokine-binding duality of hCSF-1R to a limited set of conserved interactions mediated by functionally equivalent residues on CSF-1 and IL-34 that play into the geometric requirements of hCSF-1R activation, and map the possible mechanistic consequences of somatic mutations in hCSF-1R associated with cancer. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
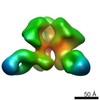
| モデル #216 |  タイプ: mix / ソフトウェア: SASREF / 対称性: P2 / カイ2乗値: 2.23603086735  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|
- 試料
試料
 試料 試料 | 名称: human CSF-1:CSF-1R extracellular signalling complex / 試料濃度: 0.50-10.00 / Entity id: 130 / 131 |
|---|---|
| バッファ | 名称: 50 mM NaH2PO4, 100 m / pH: 7.4 |
| 要素 #130 | 名称: hCSF-1 / タイプ: protein / 記述: Macrophage colony-stimulating factor 1 / 分子量: 17.436 / 分子数: 2 / 由来: Homo sapiens 配列: EEVSEYCSHM IGSGHLQSLQ RLIDSQMETS CQITFEFVDQ EQLKDPVCYL KKAFLLVQDI MEDTMRFRDN TPNAIAIVQL QELSLRLKSC FTKDYEEHDK ACVRTFYETP LQLLEKVKNV FNETKNLLDK DWNIFSKNCN NSFAECSSQ |
| 要素 #131 | 名称: hCSF-1R / タイプ: protein / 記述: Macrophage colony-stimulating factor 1 receptor / 分子量: 53.554 / 分子数: 2 / 由来: Homo sapiens 配列: IPVIEPSVPE LVVKPGATVT LRCVGNGSVE WDGPPSPHWT LYSDGSSSIL STNNATFQNT GTYRCTEPGD PLGGSAAIHL YVKDPARPWN VLAQEVVVFE DQDALLPCLL TDPVLEAGVS LVRVRGRPLM RHTNYSFSPW HGFTIHRAKF IQSQDYQCSA LMGGRKVMSI ...配列: IPVIEPSVPE LVVKPGATVT LRCVGNGSVE WDGPPSPHWT LYSDGSSSIL STNNATFQNT GTYRCTEPGD PLGGSAAIHL YVKDPARPWN VLAQEVVVFE DQDALLPCLL TDPVLEAGVS LVRVRGRPLM RHTNYSFSPW HGFTIHRAKF IQSQDYQCSA LMGGRKVMSI SIRLKVQKVI PGPPALTLVP AELVRIRGEA AQIVCSASSV DVNFDVFLQH NNTKLAIPQQ SDFHNNRYQK VLTLNLDQVD FQHAGNYSCV ASNVQGKHST SMFFRVVESA YLNLSSEQNL IQEVTVGEGL NLKVMVEAYP GLQGFNWTYL GPFSDHQPEP KLANATTKDT YRHTFTLSLP RLKPSEAGRY SFLARNPGGW RALTFELTLR YPPEVSVIWT FINGSGTLLC AASGYPQPNV TWLQCSGHTD RCDEAQVLQV WDDPYPEVLS QEPFHKVTVQ SLLTVETLEH NQTYECRAHN SVGSGSWAFI PISAGTKHHH HHH |
-実験情報
| ビーム | 設備名称:  DORIS III X33 DORIS III X33  / 地域: Hamburg / 国: Germany / 地域: Hamburg / 国: Germany  / 形状: 0.6 / 線源: X-ray synchrotron / 波長: 0.15 Å / スペクトロメータ・検出器間距離: 2.7 mm / 形状: 0.6 / 線源: X-ray synchrotron / 波長: 0.15 Å / スペクトロメータ・検出器間距離: 2.7 mm | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M-W / Pixsize x: 0.172 mm | |||||||||||||||||||||
| スキャン | 測定日: 2009年3月13日 / 保管温度: 10 °C / セル温度: 10 °C / 照射時間: 8 sec. / フレーム数: 15 / 単位: 1/nm /
| |||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||
| 結果 | コメント: SAXS data acquisition for the glycosylated ternary hCSF-1:hCSF-1R full ectodomain complex (MW including glycans: 158 kDa) and subsequent data processing were performed as described in ...コメント: SAXS data acquisition for the glycosylated ternary hCSF-1:hCSF-1R full ectodomain complex (MW including glycans: 158 kDa) and subsequent data processing were performed as described in Elegheert et al., 2011 and Felix et al., 2013 respectively (see references). Rigid-body refinements of the hCSF-1:hCSF-1R complex were performed using the online version of SASREF. For each run, data to 0.25 angstrom-1 was used while imposing P2 symmetry. The crystal structure of full length hCSF-1:hCSF-1R without D1 was taken as a starting rigid-body core (PDB ID: 4WRM). D1 was added as a separate rigid body with a contact restraint of 4 angstrom between its C-terminus and the N-terminus of hCSF-1RD2-D5, as well as 5 N-linked glycans containing a 'dummy' Asparagine residue (Asn-GlcNac2Man5) with 1 angstrom restraints to the C-alpha of truncated Asparagine residues on hCSF-1RD2-D5 (N73, N153, N240, N275 and N353). The SASREF calculated fit of the model to the experimental data gave a chi-squared of 1.65, recalculation of the fit using Crysol and FoXS gave a chi-squared of 3.0 and 1.5 respectively. Here, the fit calculated with FoXS is presented. Felix, J., Elegheert, J., Gutsche, I., Shkumatov, A.V., Wen, Y., Bracke, N., Pannecoucke, E., Vandenberghe, I., Devreese, B., Svergun, D.I., et al. (2013). Human IL-34 and CSF-1 establish structurally similar extracellular assemblies with their common hematopoietic receptor. Structure 21, 528-539. Elegheert, J., Desfosses, A., Shkumatov, A.V., Wu, X., Bracke, N., Verstraete, K., Van Craenenbroeck, K., Brooks, B.R., Svergun, D.I., Vergauwen, B., et al. (2011). Extracellular Complexes of the Hematopoietic Human and Mouse CSF-1 Receptor Are Driven by Common Assembly Principles. Structure 19, 1762-1772.
|
 ムービー
ムービー コントローラー
コントローラー


 SASDAR6
SASDAR6