[English] 日本語
 Yorodumi
Yorodumi- PDB-8yd1: CryoEM structure of M. tuberculosis ClpC1P1P2 complex bound to bo... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 8yd1 | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | CryoEM structure of M. tuberculosis ClpC1P1P2 complex bound to bortezomib, conformation 1 | ||||||||||||||||||||||||||||||
 Components Components |
| ||||||||||||||||||||||||||||||
 Keywords Keywords | HYDROLASE / Mycobacterium tuberculosis / Caseinolytic protease system / Activation | ||||||||||||||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationresponse to 11-deoxycorticosterone / response to dehydroepiandrosterone / negative regulation of lactation / potassium channel inhibitor activity / endopeptidase Clp / endopeptidase Clp complex / ATP-dependent peptidase activity / antioxidant activity / protein quality control for misfolded or incompletely synthesized proteins / cysteine-type endopeptidase inhibitor activity ...response to 11-deoxycorticosterone / response to dehydroepiandrosterone / negative regulation of lactation / potassium channel inhibitor activity / endopeptidase Clp / endopeptidase Clp complex / ATP-dependent peptidase activity / antioxidant activity / protein quality control for misfolded or incompletely synthesized proteins / cysteine-type endopeptidase inhibitor activity / negative regulation of tumor necrosis factor-mediated signaling pathway / negative regulation of canonical NF-kappaB signal transduction / protein folding chaperone / response to progesterone / peptidoglycan-based cell wall / negative regulation of inflammatory response / Golgi lumen / regulation of blood pressure / response to estradiol / response to heat / ATPase binding / serine-type endopeptidase activity / Golgi apparatus / protein homodimerization activity / ATP hydrolysis activity / extracellular space / ATP binding / metal ion binding / plasma membrane / cytosol Similarity search - Function | ||||||||||||||||||||||||||||||
| Biological species |  Mycobacterium tuberculosis H37Rv (bacteria) Mycobacterium tuberculosis H37Rv (bacteria) | ||||||||||||||||||||||||||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 2.81 Å | ||||||||||||||||||||||||||||||
 Authors Authors | Zhou, B. / Zhao, H. / Gao, Y. / Chen, X. / Zhang, T. / He, J. / Xiong, X. | ||||||||||||||||||||||||||||||
| Funding support |  China, 9items China, 9items
| ||||||||||||||||||||||||||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Activation mechanism of caseinolytic chaperone-protease system in Mycobacterium tuberculosis by the anti-cancer drug bortezomib Authors: Zhou, B. / Zhao, H. / Gao, Y. / Chen, X. / Zhang, T. / He, J. / Xiong, X. | ||||||||||||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  8yd1.cif.gz 8yd1.cif.gz | 935.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb8yd1.ent.gz pdb8yd1.ent.gz | 779.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  8yd1.json.gz 8yd1.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  8yd1_validation.pdf.gz 8yd1_validation.pdf.gz | 3 MB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  8yd1_full_validation.pdf.gz 8yd1_full_validation.pdf.gz | 3.1 MB | Display | |
| Data in XML |  8yd1_validation.xml.gz 8yd1_validation.xml.gz | 167.1 KB | Display | |
| Data in CIF |  8yd1_validation.cif.gz 8yd1_validation.cif.gz | 229.3 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/yd/8yd1 https://data.pdbj.org/pub/pdb/validation_reports/yd/8yd1 ftp://data.pdbj.org/pub/pdb/validation_reports/yd/8yd1 ftp://data.pdbj.org/pub/pdb/validation_reports/yd/8yd1 | HTTPS FTP |
-Related structure data
| Related structure data |  39162MC  8ycxC  8yd0C  8yd2C  8yd4C  9jvpC  9jvzC M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
-ATP-dependent Clp protease proteolytic subunit ... , 2 types, 14 molecules GHIJKLMNOPQRST
| #2: Protein | Mass: 19869.652 Da / Num. of mol.: 7 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Mycobacterium tuberculosis H37Rv (bacteria) Mycobacterium tuberculosis H37Rv (bacteria)Strain: H37Rv / Gene: clpP2 / Production host:  #3: Protein | Mass: 19383.111 Da / Num. of mol.: 7 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Mycobacterium tuberculosis H37Rv (bacteria) Mycobacterium tuberculosis H37Rv (bacteria)Strain: H37Rv / Gene: clpP1, clpP / Production host:  |
|---|
-Protein / Protein/peptide , 2 types, 7 molecules ABCDEFa
| #1: Protein | Mass: 73270.336 Da / Num. of mol.: 6 / Mutation: E288A, F444S, E626A Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Mycobacterium tuberculosis H37Rv (bacteria) Mycobacterium tuberculosis H37Rv (bacteria)Strain: H37Rv / Gene: clpC1 / Production host:  #4: Protein/peptide | | Mass: 2624.252 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  |
|---|
-Non-polymers , 4 types, 31 molecules 


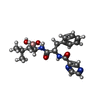



| #5: Chemical | ChemComp-MG / #6: Chemical | ChemComp-ATP / #7: Chemical | #8: Chemical | ChemComp-BO2 / |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | N |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component | Name: CryoEM structure of M. tuberculosis ClpC1P1P2 complex bound to bortezomib, conformation 1 Type: COMPLEX / Entity ID: #1-#4 / Source: RECOMBINANT |
|---|---|
| Source (natural) | Organism:  |
| Source (recombinant) | Organism:  |
| Buffer solution | pH: 7.5 |
| Specimen | Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES |
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: TFS KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal defocus max: 2400 nm / Nominal defocus min: 800 nm |
| Image recording | Electron dose: 50 e/Å2 / Film or detector model: FEI FALCON IV (4k x 4k) |
- Processing
Processing
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION |
|---|---|
| 3D reconstruction | Resolution: 2.81 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 95184 / Symmetry type: POINT |
 Movie
Movie Controller
Controller








 PDBj
PDBj






